2. 南京林业大学, 江苏 南京 210037
2. Nanjing Forestry University, Nanjing 210037, China
猴耳环(Archidendron clypearia)为含羞草科(Mimosaceae)猴耳环属植物,常绿乔木,是我国南方重要的中药材树种[1-2],其根、叶、果实、嫩枝均可入药,功用相同。具有抗菌、消炎、抗病毒、抗肿瘤等作用,被现代医药界称为“绿色抗生素”[3-5]。近年来,人类活动的干扰对猴耳环天然资源及其生境造成了严重破坏,大面积的砍伐和改造天然林使得猴耳环逐渐退化成劣势树种,原有的一些分布区内已不见其踪影,再加上气候的改变和环境的恶化,野生种群资源不断减少,严重威胁野生猴耳环的生存和生长[6-7]。因此,开展野生猴耳环遗传多样性研究对该植物的保护具有重要的理论和现实意义。
SSR (simple sequence repeats)分子标记是近年来发展起来的一种以特异引物PCR为基础的分子标记技术,具有重复性好、多态性高、稳定性强等优点,是育种研究中重要的遗传标记方法之一[8], 已广泛应用于药用植物品种图谱构建、种质资源评价与鉴定、种群遗传多样性分析、分子辅助育种等研究。胡文舜等[9]对新育成的19个枇杷(Eriobotrya japonica)杂交品种(系)进行了SSR标记鉴定分析, 建立了24份枇杷品种的分子指纹图谱; 李洪果等[10]对868份杜仲(Eucommia ulmoides)种质资源进行了SSR遗传多样性分析,表明其群体具有较高的遗传多样性水平;朱巧等[11]对黄精属(Polygonatum)的6种植物进行了SSR遗传差异分析, 推测西部地区可能是我国黄精属植物的起源中心。SSR分子标记技术为中药资源的种质鉴定提供了新思路,是快速有效的药用植物鉴定方法[12]。目前,采用分子标记技术对猴耳环属植物进行研究较少,赵经华[13]利用DNA条形码技术,对不同产地的猴耳环、亮叶猴耳环(A. lucidum)和薄叶猴耳环(A. utile)进行了分类。
本研究首次利用开发的SSR引物对野生猴耳环遗传多样性进行研究,以期全面揭示广东省天然猴耳环群体的遗传多样性及其群体间的遗传分化, 了解各群体之间的亲缘关系,为猴耳环种质资源的保护及合理利用提供理论依据。
1 材料和方法 1.1 材料于2019年10月开始对广东省野生猴耳环进行资源调查和样品采集,共收集到12个野生猴耳环群体(表 1)。每个群体采集嫩叶,采用变色硅胶迅速脱水干燥,装入自封袋中,带回实验室,–20℃低温冰箱保存,用于DNA提取。群体内个体间隔不少于50 m,对于数量较少的群体,采集全部个体。
| 表 1 野生猴耳环群体信息 Table 1 Information of Archidendron clypearia populations in Guangdong |
采用改良的CTAB法[14]提取猴耳环叶片的DNA, 用1.2%琼脂糖凝胶电泳对DNA进行检测, 选取条带清晰明亮的DNA样品置于–20℃冰箱保存备用。
利用课题组前期开发的21个标记(待发表,引物信息见表 2)对所有个体进行PCR扩增和检测。SSR引物由北京擎科生物科技有限公司广州分公司合成。
| 表 2 21对SSR引物信息和遗传多样性参数 Table 2 Information and genetic diversity parameters of 21 SSR primers |
PCR反应体系为10.0 µL,包含模板DNA 0.5 µL, 正反向引物各0.5 µL,2×Taq PCR Mix聚合酶5 µL,ddH2O 3.5 µL。PCR扩增程序为:94℃预变性2 min;然后94℃变性30 s,56℃退火30 s,72℃延伸30 s, 共30个循环;最后72℃延伸10 min,4℃保存。PCR扩增产物由北京擎科生物科技有限公司通过3730XL自动测序仪(ABI Co., Foster, CA, USA)进行检测。
1.3 数据统计分析采用GeneMarker V.2.2.0软件[15]判读电泳结果并统计,用CERVUS 3.0.7软件[16]计算各SSR位点的观测等位变异数(Na)、观测杂合度(Ho)、期望杂合度(He)、多态信息含量(PIC),用GenALEx 6.5.1[17]计算位点有效等位变异数(Ne)、Shannon多样性指数(I)和群体的Na、Ne、Ho、He、I、固定指数(F)等,同时进行分子方差分析(analysis of molecular variance, AMOVA)、计算各群体遗传距离与地理距离,并进行mantel相关性检验。采用NTSYS-PC 2.0软件[18]基于Nei氏遗传距离进行UPGMA聚类分析,构建树状聚类,基于遗传距离矩阵进行主坐标分析(principal coordinates analysis, PCoA),构建二维平面、三维空间分布图。
2 结果和分析 2.1 SSR引物的多态性和群体遗传多样性分析采用SSR标记对146份猴耳环种质资源的遗传多样性进行分析,21对SSR引物共检测到249个等位基因(表 2),引物的等位基因数(Na)为5~23个,平均每对引物扩增出11.86个等位基因,有效等位基因数(Ne)为1.857~5.147,Shannon多样性指数(I)为0.714~2.106,观测杂合度(Ho)为0.287~0.857, 期望杂合度(He)为0.478~0.920,多态信息含量(PIC)为0.401~0.911。
从表 3可见,猴耳环群体的Na、Ne、I、Ho、He、F分别为3.762~7.571、3.047~4.167、1.160~ 1.528、0.598~0.769、0.625~0.716和-0.210~0.110。
| 表 3 12个猴耳环群体的遗传多样性参数 Table 3 Genetic diversity parameters of 12 Archidendron clypearia population |
He和I能较好地度量群体的变异程度与遗传多样性。12个猴耳环群体的He为BL > DH > LM > QX > ZC > LH > XF > TS > DYS≥HD > FK > CA;I值为BL > DH > ZC > LM > QX > LH > HD > XF > FK > DYS > CA > TS。总体来看,博罗群体的遗传多样性最高,潮安群体相对较低。不同猴耳环群体间的遗传多样性存在较大差异,鼎湖群体的猴耳环资源份数最多,但遗传多样性水平并不是最高,遗传多样性水平与样本数量无关[19]。有效等位基因数(Ne)能够反映群体的遗传变异,当Ne值越接近等位基因数(Na)值时, 等位基因在群体中的分布就越均匀[20]。台山群体的有效等位基因数与等位基因数接近,说明等位基因在群体中分布较均匀,其余群体的差异较大,等位基因存在极端值。固定系数F是随机交配下群体内观测杂合度和期望杂合度的偏离值,当F > 0时,表示该群体在该位点杂合子不足;F < 0时,表示杂合子过剩[10]。潮安、清新、台山、龙门、封开5个群体的F < 0,表明杂合子过剩,大瑶山、新丰、博罗、惠东、鼎湖、增城群体的F > 0,表明杂合子不足。
2.2 猴耳环群体的遗传分化基于21对SSR引物对猴耳环群体的分化系数和基因流(Nm)进行分析。结果表明,基因流在不同引物位点间差异较大,Nm为1.654~8.019,平均为3.536,表明猴耳环群体间存在适度的基因交流,基因交流能够促进居群发生基因分化。猴耳环群体间的平均遗传分化系数(Fst)为0.071,平均近交系数(Fit)为0.069,表明群体间既存在一定程度的变异, 群体内还存在一定程度的近交。AMOVA分析表明(表 4), 猴耳环群体内的遗传变异达97%,群体间的遗传变异仅3%,说明猴耳环群体内遗传分化大于群体间的分化。
| 表 4 基于SSR的猴耳环群体分子变异分析(AMOVA) Table 4 AMOVA analysis on SSR loci of Archidendron clypearia populations |
群体间的遗传一致度和遗传距离可以判断群体间的亲缘关系,遗传一致度数值越大,表明亲缘关系越近,而遗传距离越小,其亲缘关系越近。从表 5可知,遗传一致度最大是TS群体和CA群体,为0.380,其遗传距离也最小,为0.684,表明TS群体与CA群体的亲缘关系较近。遗传一致度最小的是DH群体与BL群体,仅0.065,其遗传距离也最大,为0.937,表明DH群体与BL群体的亲缘关系较远。
| 表 5 猴耳环群体间的遗传一致度和遗传距离 Table 5 Genetic identity and genetic distance among populations of Archidendron clypearia |
对12个猴耳环群体间的遗传距离与地理距离矩阵进行Mantel检验,结果表明遗传距离与地理距离的相关关系不显著(r=0.013, P=0.330)。
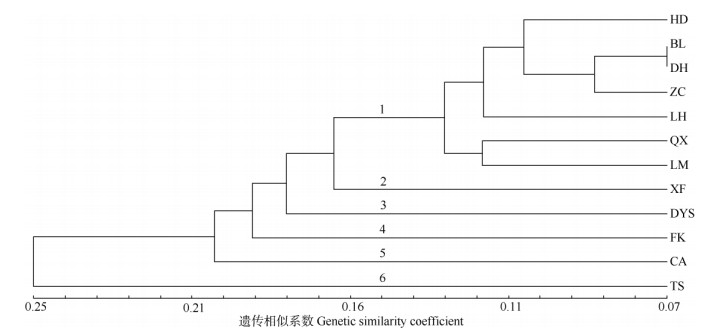
使用NTSYS-PC 2.1基于Nei氏遗传距离对猴耳环12个群体进行UPGMA聚类分析(图 1),结果表明,在遗传相似系数为0.16时,可将猴耳环12个群体分为6个类群。第1类群包括花都、博罗、鼎湖、增城、惠东、清新、龙门7个群体,在遗传系数为0.13时可将第1类群分为2个亚类,花都、博罗、鼎湖、增城、惠东聚为第1亚类,且博罗与鼎湖群体的遗传关系较近,在系数为0.07时聚在一起,清新、龙门聚为第2亚类。新丰、大瑶山、封开、潮安、台山分别聚为第2、3、4、5、6类群。台山群体在遗传系数为0.25时就最先与其他群体区分,单独聚为一类,说明台山群体与其他群体的亲缘关系较远。
|
图 1 猴耳环12个群体的UPGMA树 Fig. 1 UPGMA tree of 12 Archidendron clypearia populations |
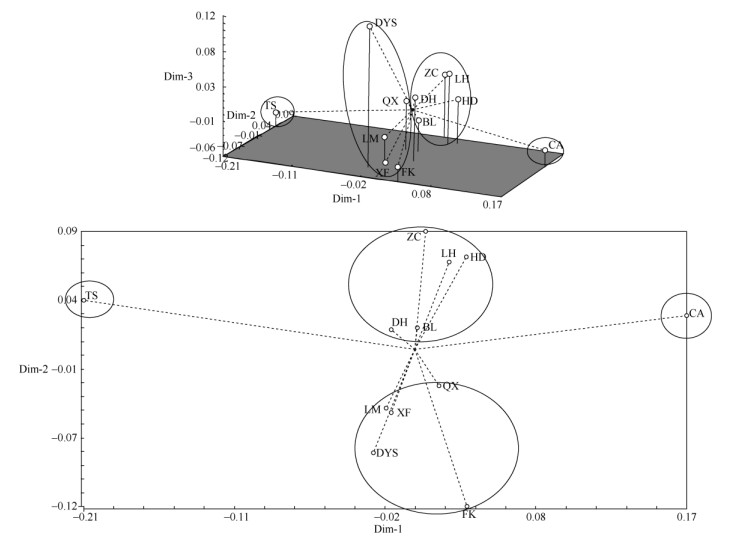
使用NTSYS-PC 2.1对猴耳环群体遗传距离矩阵进行了中心化处理,形成群体分布的三维、二维图(图 2)。主坐标分析结果表明,12个群体被明显的分为4个类群,台山群体为第1类,潮安群体为第2类,惠东、花都、增城、鼎湖、博罗聚为第3类,龙门、大瑶山、封开、清新、新丰聚为第4类。主坐标分析的分组趋势与UPGMA聚类大致相同, 鼎湖、博罗群体位于图形相对中心的区域,表明其与各群体之间的遗传关系都不太远。
|
图 2 猴耳环12个群体的主坐标二维、三维分析 Fig. 2 Two- and three-dimensions of PCoA for 12 Archidendron clypearia populations |
本研究对广东省12个猴耳环群体146株个体进行遗传多样性分析,结果表明猴耳环群体的遗传多样性水平较高,12个群体的平均期望杂合度为0.662,与其他多年生植物的平均期望杂合度(0.68)相当,且高于Nybom[21]基于微卫星分析的期望杂合度平均值(0.61±0.22),表明野生猴耳环群体有着丰富的遗传多样性,含有较高的遗传多样性水平。
有研究表明,自交和混合育种的分类群多样性显著低于异交分类群,异交、广布的植物通常具有较高的遗传多样性[22]。猴耳环作为乔木速生树种, 虽可进行实生苗繁殖且种子的萌发力强,但萌发的幼苗会在1~2 a内相继死亡,林内极少发现幼树[23], 使得其遗传关系并未发生改变,仍然保存了其较高的遗传多样性。博罗群体与鼎湖群体的遗传多样性高, 台山群体与潮安群体的遗传多样性低,这可能与台山群体与潮安群体林内有采伐现象有关,而鼎湖群体与博罗群体因位于国家自然保护区内,植被生境完好, 无采伐现象,保存了较高的遗传多样性,可见人为干扰也可对种质资源的遗传多样性产生影响[24]。
本研究中野生猴耳环群体的Fst为0.070,表明群体间的遗传分化趋于稳定,物种的群体间存在中等程度的遗传分化[25]。这与AMOVA分析结果一致,猴耳环97%的遗传变异发生在群体内,群体间几乎没有遗传分化。猴耳环主要依靠水媒、虫媒传播,流动的水源可以将其种子带到水源流经的任何地方,猴耳环的种子是黑叶猴和白头叶猴的主要觅食食物之一, 当动物觅食行为发生,就会产生传播,促进了群体之间的基因交流[26-28],使群体间的遗传分化水平不显著。这种随机因素可能也是野生猴耳环群体的遗传距离和地理距离没有显著相关性的主要原因。
本研究基于SSR分子标记分析表明,猴耳环群体的遗传多样性较高;群体间的基因流动性较大, 遗传交流水平高;群体间的遗传分化程度低,遗传变异小,短期内不会发生新的遗传分化;遗传变异主要发生在群体内,群体间仅占3%,在对其遗传多样性进行保护时,可以保存其较大种群。但随着生境破坏和人为大量的砍伐,保护野生猴耳环种质资源刻不容缓,对猴耳环资源进行人工栽植和扩大繁殖,将有利于保持野生猴耳环的遗传多样性和防止种群衰退,为野生猴耳环种质资源的保存与利用奠定坚实的基础。
| [1] |
WU D L. Flora of Guangdong, Vol[M]. Guangzhou: Guangdong Science and Technology Press, 2003: 60-80. 吴德邻. 广东植物志, 第5卷[M]. 广东: 广东科技出版社, 2003: 60-80. |
| [2] |
ZHENG W J. China Annals of Trees, Vol[M]. Beijing: China Forestry Publishing House, 1985: 2235-2337. 郑万钧. 中国树木志, 第2卷[M]. 北京: 中国林业出版社, 1985: 2235-2337. |
| [3] |
LI Y L, LI K M, SU M X, et al. Studies on antiviral constituents in stems and leaves of Pithecellibium clypearia[J]. China J Chin Mat Med, 2006, 31(5): 397-400. 李药兰, 李克明, 苏妙贤, 等. 猴耳环抗病毒有效成分研究[J]. 中国中药杂志, 2006, 31(5): 397-400. DOI:10.3321/j.issn:1001-5302.2006.05.013 |
| [4] |
LI M H, YIN M Q, FENG J H, et al. Advances in antitumor effects of gallic acid[J]. Inform Trad Chin Med, 2011, 28(1): 109-111. 李沐涵, 殷美琦, 冯靖涵, 等. 没食子酸抗肿瘤作用研究进展[J]. 中医药信息, 2011, 28(1): 109-111. DOI:10.3969/j.issn.1002-2406.2011.01.044 |
| [5] |
KANG J, LIU C, WANG H Q, et al. Studies on the bioactive flavor-noids isolated from Pithecellobium clypearia Benth[J]. Molecules, 2014, 19(4): 4479-4490. DOI:10.3390/molecules19044479 |
| [6] |
CAI X A, PENG S L, CAO H L, et al. Community analysis of a secondary evergreen broadleaved forest at Luogang, Guangzhou[J]. Chin J Appl Environ Biol, 1998, 4(2): 107-114. 蔡锡安, 彭少麟, 曹洪麟, 等. 广州罗岗村边次生常绿阔叶林群落分析[J]. 应用与环境生物学报, 1998, 4(2): 107-114. |
| [7] |
LI M, HUANG S N, CHEN Z X, et al. The research status and utili-zation prospect of medicinal tree species of Archidendron clypearia[J]. Sci Silv Sin, 2018, 54(4): 142-154. 李梅, 黄世能, 陈祖旭, 等. 药用乔木树种猴耳环研究现状及开发利用前景[J]. 林业科学, 2018, 54(4): 142-154. DOI:10.11707/j.1001-7488.20180417 |
| [8] |
ZIETKIEWICZ E, RAFALSKI A, LABUDA D. Genome fingerprinting by simple sequence repeat (SSR)-anchored polymerase chain reaction amplification[J]. Genomics, 1994, 20(2): 176-183. DOI:10.1006/geno.1994.1151 |
| [9] |
HU W S, DENG C J, XU Q Z, et al. Identification and fingerprint construction of 19 new hybrid varieties (lines) of loquat by SSR[J]. J Trop Subtrop Bot, 2020, 28(2): 153-162. 胡文舜, 邓朝军, 许奇志, 等. 19个枇杷杂交新品种(系)的SSR鉴定和指纹图谱构建[J]. 热带亚热带植物学报, 2020, 28(2): 153-162. DOI:10.11926/jtsb.4131 |
| [10] |
WANG L, LI C R, LI F G, et al. Analysis of SSR marker diversity and population structure in Eucalyptus cloeziana[J]. Mol Plant Breed, 2019, 17(13): 4470-4478. 王莉, 李昌荣, 李发根, 等. 大花序按SSR位点多样性和群体结构分析[J]. 分子植物育种, 2019, 17(13): 4470-4478. DOI:10.13271/j.mpb.017.004470 |
| [11] |
ZHU Q, DENG X, ZHANG S B, et al. Genetic diversity of 6 species in Polygonatum by SSR marker[J]. China J Chin Mat Med, 2018, 43(14): 2935-2943. 朱巧, 邓欣, 张树冰, 等. 黄精属6种植物的SSR遗传差异分析[J]. 中国中药杂志, 2018, 43(14): 2935-2943. DOI:10.19540/j.cnki.cjcmm.20180510.014 |
| [12] |
WANG G, CAO P, WEI X M, et al. Applications of molecular markers in study of germplasm resources of medicinal plants[J]. Mod Chin Med, 2019, 21(11): 1435-1444. 王刚, 曹佩, 韦学敏, 等. 分子标记技术在药用植物种质资源研究中的应用[J]. 中国现代中药, 2019, 21(11): 1435-1444. DOI:10.13313/j.issn.1673-4890.20190830006 |
| [13] |
ZHAO J H. Molecular identification and chemical component analysis of Archidendron dypearia from different regions in China[D]. Guang-zhou: Guangzhou University of Chinese Medicine, 2013: 16-30. 赵径华. 不同产地猴耳环分子鉴别及质量研究[D]. 广州: 广州中医药大学, 2013: 16-30. |
| [14] |
LI R H, XIA Y S, LIU S Z, et al. CTAB-impoved method of DNA extraction in Plant[J]. Res Explor Lab, 2009, 28(9): 14-16. 李荣华, 夏岩石, 刘顺枝, 等. 改进的CTAB提取植物DNA方法[J]. 实验室研究与探索, 2009, 28(9): 14-16. DOI:10.3969/j.issn.1006-7167.2009.09.005 |
| [15] |
HOLLAND M M, PARSON W. GeneMarker® HID: A reliable software tool for the analysis of forensic STR data[J]. J Forensic Sci, 2011, 56(1): 29-35. DOI:10.1111/j.1556-4029.2010.01565.x |
| [16] |
KALINOWSKI S T, TAPER M L, MARSHALL T C. Revising how the computer program cervus accommodates genotyping error increases success in paternity assignment[J]. Mol Ecol, 2007, 16(5): 1099-1106. DOI:10.1111/j.1365-294X.2007.03089.x |
| [17] |
PEAKALL R, SMOUSE P E. GENALEX 6:Genetic analysis in Excel: Population genetic software for teaching and research[J]. Mol Ecol Notes, 2006, 6(1): 288-295. |
| [18] |
ROHLF F J. NTSYS-pc: Numerical Taxonomy and Multivariate Analysis System Version 2[M]. New York: Exeter Publications, 2000: 236-239.
|
| [19] |
XU N, CHENG X Z, WANG L X, et al. Screening and application of SSR molecular markers for genetic diversity analysis of Chinese Adzuki bean germplasm resources[J]. Acta Agron Sin, 2009, 35(2): 219-227. 徐宁, 程须珍, 王丽侠, 等. 用于中国小豆种质资源遗传多样性分析SSR分子标记筛选及应用[J]. 作物学报, 2009, 35(2): 219-227. DOI:10.3724/SP.J.1006.2009.00219 |
| [20] |
ZHANG X F, ZHANG S H, LI Y, et al. Genetic diversity analysis of chestnut germplasm in Yanshan region based on SSR markers[J]. J Chin Agric Univ, 2020, 25(4): 61-71. 张馨方, 张树航, 李颖, 等. 基于SSR标记的燕山板栗种质资源遗传多样性分析[J]. 中国农业大学学报, 2020, 25(4): 61-71. DOI:10.11841/j.issn.1007-4333.2020.04.07 |
| [21] |
NYBOM H. Comparison of different nuclear DNA markers for esti-mating intraspecific genetic diversity in plants[J]. Mol Ecol, 2004, 13(5): 1143-1155. DOI:10.1111/j.1365-294X.2004.02141.x |
| [22] |
HAMRICK J L. Isozymes and the analysis of genetic structure in plant populations[M]//SOLTIS D E, SOLTIS P S. Isozymes in Plant Biology. London: Chapman and Hall, 1990: 87-105
|
| [23] |
LI H, HUANG S N, WANG W W, et al. Effects of different solar-shading treatments on growth performance of Archidendron clypearia seedlings[J]. J CS Univ For Technol, 2012, 32(10): 147-150, 197. 李浩, 黄世能, 王卫文, 等. 不同遮阳处理对猴耳环苗期生长的影响[J]. 中南林业科技大学学报, 2012, 32(10): 147-150, 197. DOI:10.14067/j.cnki.1673-923x.2012.10.018 |
| [24] |
HU W, LUO Y, YANG Y, et al. Genetic diversity and population genetic structure of wild sweet osmanthus revealed by microsatellite markers[J]. Acta Hort Sin, 2014, 41(7): 1427-1435. 胡菀, 罗意, 阳亿, 等. 野生桂花的遗传多样性和遗传结构研究[J]. 园艺学报, 2014, 41(7): 1427-1435. DOI:10.16420/j.issn.0513-353x.2014.07.018 |
| [25] |
WRIGHT S. Evolution and the Genetics of Populations, Volume 4:Variability within and among Natural Populations[M]. Chicago: University of Chicago Press, 1978: 1191-1192.
|
| [26] |
HUANG Z H. Feeding ecology of Françoisi langurs (Trachypithecus françoisi) in Nonggang Nature Reserve, China[D]. Guilin: Guangxi Normal University, 2007: 13-32. 黄中豪. 弄岗黑叶猴(Trachypithecus françoisi)的觅食生态学[D]. 桂林: 广西师范大学, 2007: 13-32. |
| [27] |
ZHOU Q H, TANG Z, LI Y B, et al. Food diversity and choice of white-headed langur in fragmented limestone hill habitat in Guangxi, China[J]. Acta Ecol Sin, 2013, 33(2): 109-113. DOI:10.1016/j.chnaes.2013.01.007 |
| [28] |
PETIT R J, AGUINAGALDE I, DE BEAULIEU J L, et al. Glacial refugia: Hotspots but not melting pots of genetic diversity[J]. Science, 2003, 300(5625): 1563-1565. DOI:10.1126/science.1083264 |
 2021, Vol. 29
2021, Vol. 29


