金线莲(Anoectochilus roxburghii)是兰科(Orchidaceae)开唇兰属植物,其主要活性成分有金线莲苷[1]、黄酮[2]、多糖[3]等,具有降血糖[4]、保肝[5]和抑制肿瘤[6]等功效,是福建道地中药材。金线莲对生境条件要求苛刻且胚发育不全,须与菌根真菌共生,才能萌发,种子自然条件下萌发率低,野生资源濒临灭绝。虽然,近年来经过科研工作者的努力,金线莲在组织培养和人工栽培的关键技术上取得重大突破[7–8],但是,长期的无性繁殖,也造成了金线莲品质的退化,因此,开展金线莲有性杂交育种研究已经迫在眉睫。花器官是植物有性繁殖器官,有关金线莲花器官发育已有少量的研究报道。邢丙聪等[9]筛选了23条金线莲的WRKY转录因子,其中ArWRKY5和ArWRKY20基因在叶芽期、花芽期和开花期的花中表达量都显著高于根、茎、叶, 可能具有调控花发育的功能。陈育青等[10–12]利用电子显微镜观察了金线莲花药发育过程的形态、钙离子、多糖和脂滴的分布特征。
YABBY家族转录因子是一类植物特有转录因子,在植物生长、发育和形态发生中发挥重要作用,包含有N端C2C2锌指结构域和C端类似HMG框的螺旋-环-螺旋的YABBY结构域[13]。目前已经从拟南芥(Arabidopsis thaliana)[14]、水稻(Oryza sativa)[15]、荷花(Nelumbo nucifera)[16]、人参(Panax ginseng)[17]、小桐子(Jatropha curcas)[18]、巴西橡胶树(Hevea brasiliensis)[19]、毛果杨(Populus trichocarpa)[20]等作物中分别鉴定出6、8、9、16、7、11、12个YABBY基因。双子叶植物YABBY基因家族被分为CRABS CLAW (CRC)、FILAMENTOUS FLOWER (FIL)/ YABBY3 (YAB3)、INNER NO OUTER (INO)、YABBY2 (YAB2)和YABBY5 (YAB5)等5个亚家族,单子叶植物被分为4个亚家族,缺少YAB5亚家族[21]。
CRC亚家族基因与心皮发育、蜜腺形成、花分生组织终止、果实发育和叶片中脉形成有关,同源基因在不同作物间存在功能差异。豌豆(Pisum sativum)的PsCRC基因调控心皮的融合、蜜腺的发育[22]。玉米(Zea mays)的drl1和drl2是花分生组织决定和花器官发育所必需的[23]。黄瓜(Cucumis sativus)的CsCRCG通过转录激活CsARP1,从而增强细胞扩增,正向调节果实伸长[24]。目前,未见金线莲CRC基因的相关研究报道。
鉴于CRC亚家族基因在植物花器官、果实和叶片发育过程发挥重要作用,本研究在前期福建种源金线莲全长转录组测序的基础上,筛选克隆了1条CRC基因的CDS序列,并开展生物信息学分析、亚细胞定位以及不同器官表达分析,以期为进一步验证基因功能奠定基础。
1 材料和方法 1.1 材料试验材料为福建种源金线莲(Anoectochilus roxburghii)种植苗,种植于福建省农业科学院亚热带农业研究所温室大棚;亚细胞定位材料为本氏烟草(Nicotiana benthamiana)。
1.2 ArCRC基因的克隆取开花期金线莲健康植株,分别取根、茎、叶、花、叶中脉和叶外缘组织,液氮速冻后-70 ℃保存。利用湖南艾科瑞生物工程有限公司的SteadyPure植物RNA提取试剂盒(货号:AG21019)和Evo M-MLV反转录试剂盒(货号:AG11706)提取总RNA和合成单链cDNA。
根据金线莲全长转录组数据获得的基因序列, 设计扩增全长CDS引物ArABBY-F和ArABBY-R (表 1)。以根、茎、叶和花混合的cDNA为模板, 进行PCR扩增。反应条件为:94 ℃预变性30 s;98 ℃变性10 s,58 ℃退火30 s,72 ℃延伸40 s,35个循环;72 ℃延伸2 min。胶回收目的条带,连接载体pMD18T,转化大肠杆菌DH5α感受态细胞,阳性克隆进行测序。测序结果用DNAMAN V6比对, 提取比对正确克隆的质粒,命名为18T-ArCRC。
| 表 1 引物 Table 1 Primer |
利用Protein BLAST (https://blast.ncbi.nlm.nih.gov/Blast.cgi)和ProtParam (https://web.expasy.org/protparam/)在线搜索和预测蛋白的保守结构域和理化性质。用MEGA 6.0软件[25]构建系统进化树(Neighbor-Joining Tree, Bootstrap 1000)。
1.4 ArCRC亚细胞定位分析ArCRC亚细胞定位分析委托北京启迪利泰科技有限公司完成。构建pSuper1300-35S-ArCRC-GFP重组质粒并转化农杆菌GV3101,与含有pCAMBIA 1300-35S-Histone-mcherry质粒(细胞核定位蛋白)的农杆菌GV3101以1:1体积比混合,选取生长状态良好的烟草,放于白色荧光灯下1 h, 选择倒3和倒4叶片下表面注射,暗培养48 h后激光共聚焦显微镜(Lecia sp8, 德国)下观察叶片,并捕捉荧光信号获取图像。
1.5 原核表达与融合蛋白的Western blot检测用BamH Ⅰ和Xho Ⅰ双酶切质粒18T-ArCRC和pGEX-4T-1,经胶回收、连接后转化大肠杆菌DH5α感受态细胞,提取阳性克隆质粒,获得重组原核表达质粒pGEX-4T-ArCRC。将pGEX-4T-ArCRC转化大肠杆菌(E. coli) BL21(DE3),诱导表达参照林江波等[26]的方法,蛋白分子量标准使用翌圣生物科技(上海)股份有限公司的三色预染蛋白质分子量标准(10~ 180 kD) (货号:20351ES72)。Western blot检测参考教杨等[27]的方法,一抗和二抗分别为鼠抗GST单克隆抗体和HRP标记的羊抗鼠IgG抗体。
1.6 ArCRC基因表达分析用Primer Premier 5.0软件设计ArCRC基因引物bby-F和bby-R,产物长度110 bp,以Actin作为内参基因[28],引物为ArACT1和ArACT2。以金线莲的根、茎、叶、花、叶中脉和叶外缘的第1链cDNA作为模板,荧光定量PCR试剂盒为湖南艾科瑞生物工程有限公司的SYBR Green Pro Taq HS预混型qPCR试剂盒(货号:AG11701),具体操作参照林江波等[29]方法进行,仪器为Roche LightCycler 96。以茎的表达量作为参考因子,采用2–ΔΔCT法计算基因相对表达量。
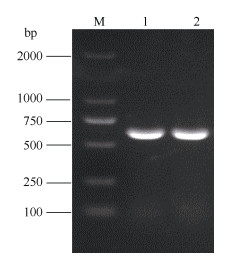
2 结果和分析 2.1 ArCRC基因克隆与编码蛋白理化性质分析以金线莲根、茎、叶和花混合的cDNA为模板,利用引物ArABBY-F和ArABBY-R进行PCR扩增,获得1条约580 bp的特异性条带(图 1)。目的条带测序结果表明克隆到ArCRC基因的开放阅读框, 长度为576 bp (GenBank登录号OR394646),编码191个氨基酸。ProtParam预测ArCRC蛋白的分子量为21.514 kD,理论等电点为9.16,不稳定系数41.12,属于不稳定蛋白。
|
图 1 PCR电泳结果。M: DNA marker 2000; 1, 2: ArCRC基因全长CDS。 Fig. 1 PCR products by electrophoresis. M: DNA marker 2000; 1 and 2: CDS of ArCRC. |
Protein BLAST在线搜索比对,ArCRC蛋白在5~144氨基酸具有YABBY superfamily保守结构域,E-value为1.06×10–56,在107~150氨基酸含有HMG-box_SF superfamily保守结构域,E-value为4.74× 10–4 (图 2)。

|
图 2 ArCRC蛋白保守结构域 Fig. 2 Conserved domains of ArCRC protein |
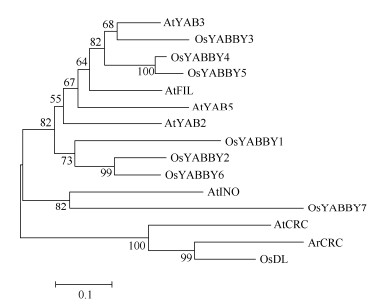
把金线莲ArCRC蛋白与8个水稻和6个拟南芥的YABBY蛋白进行聚类分析,结果表明(图 3),ArCRC与水稻的OsDL和拟南芥的AtCRC聚为一类,亲缘关系最近,属于CRC亚家族,因此将基因命名为ArCRC。
|
图 3 ArCRC与水稻和拟南芥YABBY蛋白聚类分析 Fig. 3 Cluster analysis of ArCRC and YABBY proteins in rice and Arabidopsis |
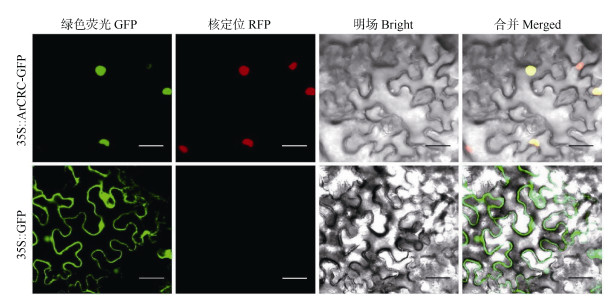
将ArCRC蛋白与植物细胞核定位Marker在烟草叶片中共定位,通过激光共聚焦扫描显微镜观察,ArCRC与植物细胞核定位Marker定位一致(图 4),说明ArCRC定位于细胞核。
|
图 4 ArCRC蛋白在烟草叶片下表皮细胞中的亚细胞定位。标尺=25 µm Fig. 4 Subcellular localization of ArCRC protein in lower epidermal cells of tobacco leaves. Bars=25 µm |
将转化了pGEX-4T-ArCRC的大肠杆菌BL21 (DE3), 37 ℃诱导表达3 h后进行SDS PAGE分析蛋白表达。GST蛋白分子量为26 kD,ArCRC蛋白分子量为21.514 kD,pGEX-4T-ArCRC融合蛋白分子量为47.514 kD。从图 5可见,与诱导前相比,诱导后的E. coil菌液出现1条约47.514 kD的差异条带。蛋白表达产物用GST标签的单克隆抗体进行Western blot检测,诱导表达的蛋白与GST单克隆抗体在47.514 kD处有明显的杂交条带,未诱导的无条带,结果说明ArCRC基因被成功诱导表达。

|
图 5 SDS-PAGE电泳和Western blot检测。M: Marker (10~180 kD); 1: 诱导前; 2: 诱导后。 Fig. 5 SDS-PAGE electrophoresis and western blot detection. M: Marker (10-180 kD); 1: Pre-induction; 2: After induction. |
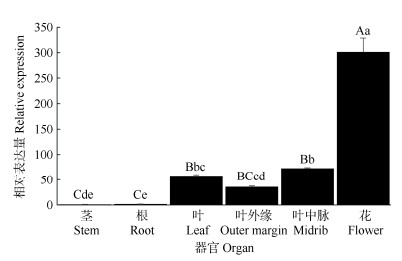
利用qRT-PCR检测ArCRC基因的表达模式, 结果表明(图 6),ArCRC基因在金线莲的根、茎、叶和花中都能检测到表达,花的表达量最高,其次是叶,分别是茎表达量的301.29和57.88倍,达极显著差异;花的表达量是叶片的5.21倍,达极显著差异; 茎和根的表达量很低,差异不显著。ArCRC基因在叶片中脉的表达量显著高于叶片外缘。
|
图 6 ArCRC基因在金线莲器官中的相对表达量。柱上不同大、小写字母分别表示差异极显著(P < 0.01)和差异显著(P < 0.05)。 Fig. 6 Relative expression of ArCRC in organs of Anoectochilus roxburghii. Different capital and small letters upon column indicated significant difference at 0.01 and 0.05 levels, respectively. |
YABBY基因家族是高等植物特有的一类转录因子,主要参与调控植物花器官和叶片发育,CRC是其中的一类亚家族。拟南介AtCRC基因是心皮和蜜腺发育的主要决定因素,CRC功能缺失导致心皮未融合、蜜腺缺失[13, 30]。水稻DL (DROOPING LEAF)基因与拟南芥CRC是同源异型基因,主要和水稻叶片中脉的形成和花的发育相关,水稻DL基因功能缺失突变,心皮同源转化为雄蕊,叶片中脉缺失[31–32]。本研究从金线莲中克隆了ArCRC基因编码区序列,编码的氨基酸序列含有YABBY superfamily和HMG-box_SF superfamily保守结构域,属于不稳定蛋白,本氏烟草瞬时表达确定ArCRC定位于细胞核。将重组原核表达质粒pGEX-4T-ArCRC导入大肠杆菌BL21(DE3),经IPTG诱导表达后, Western blot进一步验证,获得与预测大小一致的GST-ArCRC融合表达蛋白,说明ArCRC基因可以在大肠杆菌中正确表达。序列进化分析表明ArCRC与水稻的OsDL和拟南芥的AtCRC聚为一类,属于CRC亚家族。推测ArCRC与水稻的OsDL和拟南芥的AtCRC具有相似的功能。
CRC基因在植物中的表达具有组织特异性。番茄SlCRCa基因在花瓣和雄蕊中高度表达,响应赤霉素处理,通过影响细胞分裂和细胞扩张来负调控果实和花器官大小[33]。小桐子JcCRC基因仅在花器官中检测到表达[18]。巴西橡胶树HbCRC1基因在叶片中高表达,且在叶片发育过程呈略微上调趋势,HbCRC2基因在雌花中高表达,在叶片中不表达[19]。水稻DL基因在叶原基的中心区域和心皮原基表达,促进叶片中脉的形成和心皮的发育[32]。为了预测金线莲ArCRC基因在金线莲生长发育过程中潜在的功能,本研究利用荧光定量PCR分析了ArCRC基因在不同器官中的表达水平,ArCRC基因在花中表达量最高,其次是叶片,且叶中脉表达量显著高于外缘,花的表达量是叶片的5.21倍,推测金线莲ArCRC基因在金线莲花器官发育中发挥功能,同时还参与调控叶片中脉的形成。
综上,从金线莲中克隆出YABBY家族转录因子CRC亚家族的ArCRC基因,编码191个氨基酸。金线莲ArCRC基因含有YABBY superfamily和HMG-box_SF superfamily保守结构域,属于YABBY家族的CRC亚家族。ArCRC是细胞核定位的转录因子,在金线莲花和叶中脉高表达,表明ArCRC可能在金线莲花发育和叶中脉形成中发挥重要功能。
| [1] |
CHEN Y, WANG W Y, CHEN S N, et al. Changes in kinsenoside content of different strains of Anoectochilus roxburghii at different growth periods[J]. Mod Chin Med, 2021, 23(8): 1423-1429. 陈莹, 王文义, 谌赛男, 等. 不同品系及生长期金线莲的金线莲苷含量变化研究[J]. 中国现代中药, 2021, 23(8): 1423-1429. DOI:10.13313/j.issn.1673‑4890.20200911001 |
| [2] |
XUE M H, LU S K, WENG W, et al. Effects of planting methods on the content of flavonoids in Anoectochilus roxburghii[J]. Fujian Anal Test, 2021, 30(1): 33-36. 薛梅花, 卢石孔, 翁文, 等. 不同种植方式对金线莲黄酮类化合物含量的影响[J]. 福建分析测试, 2021, 30(1): 33-36. DOI:10.3969/j.issn.1009-8143.2021.01.07 |
| [3] |
CHEN C L, LI W L, AN X, et al. Effects of light intensities on polysaccharide and flavonoid contents in Anoectothenia roxburghii[J]. Mol Plant Breed, 2023, 21(15): 5103-5109. 陈常理, 李文略, 安霞, 等. 光照强度对金线莲多糖和黄酮类物质含量的影响[J]. 分子植物育种, 2023, 21(15): 5103-5109. DOI:10.13271/j.mpb.021.005103 |
| [4] |
TANG F, ZHANG X Q, XU J T, et al. Screening on hypoglycemic effective part of Anoectochilus roxburghii[J]. Chin Trad Herb Drugs, 2011, 42(2): 340-342. 唐菲, 张小琼, 徐江涛, 等. 金线莲降血糖活性部位的筛选[J]. 中草药, 2011, 42(2): 340-342. |
| [5] |
ZHANG X Y, YU X L, YE H H. Research progress on hepatoprotective effect and mechanism of Anoectochilus roxburghii (Wall.) Lindl.[J]. Mod Chin Med, 2022, 24(5): 920-925. 张晓颖, 俞晓玲, 叶寒辉. 金线莲保肝作用及作用机制研究进展[J]. 中国现代中药, 2022, 24(5): 920-925. DOI:10.13313/j.issn.1673-4890.20210223004 |
| [6] |
LÜ J G, ZHANG H, ZHOU H H, et al. Effects of kinsenoside on the proliferation, migration and invasion of human hepatoma HepG2 cells[J]. J Hubei Univ Sci Technol (Med Sci), 2022, 36(1): 5-8. 吕建国, 张晗, 周荟慧, 等. 金线莲苷对人肝癌HepG2细胞增殖、迁移和侵袭的影响[J]. 湖北科技学院学报(医学版), 2022, 36(1): 5-8. DOI:10.16751/j.cnki.2095-4646.2022.01.005 |
| [7] |
XU S S, LI Y Q, FENG H M, et al. Study on the formulation optimization technology of 'Hongxia' Anoectochilus roxburghii tissue culture medium[J]. Anhui Agric Sci Bull, 2021, 27(24): 15-17. 徐双双, 李怡清, 冯慧敏, 等. '红霞'金线莲组培培养基配方优化技术研究[J]. 安徽农学通报, 2021, 27(24): 15-17. DOI:10.3969/j.issn.1007-7731.2021.24.005 |
| [8] |
SHI M R, LUO Y F. Study on the establishment and optimization of the sterile system of wild Anoectochilus roxburghii from eastern Fujian Province[J]. J Anhui Agric Sci, 2022, 50(7): 96-98. 施满容, 罗义发. 闽东野生金线莲无菌体系建立与优化研究[J]. 安徽农业科学, 2022, 50(7): 96-98. DOI:10.3969/j.issn.0517-6611.2022.07.022 |
| [9] |
XING B C, SU L Y, WAN S Q, et al. Screening and cloning analysis of WRKY transcription factor regulating embryo development from Anoectochilus roxburghii[J]. Chin Trad Herb Drugs, 2022, 53(12): 3745-3754. 邢丙聪, 苏立样, 万思琦, 等. 金线莲中调控胚胎发育WRKY转录因子筛选及克隆分析[J]. 中草药, 2022, 53(12): 3745-3754. DOI:10.7501/j.issn.0253-2670.2022.12.021 |
| [10] |
CHEN Y Q, LI Q J, LAI Z X, et al. Characteristics of calcium distribution in the developing anthers of Anoectochilus roxburghii[J]. Acta Bot Boreali-Occid Sin, 2021, 41(6): 977-983. 陈育青, 李秋静, 赖钟雄, 等. 金线莲花药发育中的钙离子分布特征[J]. 西北植物学报, 2021, 41(6): 977-983. DOI:10.7606/j.issn.1000-4025.2021.06.0977 |
| [11] |
CHEN Y Q, HUANG Z H, LI Q J, et al. Microstructure of anther development of Anoectochilus roxburghii[J]. J Chin Elect Microsc Soc, 2021, 40(4): 425-431. 陈育青, 黄泽豪, 李秋静, 等. 福建濒危药用植物金线莲花药发育的形态观察[J]. 电子显微学报, 2021, 40(4): 425-431. DOI:10.3969/j.issn.1000-6281.2021.04.011 |
| [12] |
CHEN Y Q, LIN H Q, LIN M Z, et al. Distribution characteristics of polysaccharides and lipids in the developing anther of Anoectochilus roxburghii[J]. Acta Bot Boreali-Occid Sin, 2020, 40(8): 1333-1338. 陈育青, 林汉钦, 林美珍, 等. 金线莲花药发育中多糖和脂滴的分布特征[J]. 西北植物学报, 2020, 40(8): 1333-1338. DOI:10.7606/j.issn.1000-4025.2020.08.1333 |
| [13] |
BOWMAN J L, SMYTH D R. CRABS CLAW, a gene that regulates carpel and nectary development in Arabidopsis, encodes a novel protein with zinc finger and helix-loop-helix domains[J]. Development, 1999, 126(11): 2387-2396. DOI:10.1242/dev.126.11.2387 |
| [14] |
SIEGFRIED K R, ESHED Y, BAUM S F, et al. Members of the YABBY gene family specify abaxial cell fate in Arabidopsis[J]. Development, 1999, 126(18): 4117-4128. DOI:10.1242/dev.126.18.4117 |
| [15] |
DONG H, LI Y X, WANG T K, et al. Genome-wide identification and expression analysis of YABBY gene family in rice[J]. Mol Plant Breed, 2020, 18(15): 4845-4854. 董皓, 李懿星, 王天抗, 等. 水稻YABBY家族的全基因组鉴定与表达分析[J]. 分子植物育种, 2020, 18(15): 4845-4854. DOI:10.13271/j.mpb.018.004845 |
| [16] |
XU Y, QIAO Z L, ZHAO L, et al. Genome-wide and transcriptome analysis of YABBY family gene in Lotus (Nelumbo nucifera Gaertn.)[J]. Mol Plant Breed, 2022, 20(22): 7342-7353. 徐逸, 谯正林, 赵琳, 等. 荷花YABBY家族的全基因组和转录组分析[J]. 分子植物育种, 2022, 20(22): 7342-7353. DOI:10.13271/j.mpb.020.007342 |
| [17] |
LIU X B, WANG S J, SUN J Y, et al. Identification and expression analysis of YABBY gene family in Panax ginseng C. A. Meyer[J]. J Chin Med Mat, 2021, 44(12): 2970-2975. 刘秀波, 王思嘉, 孙嘉莹, 等. 人参中YABBY基因家族鉴定与表达分析[J]. 中药材, 2021, 44(12): 2970-2975. DOI:10.13863/j.issn1001-4454.2021.12.044 |
| [18] |
ZHANG Z S, GONG M, WU D D, et al. Genome-wide characterization, expression profiles and alternative splicing events of YABBY family genes in Jatropha curcas[J]. J Trop Subtrop Bot, 2023, 31(2): 249-262. 张作胜, 龚明, 吴丹丹, 等. 小桐子YABBY全基因组家族成员的鉴定、表达和可变剪接分析[J]. 热带亚热带植物学报, 2023, 31(2): 249-262. DOI:10.11926/jtsb.4570 |
| [19] |
ZHANG H T, XIAO X H, YANG J H, et al. Identification and expression analysis of YABBY gene family in Hevea brasiliensis[J]. Chin J Trop Crops, 2022, 43(11): 2188-2198. 张鸿韬, 肖小虎, 阳江华, 等. 巴西橡胶树YABBY基因家族鉴定及表达分析[J]. 热带作物学报, 2022, 43(11): 2188-2198. DOI:10.3969/j.issn.1000-2561.2022.11.002 |
| [20] |
MIAO J Q, HUANG Y, NING R, et al. Bioinformatics analyses on YABBY gene family in Populus trichocarpa[J]. Mol Plant Breed, 2023, 21(10): 3245-3252. 苗嘉琪, 黄颖, 宁蕊, 等. 毛果杨YABBY基因家族生物信息学分析[J]. 分子植物育种, 2023, 21(10): 3245-3252. DOI:10.13271/j.mpb.021.003245 |
| [21] |
ZHANG X Y, LIU X. Research progress of transcription factors and leaf development[J]. Plant Physiol J, 2022, 58(1): 91-100. 张雪莹, 刘欣. 转录因子与叶片发育的研究进展[J]. 植物生理学报, 2022, 58(1): 91-100. DOI:10.13592/j.cnki.ppj.2021.0253 |
| [22] |
FOURQUIN C, PRIMO A, MARTÍNEZ-FERNÁNDEZ I, et al. The CRC orthologue from Pisum sativum shows conserved functions in carpel morphogenesis and vascular development[J]. Ann Bot, 2014, 114(7): 1535-1544. DOI:10.1093/aob/mcu129 |
| [23] |
STRABLE J, VOLLBRECHT E. Maize YABBY genes drooping leaf1 and drooping leaf2 regulate floret development and floral meristem determinacy[J]. Development, 2019, 146(6): dev171181. DOI:10.1242/dev.171181 |
| [24] |
CHE G, PAN Y P, LIU X F, et al. Natural variation in CRABS CLAW contributes to fruit length divergence in cucumber[J]. Plant Cell, 2023, 35(2): 738-755. DOI:10.1093/plcell/koac335 |
| [25] |
TAMURA K, STECHER G, PETERSON D, et al. MEGA6: Molecular evolutionary genetics analysis version 6.0[J]. Mol Biol Evol, 2013, 30(12): 2725-2729. DOI:10.1093/molbev/mst197 |
| [26] |
LIN J B, WANG W Y, LI H M, et al. Cloning and expression analysis of NtPLATZ1 gene from Narcissus tazetta var. chinensis[J]. J NW A&F Univ (Nat Sci), 2016, 44(10): 165-170. 林江波, 王伟英, 李海明, 等. 中国水仙锌指蛋白NtPLATZ1的克隆与表达分析[J]. 西北农林科技大学学报(自然科学版), 2016, 44(10): 165-170. DOI:10.13207/j.cnki.jnwafu.2016.10.023 |
| [27] |
JIAO Y, QU M, GUI B B, et al. Prokaryotic expression and western blot identification of FUT10 protein in Crassostrea gigas[J]. J Food Saf Qual, 2022, 13(8): 2573-2579. 教杨, 曲梦, 桂彬彬, 等. 太平洋牡蛎中类FUT10蛋白的原核表达及免疫印迹鉴定[J]. 食品安全质量检测学报, 2022, 13(8): 2573-2579. DOI:10.19812/j.cnki.jfsq11-5956/ts.2022.08.023 |
| [28] |
LIN J B, WANG W Y, ZOU H, et al. Expression stabilities of three housekeeping genes of Anoectochilus roxburghii[J]. Fujian J Agric Sci, 2018, 33(11): 1125-1129. 林江波, 王伟英, 邹晖, 等. 金线莲3个持家基因表达稳定性分析[J]. 福建农业学报, 2018, 33(11): 1125-1129. DOI:10.19303/j.issn.1008-0384.2018.11.001 |
| [29] |
LIN J B, ZOU H, WANG W Y, et al. Cloning and expressions of LIS in Dendrobium officinale[J]. Fujian J Agric Sci, 2020, 35(10): 1071-1077. 林江波, 邹晖, 王伟英, 等. 基因克隆与茉莉酸甲酯诱导表达分析[J]. 福建农业学报, 2020, 35(10): 1071-1077. DOI:10.19303/j.issn.1008-0384.2020.10.004 |
| [30] |
LEE J Y, BAUM S F, OH S H, et al. Recruitment of CRABS CLAW to promote nectary development within the eudicot clade[J]. Development, 2005, 132(22): 5021-5032. DOI:10.1242/dev.02067 |
| [31] |
YAMAGUCHI T, NAGASAWA N, KAWASAKI S, et al. The YABBY gene DROOPING LEAF regulates carpel specification and midrib development in Oryza sativa[J]. Plant Cell, 2004, 16(2): 500-509. DOI:10.1105/tpc.018044 |
| [32] |
OHMORI Y, TORIBA T, NAKAMURA H, et al. Temporal and spatial regulation of DROOPING LEAF gene expression that promotes midrib formation in rice[J]. Plant J, 2011, 65(1): 77-86. DOI:10.1111/j.1365-313X.2010.04404.x |
| [33] |
YANG T W, HE Y, NIU S B, et al. A YABBY gene CRABS CLAW a (CRCa) negatively regulates flower and fruit sizes in tomato[J]. Plant Sci, 2022, 320: 111285. DOI:10.1016/j.plantsci.2022.111285 |
 2025, Vol. 33
2025, Vol. 33



