2. 福建省农业科学院果树研究所, 福州 350013;
3. 福建省落叶果树工程技术研究中心, 福州 350013
2. Fruit Research Institute, Fujian Academy of Agricultural Sciences, Fuzhou 350013, China;
3. Research Centre for Engineering Technology of Fujian Deciduous Fruits, Fuzhou 350013, China
几丁质酶(Chitinase, CHi)是以N-乙酰-d-氨基葡萄糖为单体,通过β-1, 4-糖苷键连接而成的高分子多聚物[1],能够水解几丁质、壳聚糖、脂壳寡糖、肽聚糖、阿拉伯半乳聚糖和含有N-乙酰葡糖胺的糖苷水解酶[2]。根据催化结构域不同,几丁质酶分为糖苷水解酶家族(glycoside hydrolase family) 18 (GH18)和糖苷水解酶家族19 (GH19)[3–4]。GH18成员广泛存在于植物、动物、细菌、真菌和病毒等生物体中, 而GH19成员只存在于植物和链霉菌属中[5]。
目前,拟南芥(Arabidopsis thaliana)[6]、棉花(Gossypium sp.)[7]、水稻(Oryza sativa)[8]、辣椒(Cap-sicum annuum)[9]等植物的几丁质酶家族基因已被鉴定。CHi基因呈组织器官特异性表达,参与植物抵御外界胁迫过程[10]和生长发育[11]调控过程。研究表明植物受到病原菌[12]、虫害[13]、干旱[14]、低温[8]等胁迫时,CHi的表达量会显著增加。香蕉(Musa nana)果实采后CHi的表达与乙烯释放量的变化趋势一致,说明CHi对采后香蕉果实的成熟有影响[15]。在桃(Prunus persica)果实发育过程中,CHi的VIGS沉默会导致果实硬度和纤维素含量降低,影响果实纤维素的生物合成,在保持果实硬度方面起着重要作用[16]。
猕猴桃(Actinidia chinensis)为猕猴桃科(Actinidiaceae)猕猴桃属落叶木质藤本果树,在我国栽培范围较广。生产上猕猴桃作为经济作物具有极高的生产价值,然而猕猴桃是典型的呼吸跃变型果实,有明显的生理后熟过程,采后生理变化强烈影响果实的贮藏期限和果实品质[17]。在猕猴桃果实贮藏过程中,外源ABA处理会促进采后果实生成乙烯,并加速果实软化[18],而低温贮藏可以通过降低果实的呼吸作用,延缓果实的软化速度[19]。研究表明,CHi对果实贮藏过程中的成熟软化有重要作用[15–16],但猕猴桃几丁质酶家族基因鉴定的研究尚未见详细报道。鉴于此,本研究从猕猴桃中鉴定到18个几丁质酶基因(AcCHis),并对其理化特性、系统进化关系、保守结构域、基因结构、蛋白质结构、靶向miRNA以及在猕猴桃不同组织部位及果实贮藏软化过程中的表达特征进行分析,以期为解析AcCHis在猕猴桃果实贮藏过程中的作用奠定理论基础。
1 材料和方法 1.1 AcCHis基因的鉴定从猕猴桃基因组数据库(https://plants.ensembl.org/Actinidia_chinensis/Info/Index)下载蛋白序列。同时从Pfam数据库(https://www.ebi.ac.uk/interpro/)获取几丁质酶GH18 (PF00704)和GH19 (PF00182)隐马尔科夫模型(HMM),利用HMM Search筛选含GH18和/或GH19结构域的蛋白序列(E<1e-10)。进一步通过NCBI-CDD (https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)数据库验证其保守结构域,删除结构域缺失的序列,获得AcCHis氨基酸序列。利用在线工具Expasy (https://www.expasy.org/)对AcCHis蛋白进行理化性质分析,并使用WoLF PSORT (https://wolfpsort.hgc.jp/)分析其亚细胞定位。
1.2 AcCHis的系统进化、保守结构域和其基因结构通过MEGA11软件的邻接法(Neighbour-Joining, NJ)对拟南芥[6]、桑(Morus alba)[20]的CHi和AcCHis氨基酸序列构建系统进化树,自展值(bootstrap)参数设置为1 000。利用MEME (http://meme-suite.org/tools/meme)分析AcCHis氨基酸序列的基序特征,最大基序数设为20,其他设定为默认参数。根据猕猴桃基因组的注释信息GFF文件,通过TBtools软件将AcCHis的系统进化分析结果、保守结构域及其基因结构进行可视化。
1.3 AcCHis蛋白结构的预测分析分别通过SOPMA (NPS@: SOPMA secondary structure prediction (ibcp.fr))和SWISS-MODEL (https://swissmodel.expasy.org/)构建AcCHis蛋白的2D、3D结构。
1.4 AcCHis基因启动子区的顺式作用元件分析从猕猴桃基因组中提取AcCHis起始密码子上游2 kb的序列,并利用在线软件PlantCare (https://bioinformatics.psb.ugent.be/webtools/plantcare/html/)和BDGP (https://www.fruitfly.org)分析顺势作用元件。
1.5 AcCHis基因染色体定位、共线性和同源性分析利用Circos软件绘制基因分布图,使用本地Blast对猕猴桃的AcCHis氨基酸序列文件进行建库, 以此氨基酸序列文件作为query序列进行blastp比对,设置evalue为1e-10,利用MCScanX软件(http://chibba.pgml.uga.edu/mcscan2/)获得CHi在猕猴桃基因组中的复制类型,并对AcCHis进行共线性分析,使用TBtools计算AcCHis基因复制中Ka/Ks值,并用Circos进行可视化[21–22]。
为探索AcCHis在双子叶植物中的同源关系,分别下载猕猴桃(https://plants.ensembl.org/Actinidia_chinensis/Info/Index)、番茄(Lycopersicon esculentum) (https://data.jgi.doe.gov/refine-download/phytozome?genome_id=390&_gl=1*1nss0sx*_ga*MjU5NDc3NDU4LjE2ODE5NTYzMTE.*_ga_YBLMHYR3C2*MTcwMjc3OTg3My45LjAuMTcwMjc3OTg3My4wLjAuMA)和拟南芥(http://plants.ensembl.org/Arabidopsis_thaliana/Info/Index)的DNA序列文件和注释信息文件, 通过TBtools软件分析基因同源性,并将结果可视化。
1.6 靶向AcCHis基因的miRNA预测使用psRNATarget (https://www.zhaolab.org/psRNATarget/analysis)对调控AcCHis的miRNA进行分析(参数期望值设置为5,其他为默认参数)。
1.7 AcCHis基因的表达分析采集健康猕猴桃植株的茎、叶、花、幼果、成熟果,进行转录组测序,分析AcCHis基因在不同组织部位的表达特征。以无病虫害和无损伤的成熟果实(花后160 d)为材料,分别置于室温贮藏0周(对照,CK);4 ℃贮藏1周(Four-1W)、2周(Four-2W)、3周(Four-3W);室温(25 ℃)下贮藏1周(RT-1W)、50 mg/L ABA浸泡2 min后25 ℃贮藏1周(ABA-1W),随后进行转录组测序,分析AcCHis家族基因在猕猴桃果实后熟软化过程中的表达。RNA的提取按照天根生化科技(北京)有限公司的多糖多酚植物总RNA提取试剂盒说明书操作,样品委托天津诺禾致源生物信息科技有限公司进行转录组测序。从猕猴桃转录组数据中提取AcCHis基因的FPKM值,进行log2转换后绘制表达量聚类热图。
2 结果和分析 2.1 AcCHis基因的鉴定和序列特征分析使用隐马尔可夫模型(HMM)筛选猕猴桃基因组的CHi家族成员,并利用NCBI-CDD数据库验证初筛成员的保守结构域,保留至少1个完整CHi保守结构域,最终共鉴定到18个AcCHis基因,根据它们在染色体上的顺序和位置,分别命名为AcCHi1~AcCHi18 (表 1)。
| 表 1 AcCHis的理化性质 Table 1 Physicochemical properties of AcCHis |
18个AcCHis基因分别编码272~428个氨基酸, 理化性质分析表明,分子量为29.13~47.28 kD,等电点为4.48~8.47,其中12个成员为酸性蛋白,6个成员为碱性蛋白。AcCHis多为稳定亲水性蛋白, 只有AcCHi9、AcCHi16和AcCHi17为不稳定蛋白; AcCHi3、AcCHi7和AcCHi18为疏水性蛋白。亚细胞定位预测表明,18个AcCHis基因中有12个定位于细胞外、2个定位于细胞核、2个定位于叶绿体、各有1个定位于过氧化物酶体和细胞骨架(表 1)。
2.2 AcCHis的系统发育、结构特征和保守结构域将AcCHis与其他物种的CHi氨基酸序列构建系统进化树(图 1),结果表明18个AcCHis分别聚在GH18和GH19分支,每个分支又可分为3小组,共6个小组:GH18-a组(4个成员)、GH18-b组(5)、GH18-c组(2)、GH19-f组(1)、GH19-g组(2)、GH19-h组(4)。
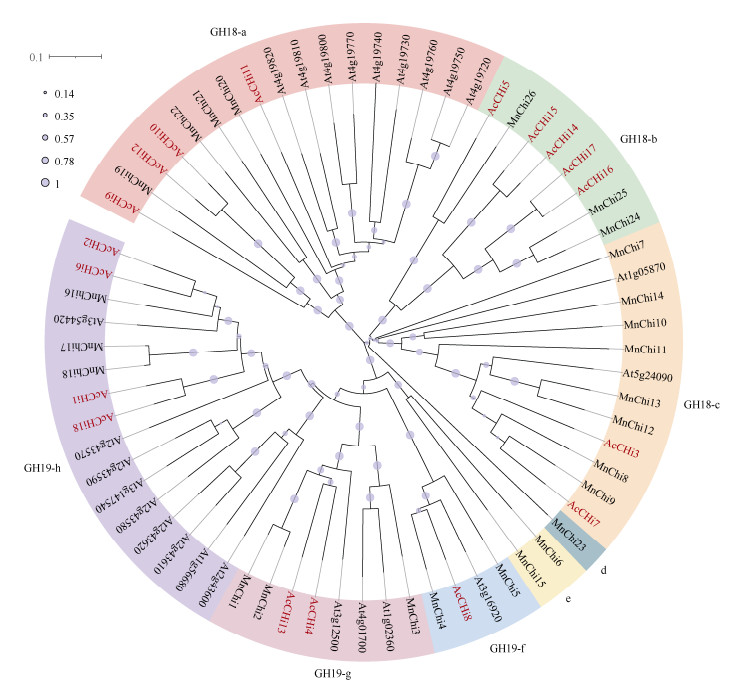
|
图 1 拟南芥(At)、桑(Mn)、猕猴桃(Ac)的CHis系统进化树 Fig. 1 Phylogenetic tree of CHis in Arabidopsis thaliana (At), Morus alba (Mn), and Actinidia chinensis (Ac) |
对AcCHis进行蛋白保守结构域分析表明(图 2: A),系统进化树上各组成员所含保守基序存在差异。值得注意的是GH19-f、GH19-g、GH19-h组成员间的保守基序种类、数量、位置差异较小,这与系统进化树分析结果一致,且GH19-g、GH19-h组成员都含有GH19-f组成员的全部保守结构域。
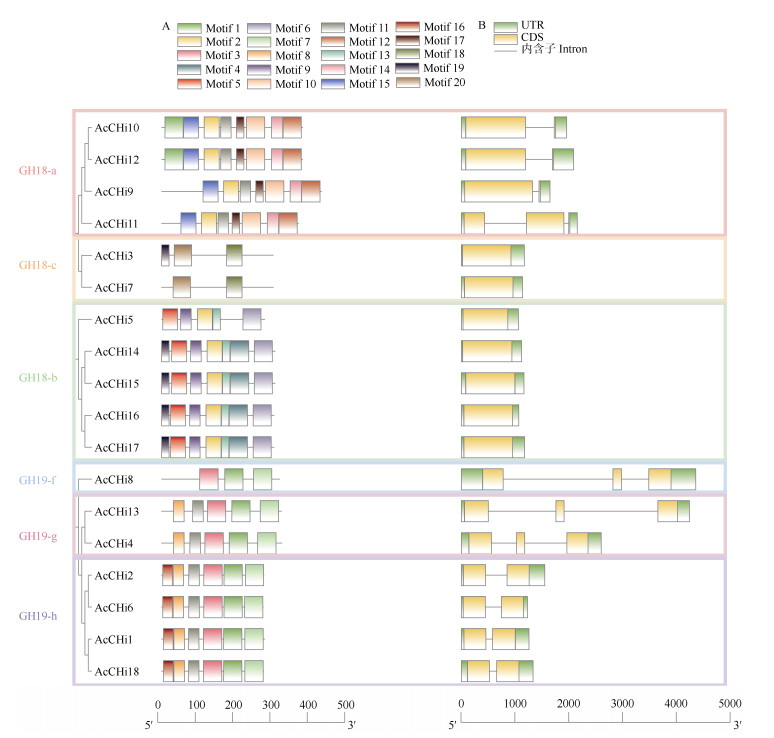
|
图 2 AcCHis的保守结构域(A)和AcCHis基因结构(B) Fig. 2 Conservative domain of AcCHis (A) and gene structure of AcCHis (B) |
基因结构在基因家族的进化中起关键作用[23], AcCHis基因结构可视化结果表明(图 2: B),GH18成员含有1~3个外显子,有7个成员只有1个外显子。此外,AcCHi11含有2个内含子,3个外显子。GH19成员含有2~3个外显子,其中,AcCHi8、AcCHi13、AcCHi4基因内含子长度比较长。可见, GH18与GH19成员AcCHis蛋白的保守结构域和AcCHis基因的外显子、内含子结构具有一定相似性。
2.3 AcCHis蛋白质结构的构建和分析二级结构预测结果表明(表 2),AcCHis二级结构主要由α螺旋、β折叠、无规卷曲、延伸链等组成,各成员上述二级结构分别占24.92%~38.16%、4.28%~7.69%、36.54%~58.88%、10.66%~22.55%。GH18和GH19成员间的α螺旋和β折叠结构比例差异较小,但GH18延伸链占比明显大于GH19, 而GH19中无规卷曲较GH18稍多。进一步分析表明,GH18、GH19成员的蛋白质三级结构存在差异(图 3),这与蛋白质二级结构预测结果一致。
| 表 2 AcCHis蛋白的二级结构预测 Table 2 AcCHis protein secondary structure prediction |
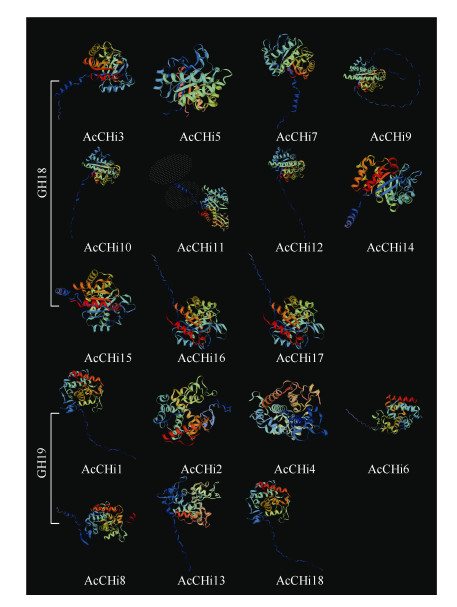
|
图 3 AcCHis蛋白三级结构预测 Fig. 3 AcCHis protein tertiary structure prediction |
为进一步分析AcCHis基因的功能,对起始密码子上游2 kb区域的启动子序列进行顺式元件分析, 结果表明AcCHis所含顺式元件主要涉及激素响应、非生物胁迫应答和生长发育调控3类(图 4)。

|
图 4 AcCHis基因启动子区域的顺式作用元件 Fig. 4 cis-Acting elements in promoter region of AcCHi genes |
激素响应元件主要有5种,分别为脱落酸响应元件(ABRE)、茉莉酸响应元件(CGTCA/TGACG-motif)、赤霉素响应元件(GARE和P/TATC-box-motif)、生长素响应元件(AuxRR-core和TGA-element)和水杨酸响应元件(TCA-element)。10个成员存在ABRE, 以AcCHi13最多(7个);13个成员存在CGTCA/TGACG-motif,以AcCHi11和AcCHi18最多(均6个);10个成员存在GARE和P/TATC-box-motif,AcCHi10和AcCHi12最多(均2个);8个成员存在AuxRR-core和TGA-element,以AcCHi12最多(3个);8个成员存在TCA-element,以AcCHi4最多(3个)。
非生物胁迫应答元件共6种,分别为厌氧诱导元件(ARE)、低温响应元件(LTR)、防御与胁迫响应元件(TC-rich repeats)、干旱诱导响应元件(MBS)、缺氧诱导元件(GC-motif)和昼夜节律响应元件(circadian)。16个成员存在ARE;5个成员存在LTR;6个成员存在TC-rich repeats;5个成员存在MBS。此外,只有AcCHi11存在GC-motif;AcCHi4存在circadian。
生长发育相关作用元件共4种,6个成员存在分生组织表达元件(CAT-box),6个成员存在玉米醇溶蛋白代谢调节元件(O2-site),4个成员存在胚乳表达元件(GCN4_motif),5个成员存在种子特异性调控元件(RY-element)。
为探究AcCHis在双子叶植物中的进化特征, 分析了猕猴桃与番茄[24]和拟南芥[6]中CHis基因的共线性(图 5)。结果表明AcCHis与番茄和拟南芥基因组中多个CHis基因存在共线性关系,其中10个AcCHis基因(AcCHi1、AcCHi5、AcCHi6、AcCHi7、AcCHi10、AcCHi11、AcCHi12、AcCHi13、AcCHi14和AcCHi15)与10个番茄CHis基因具有共线性; AcCHi1与1个拟南芥CHi基因具有共线性。
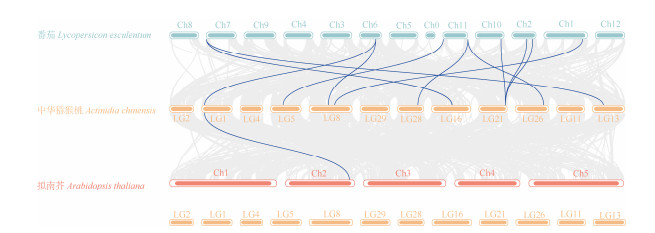
|
图 5 猕猴桃、番茄和拟南芥中CHis基因同源性分析 Fig. 5 Homology analysis of CHi genes in Actinidia chinensis, Lycopersicon esculentum and Arabidopsis thaliana |
从图 6可见,18个AcCHis不均匀地分布在12条染色体上,其中在LG8、LG28染色体上分布的AcCHis基因最多(均为3个)。27.78%的AcCHis基因发生片段重复。4对片段复制基因对为AcCHi10与AcCHi11、AcCHi14与AcCHi15、AcCHi10与PSS36148和AcCHi13与PSS29947,其中PSS36148、PSS29947缺失几丁质酶保守结构域,因而未进行命名。
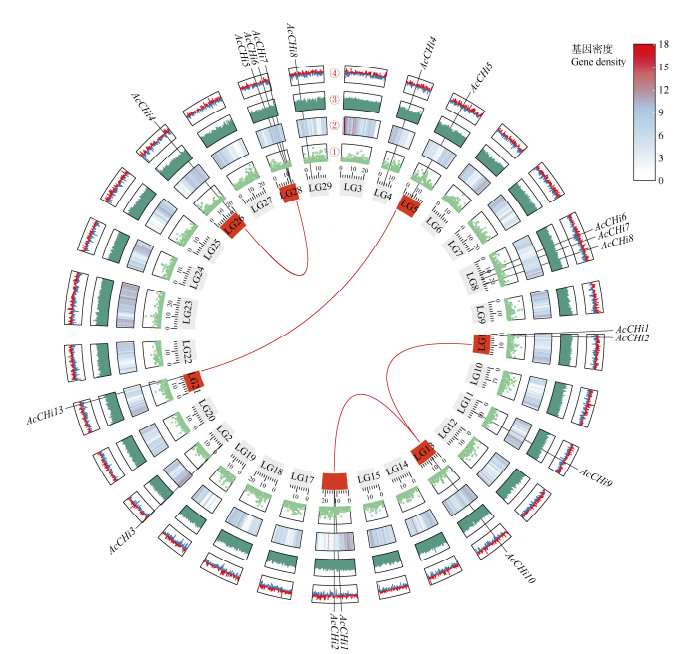
|
图 6 AcCHis基因在染色体上的分布。①: 未知碱基分布; ②: 染色体基因密度; ③: GC含量; ④: GC偏移。 Fig. 6 Chromosome distribution of AcCHis. ①: Unknown base distribution; ②: Gene density of chromosome; ③: GC content; ④: GC skew. |
为分析AcCHis基因家族复制过程中的选择压力和发散率,对该家族中的重复基因序列进行了Ka (非同义置换)与Ks (同义置换)值的计算。从AcCHis的Ka/Ks来看,2对同源基因对的Ka/Ks值(0.088 212、0.306 616)均小于1,说明所有AcCHis重复基因在进化选择中受到反向纯化选择,很大程度上受到进化选择压力。
2.6 AcCHis基因的miRNA预测分析进一步分析AcCHis受miRNA的调控情况(表 3),AcCHi12是3个ach-miR172成员(ach-miR172a-5p、ach-miR172b-5p、ach-miR172j)的靶基因;而ach-miR408家族(ach-miR408-5p、ach-miR408b、ach-miR408d)靶向3个AcCHis基因(AcCHi4、AcCHi16、AcCHi17)。其中,ach-miR408成员在AcCHi16、AcCHi17上的靶位点一致,即ach-miR408b、ach-miR408d同时调控AcCHi16、AcCHi17 2个基因。
| 表 3 AcCHis基因的miRNA预测分析 Table 3 Prediction analysis of AcCHi genes miRNA |
从图 7可见,AcCHis的GH18、GH19成员间的转录水平差异明显。GH18成员中,4个基因(AcCHi5、AcCHi9、AcCHi14和AcCHi15)在各组织中不表达或表达量较低;7个基因(AcCHi3、AcCHi7、AcCHi10、AcCHi11、AcCHi12、AcCHi16和AcCHi17)在不同组织中呈现差异表达,其中AcCHi10在叶中最高(是成熟果的65倍),其次是茎,而在幼果与花中较低(仅为成熟果的1倍)。GH19成员中,2个基因(AcCHi4和AcCHi8)在各组织中高表达;5个基因(AcCHi1、AcCHi2、AcCHi6、AcCHi8和AcCHi18)在不同组织中呈现差异表达,均在叶片高表达;成熟果中AcCHi18的表达比幼果、叶、茎、花中低,其在花中最高(是成熟果的108倍);而成熟果中AcCHi13的表达比幼果、叶、茎、花中高,其在茎中最低(仅为成熟果的10%)。
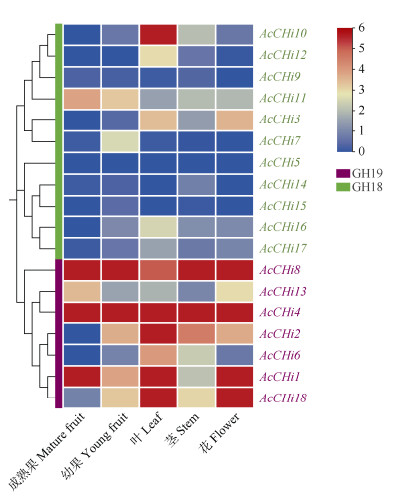
|
图 7 AcCHis基因在不同组织中的表达模式 Fig. 7 Expression patterns of AcCHi genes in different tissues |
从图 8可见,GH18和GH19的AcCHis成员间表达差异明显。GH18成员中,10个基因(AcCHi3、AcCHi5、AcCHi7、AcCHi9、AcCHi10、AcCHi12、AcCHi14、AcCHi15、AcCHi16和AcCHi17)在果实贮藏过程不表达或表达量较低。与对照(室温贮藏0周)相比,AcCHi11在室温25 ℃贮藏1周、ABA浸泡后25 ℃贮藏1周和4 ℃贮藏1~3周的果实中均表达下调,其中ABA浸泡后25 ℃贮藏1周的果实中AcCHi11表达量显著下降(仅为CK的7%)。GH19成员中,2个基因(AcCHi2和AcCHi6)在果实贮藏过程中不表达或表达量较低,其余5个基因(AcCHi1、AcCHi4、AcCHi8、AcCHi13和AcCHi18)呈现差异表达,其中AcCHi8在25 ℃贮藏1周和4 ℃贮藏1~3周均表达上调,以4 ℃贮藏3周的最大(为CK的188倍),而ABA浸泡后25 ℃贮藏1周表达下调。这表明AcCHis在果实后熟软化过程的表达水平存在差异,随贮藏时间、贮藏温度和外源激素呈动态变化,说明AcCHis可能影响猕猴桃果实的后熟软化过程。
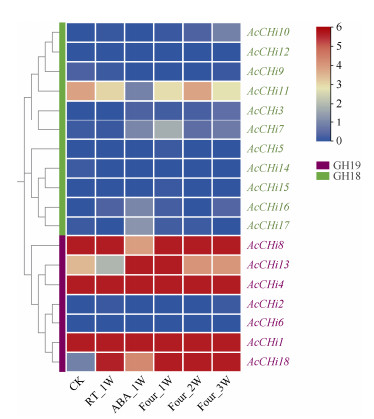
|
图 8 成熟果实贮藏过程中AcCHis基因的表达模式。CK: 室温贮藏0周; RT_1W: 25 ℃ 1周; ABA_1W: 50 mg/L ABA浸泡2 min后25 ℃ 1周; Four_1W: 4 ℃ 1周; Four_2W: 4 ℃ 2周; Four_3W: 4 ℃ 3周。 Fig. 8 Expression pattern of AcCHis gene during mature fruit storage. CK: Storage at room temperature for 0 weeks; RT_ 1W: 25 ℃ for 1 week; ABA_ 1W: 25 ℃ for 1 week after soaking 50 mg/L ABA for 2 min; Four_ 1W: 4 ℃ for 1 week; Four_ 2W: 4 ℃ for 2 weeks; Four_ 3W: 4 ℃ for 3 weeks. |
本研究从猕猴桃基因组中共鉴定出18个AcCHis基因, 相比苹果(Malus pumila, 83个)[25]、桑(26个)[20]、番茄(43个)、拟南芥(24个)较少,系统进化树分析表明,18个AcCHis可分为6组。AcCHis保守结构域、基因结构在GH18和GH19成员间存在较高相似性,说明AcCHis基因之间存在功能相似性。
猕猴桃和番茄的AcCHis主要在9条染色体上分布有12对共线同源基因组,猕猴桃的LG1染色体与拟南芥2号染色体间有1对为共线同源基因组, 说明AcCHis基因家族与番茄、拟南芥都具有共线性关系,AcCHis基因在双子叶物种中存在较高的同源性,且在进化过程中具有一定保守性。
基因复制事件是影响植物进化、基因扩增的关键因素,分为串联复制事件和片段复制事件[26]。不同植物中几丁质酶基因家族数量差异明显[27–29],基因复制事件在此过程中具有重要作用。AcCHis基因在染色体上呈不均匀分布,表明进化过程中AcCHis基因通过大片段复制来扩增基因数量。其他植物中也存在基因家族通过大片段复制以扩张的现象,如烟草(Nicotiana tabacum)的61个Hsp70基因中,至少15对发生了基因片段复制和串联重复[30];睡莲(Nymphaea)的69个WRKY家族中发生了16个基因复制事件[31],同样胡桃(Juglans regia)的102个bHLH基因家族成员中,有42个通过基因复制参与了bHLH基因家族的扩增[32]。本研究中AcCHis基因也存在基因复制现象,27.78%的AcCHis基因发生片段重复, 推测AcCHis成员在猕猴桃中的扩增主要是以片段复制为主。结合AcCHis重复基因序列的Ka与Ks值, 表明2对同源基因对在进化选择中受到反向纯化选择,在很大程度上受到了净化选择压力。
3.2 AcCHis在植物生长发育和果实贮藏过程中具有重要作用植物作为固着生物,在生长发育过程中,通过漫长的进化获得了复杂的基因调控机制[33–34]。而顺式作用元件在决定基因表达调控上具有关键性作用[35]。本研究表明,AcCHis成员所含顺式元件主要涉及激素响应、非生物胁迫应答和生长发育调控3类。这与其他物种的CHis功能一致,外源乙烯会加速香蕉果实Ⅲ类酸性几丁质酶基因的下调表达并使其活性下降,Ⅲ类酸性几丁质酶基因可能参与香蕉果实采后成熟的激素调控[36]。草莓(Fragaria × ananassa)果实受到干旱胁迫后,FaChi1~FaChi4的表达均被诱导,说明其在果实对生物及非生物胁迫的响应中有一定调节作用[37]。VvChi4在葡萄(Vitis vinifera)开花后10周时首次表达,且在开花后12~16周的果实中高度表达,表明VvChi4在葡萄成熟过程中诱导了几丁质酶活性[38]。AcCHis基因家族成员可能参与了猕猴桃相关生理过程的调控。
植物miR172和miR408可以通过调节靶基因的表达进而参与植物生长发育、激素响应等多种生理代谢调控过程。有研究表明,miR172过表达会影响番茄花器官的发育以及乙烯的生物合成[39];小麦(Triticum aestivum)中miR172及其靶基因Q (AP2L5)变化会影响颖片向外过渡程度进而影响开花[40];马铃薯(Solanum tuberosum)中miR172在根与块茎发育阶段显著表达,表明miR172可能在马铃薯根与块茎发育中发挥作用[41]。苹果中miR408a过表达会显著增加与Cu结合转运相关基因的表达,导致苹果植株与果实积累花青素[42]。龙眼(Dimocarpus longan)中miR408已被证实参与其早期球形胚细胞增殖和分化,能够促进龙眼早期体胚发生[43]。此外,几丁质酶基因还参与香蕉[15]、桃[16]和葡萄[38]等植物的生长发育和果实成熟过程,并具有重要调控作用。本研究表明,AcCHi12靶向最多的为miR172家族; 靶向最多AcCHis靶基因的为miR408家族,推测AcCHis基因的表达主要受到miR172与miR408的调控, 进一步说明AcCHis可能对猕猴桃生长发育和果实后熟软化过程具有重要作用,相关调控方式仍有待进一步研究。
几丁质酶基因在植物各组织器官呈特异性表达[28, 44–46]。对转录组数据的分析表明,GH18和GH19成员AcCHis在不同组织部位(成熟果、幼果、叶、茎、花)中的转录差异明显,GH18成员中,4个基因在各组织部位不表达或表达量较低,7个基因在不同组织中呈差异表达,其中AcCHi10在叶中的转录水平最高;GH19成员中,2个基因在各组织部位高表达;5个基因在不同组织中呈差异表达,均在叶片高表达。在猕猴桃果实贮藏过程中,GH18与GH19成员的表达存在差异,GH18成员中,10个基因在果实贮藏过程不表达或表达量较低。在GH19成员中, 2个基因在果实贮藏过程中不表达或表达量较低, 其余5个基因在果实贮藏过程中呈现差异表达。已有研究表明,CHis参与果实发育与成熟过程,在草莓中,FaChi4在果实中的表达水平伴随着果实的发育逐渐上升,在发育的后期有较高的表达量[37]。香蕉果实用1-甲基-环丙烯处理、乙烯利处理后,几丁质酶表达有差异且与几丁质酶活性变化的曲线一致,说明几丁质酶基因与香蕉采后果实的成熟过程相关[15],推测AcCHis表达变化可能影响果实后熟过程中乙烯、纤维素的生物合成,具体调控机理还有待进一步研究验证。
综上,从猕猴桃基因组中鉴定出18个AcCHis基因,不均匀分布在12条染色体上,其中5个基因发生了片段重复。AcCHis的表达可能受到miR172、miR408的调控,AcCHis家族基因在猕猴桃不同组织部位和果实贮藏软化过程中呈现差异表达,推测AcCHi1、AcCHi4、AcCHi8、AcCHi11、AcCHi13和AcCHi18基因可能参与猕猴桃采后果实贮藏软化过程。
| [1] |
BHATTACHARYA D, NAGPURE A, GUPTA R K. Bacterial chitinases: Properties and potential[J]. Crit Rev Biotechnol, 2007, 27(1): 21-28. DOI:10.1080/07388550601168223 |
| [2] |
GROVER A. Plant chitinases: Genetic diversity and physiological roles[J]. Crit Rev Plant Sci, 2012, 31(1): 57-73. DOI:10.1080/07352689.2011.616043 |
| [3] |
CANTAREL B L, COUTINHO P M, RANCUREL C, et al. The carbohydrate-active enzymes database (CAZy): An expert resource for glycogenomics[J]. Nucl Acids Res, 2009, 37(S1): D233-D238. DOI:10.1093/nar/gkn663 |
| [4] |
HENRISSAT B. A classification of glycosyl hydrolases based on amino acid sequence similarities[J]. Biochem J, 1991, 280(2): 309-316. DOI:10.1042/bj2800309 |
| [5] |
CLETUS J, BALASUBRAMANIAN V, VASHISHT D, et al. Transgenic expression of plant chitinases to enhance disease resistance[J]. Biotechnol Lett, 2013, 35(11): 1719-1732. DOI:10.1007/s10529-013-1269-4 |
| [6] |
PASSARINHO P A, DE VRIES S C. Arabidopsis chitinases: A genomic survey[J]. Arabidops Book, 2002, 1: e0023. DOI:10.1199/tab.0023 |
| [7] |
XU J, XU X Y, TIAN L L, et al. Discovery and identification of candidate genes from the chitinase gene family for Verticillium dahliae resistance in cotton[J]. Sci Rep, 2016, 6: 29022. DOI:10.1038/srep29022 |
| [8] |
PAN X X, HU M Y, WANG Z W, et al. Genome-wide analysis of the rice chitinases gene family and their expression profiles under different stress treatments[J]. Plant Physiol J, 2022, 58(4): 746-756. 潘晓雪, 胡明瑜, 王忠伟, 等. 水稻几丁质酶基因家族的全基因组鉴定及表达分析[J]. 植物生理学报, 2022, 58(4): 746-756. DOI:10.13592/j.cnki.ppj.2021.0401 |
| [9] |
LIU Z Y, YU W F, CAI W L, et al. Genome-wide identification and expression analysis of CTL gene family memebers in Capsicum annuum L.[J]. Chin J Trop Crops, 2021, 42(11): 3101-3110. 刘卓毅, 于文菲, 蔡文丽, 等. 辣椒几丁质酶类基因家族的全基因组鉴定和表达特征分析[J]. 热带作物学报, 2021, 42(11): 3101-3110. DOI:10.3969/j.issn.1000-2561.2021.11.008 |
| [10] |
GONG K L, CHEN S H, JI X C, et al. The research progress of plant chitinases[J]. Mol Plant Breed, 2019, 17(20): 6840-6849. 巩凯玲, 陈双慧, 纪晓晨, 等. 植物几丁质酶的研究进展[J]. 分子植物育种, 2019, 17(20): 6840-6849. DOI:10.13271/j.mpb.017.006840 |
| [11] |
SINGH H R, DEKA M, DAS S. Enhanced resistance to blister blight in transgenic tea [Camellia sinensis (L.) O. Kuntze] by overexpression of class Ⅰ chitinase gene from potato (Solanum tuberosum)[J]. Funct Integr Genom, 2015, 15(4): 461-480. DOI:10.1007/s10142-015-0436-1 |
| [12] |
LIN Q M, WANG X, ZHAO D F, et al. Identification of banana chitinase gene family and analysis of its expression in fusarium wilt infection[J]. J Plant Genet Resources, 2022, 23(1): 272-280. 林秋妹, 王霞, 赵东方, 等. 香蕉几丁质酶基因家族的鉴定及在枯萎病侵染时的表达分析[J]. 植物遗传资源学报, 2022, 23(1): 272-280. DOI:10.13430/j.cnki.jpgr.20210622001 |
| [13] |
JU D. Induced-responses of the Chilo suppressalis to resistant rice cultivars and the expression pattern of chitinase gene OsCHIT15[D]. Shenyang: Shenyang Agricultural University, 2019. 鞠迪. 抗螟水稻诱导二化螟防御反应及几丁质酶基因OsCHIT15的表达模式研究[D]. 沈阳: 沈阳农业大学, 2019. |
| [14] |
ZHU C, ZHANG S T, CHANG X J, et al. Cloning and its expression analysis of chitinase under drought stress in Camellia sinensis[J]. Chin J Trop Crops, 2017, 38(5): 894-902. 朱晨, 张舒婷, 常笑君, 等. 茶树几丁质酶基因的克隆及其在干旱胁迫下的表达分析[J]. 热带作物学报, 2017, 38(5): 894-902. DOI:10.3969/j.issn.1000-2561.2017.05.018 |
| [15] |
LIANG Q F. The chitinase activities and gene expression of banana and the ripening after harvest[D]. Danzhou: South China University of Tropical Agriculture, 2006. 梁秋芬. 香蕉几丁质酶活性及基因表达与采后成熟[D]. 儋州: 华南热带农业大学, 2006. |
| [16] |
XU Z, DAI J Y, LIANG L P, et al. Chitinase-like protein PpCTL1 contributes to maintaining fruit firmness by affecting cellulose biosynthesis during peach development[J]. Foods, 2023, 12(13): 2503. DOI:10.3390/foods12132503 |
| [17] |
HUANG W J, ZHONG C H. Research advances in the postharvest physiology of kiwifruit[J]. Plant Sci J, 2017, 35(4): 622-630. 黄文俊, 钟彩虹. 猕猴桃果实采后生理研究进展[J]. 植物科学学报, 2017, 35(4): 622-630. DOI:10.11913/PSJ.2095-0837.2017.40622 |
| [18] |
CHEN K S, ZHANG S L, LV J L, et al. Variation of abscisic acid, indole-3-acetic acid and ethylene in kiwifruit during fruit ripening[J]. Sci Agric Sin, 1997, 30(2): 54-57. 陈昆松, 张上隆, 吕均良, 等. 脱落酸、吲哚乙酸和乙烯在猕猴桃果实后熟软化进程中的变化[J]. 中国农业科学, 1997, 30(2): 54-57. |
| [19] |
ZHANG M F, HE L, ZHANG M L, et al. Advances in preservation methods for kiwifruit[J]. Food Sci, 2014, 35(11): 343-347. 张美芳, 何玲, 张美丽, 等. 猕猴桃鲜果贮藏保鲜研究进展[J]. 食品科学, 2014, 35(11): 343-347. DOI:10.7506/spkx1002-6630-201411066 |
| [20] |
XIN Y C, WANG D H, HAN S M, et al. Characterization of the chitinase gene family in mulberry (Morus notabilis) and MnChi18 Involved in resistance to botrytis cinerea[J]. Genes (Basel), 2021, 13(1): 98. DOI:10.3390/genes13010098 |
| [21] |
LEI J, BAN X W, LIU R, et al. Genome-wide identification and expression analysis of TCP gene family under abiotic stress in Coix lacryma-jobi L.[J]. Mol Plant Breed, 2023, 1-22. 雷静, 班秀文, 刘荣, 等. 薏苡(Coix lacryma-jobi L.) TCP家族全基因组鉴定及响应非生物胁迫的表达规律[J]. 分子植物育种, 2023, 1-22. |
| [22] |
DUAN Q, HE Z B, WANG G L, et al. Genome-wide identification, evolution and expression analysis of the SBP gene family in castor beans (Ricinus communis L. )[J]. Mol Plant Breed, 2024. 段强, 何智彪, 王桂玲, 等. 蓖麻SBP基因家族全基因组鉴定、进化和表达分析[J/OL]. 分子植物育种, 2024. [2023-05-10]. https://kns.cnki.net/kcms/detail/46.1068.S.20230509.1513.012.html. |
| [23] |
JI X R, YU Y H, NI P Y, et al. Genome-wide identification of small heat-shock protein (HSP20) gene family in grape and expression profile during berry development[J]. BMC Plant Biol, 2019, 19(1): 433. DOI:10.1186/s12870-019-2031-4 |
| [24] |
CAO J, TAN X N. Comprehensive analysis of the chitinase family genes in tomato (Lycopersicon esculentum)[J]. Plants (Basel), 2019, 8(3): 52. DOI:10.3390/plants8030052 |
| [25] |
HAXIM Y, KAHAR G, ZHANG X C, et al. Genome-wide characterization of the chitinase gene family in wild apple (Malus sieversii) and domesticated apple (Malus domestica) reveals its role in resistance to Valsa mali[J]. Front Plant Sci, 2022, 13: 1007936. DOI:10.3389/fpls.2022.1007936 |
| [26] |
CANNON S B, MITRA A, BAUMGARTEN A, et al. The roles of segmental and tandem gene duplication in the evolution of large gene families in Arabidopsis thaliana[J]. BMC Plant Biol, 2004, 4: 10. DOI:10.1186/1471-2229-4-10 |
| [27] |
XU W, LIU J F, ZHANG G, et al. Genome-wide identification and expression analysis under Fusarium graminearum stress of chitinase gene family in Triticum aestivum L.[J]. J Henan Agric Sci, 2019, 48(11): 7-17. 徐武, 刘建丰, 张戈, 等. 小麦几丁质酶基因家族的全基因组鉴定及禾谷镰刀菌胁迫下的表达分析[J]. 河南农业科学, 2019, 48(11): 7-17. DOI:10.15933/j.cnki.1004-3268.2019.11.002 |
| [28] |
HU Y P, GUO Y J, JI Q H, et al. Analysis of structure, classification and evolution of chitinase gene family in citrus[J]. S China Fruits, 2022, 51(6): 16-21. 胡亚平, 郭雁君, 吉前华, 等. 柑桔几丁质酶基因家族的结构、分类与进化分析[J]. 中国南方果树, 2022, 51(6): 16-21. DOI:10.13938/j.issn.1007-1431.20210291 |
| [29] |
LI H M, LI L L, CHEN Y M, et al. Bioinformatics analysis of chitinase gene family in Siraitia grosvenorii[J]. Chin J Bioinform, 2019, 17(3): 167-174. 李惠敏, 李璐璐, 陈玉梅, 等. 罗汉果几丁质酶基因家族的生物信息学分析[J]. 生物信息学, 2019, 17(3): 167-174. DOI:10.12113/j.issn.1672-5565.201903006 |
| [30] |
SONG Z P, PAN F L, LOU X P, et al. Genome-wide identification and characterization of Hsp70 gene family in Nicotiana tabacum[J]. Mol Biol Rep, 2019, 46(2): 1941-1954. DOI:10.1007/s11033-019-04644-7 |
| [31] |
PAN X F, YE F T, MAO Z J, et al. Genomic identification and molecular evolution of the WRKY family in Nymphaea colorata[J]. Acta Hort Sin, 2022, 49(5): 1121-1135. 潘鑫峰, 叶方婷, 毛志君, 等. 睡莲WRKY家族的全基因组鉴定和分子进化分析[J]. 园艺学报, 2022, 49(5): 1121-1135. DOI:10.16420/j.issn.0513-353x.2021-0092 |
| [32] |
ZHAO W, LIU Y H, LI L, et al. Genome-wide identification and characterization of bHLH transcription factors related to anthocyanin biosynthesis in red walnut (Juglans regia L.)[J]. Front Genet, 2021, 12: 632509. DOI:10.3389/fgene.2021.632509 |
| [33] |
XU J L, ZHANG W J, XIANG F N. Advances in stress inducible promoter and cis-acting elements in higher plants[J]. Plant Physiol J, 2021, 57(4): 759-766. 徐金龙, 张文静, 向凤宁. 植物盐胁迫诱导启动子及其顺式作用元件研究进展[J]. 植物生理学报, 2021, 57(4): 759-766. DOI:10.13592/j.cnki.ppj.2020.0221 |
| [34] |
ZHU J K. Abiotic stress signaling and responses in plants[J]. Cell, 2016, 167(2): 313-324. DOI:10.1016/j.cell.2016.08.029 |
| [35] |
LI Z X, CHEN X B. Research advances on plant inducible promoters and related cis-acting elements[J]. Biotechnol Bull, 2015, 31(10): 8-15. 李濯雪, 陈信波. 植物诱导型启动子及相关顺式作用元件研究进展[J]. 生物技术通报, 2015, 31(10): 8-15. DOI:10.13560/j.cnki.biotech.bull.1985.2015.10.006 |
| [36] |
LIU J H, XU B Y, ZHANG J B, et al. A class Ⅲ acidic chitinase gene was closely correlated with postharvest banana fruit ripening[J]. Acta Bot Boreali-Occid Sin, 2010, 30(10): 2022-2027. 刘菊华, 徐碧玉, 张建斌, 等. 香蕉一个Ⅲ类酸性几丁质酶基因与果实成熟关系的研究(英文)[J]. 西北植物学报, 2010, 30(10): 2022-2027. |
| [37] |
WANG Y, HE S H, PEI Y L, et al. Transcription characterization of FaChi1-FaChi4 and their responses to drought stress, exogenous abscisic acid and Botrytis cinerea in strawberry fruit[J]. J China Agric Univ, 2015, 20(6): 108-116. 王亚, 贺绥欢, 裴越琳, 等. 草莓几丁质酶基因FaChi1-FaChi4的转录特性及其对干旱胁迫、外施脱落酸及灰霉菌的响应[J]. 中国农业大学学报, 2015, 20(6): 108-116. DOI:10.11841/j.issn.1007-4333.2015.06.14 |
| [38] |
ROBINSON S P, JACOBS A K, DRY I B. A class Ⅳ chitinase is highly expressed in grape berries during ripening[J]. Plant Physiol, 1997, 114(3): 771-778. DOI:10.1104/pp.114.3.771 |
| [39] |
CHUNG M Y, NATH U K, VREBALOV J, et al. Ectopic expression of miRNA172 in tomato (Lycopersicon esculentum) reveals novel function in fruit development through regulation of an AP2 transcription factor[J]. BMC Plant Biol, 2020, 20(1): 283. DOI:10.1186/s12870-020-02489-y |
| [40] |
DEBERNARDI J M, GREENWOOD J R, FINNEGAN E J, et al. APETALA 2-like genes AP2L2 and Q specify lemma identity and axillary floral meristem development in wheat[J]. Plant J, 2020, 101(1): 171-187. DOI:10.1111/tpj.14528 |
| [41] |
LAKHOTIA H, JOSHI G, BHARDWAJ A R, et al. Identification and characterization of miRNAome in root, stem, leaf and tuber developmental stages of potato (Solanum tuberosum L.) by high-throughput sequencing[J]. BMC Plant Biol, 2014, 14: 6. DOI:10.1186/1471-2229-14-6 |
| [42] |
HU Y J, JI J Y, CHENG H, et al. The miR408a-BBP-LAC3/CSD1 module regulates anthocyanin biosynthesis mediated by crosstalk between copper homeostasis and ROS homeostasis during light induction in Malus plants[J]. J Adv Res, 2023, 51: 27-44. DOI:10.1016/j.jare.2022.11.005 |
| [43] |
XU X P. The whole-genome wide identification of miRNA and functional study of miR408 during early somatic embryogenesis in Dimocarpus longan Lour [D]. Fuzhou: Fujian Agriculture and Forestry University, 2022. 徐小萍. 龙眼体胚发生早期miRNA全基因组鉴定与miR408功能研究[D]. 福州: 福建农林大学, 2022. |
| [44] |
YU W F. Functional study of chitinase-like gene PaCTL1 in petunia[D]. Fuzhou: Fujian Agriculture and Forestry University, 2022. 于文菲. 矮牵牛几丁质酶类基因PaCTL1的功能研究[D]. 福州: 福建农林大学, 2022. |
| [45] |
YUAN X Y, XU S P, SONG C X, et al. Bioinformatics and expression analysis of a chitinase gene from Phalaenopsis spp.[J]. Acta Bot Boreali-Occid Sin, 2016, 36(2): 241-248. 袁秀云, 许申平, 宋彩霞, 等. 蝴蝶兰几丁质酶基因的克隆及表达特性分析[J]. 西北植物学报, 2016, 36(2): 241-248. DOI:10.7606/j.issn.1000-4025.2016.02.0241 |
| [46] |
ZHOU J, HUANG J. Cloning and functional identification of chitinase gene SlChi in Salix[J]. Mol Plant Breed, 2018, 16(24): 8013-8021. 周洁, 黄婧. 柳树几丁质酶基因SlChi的克隆和功能验证[J]. 分子植物育种, 2018, 16(24): 8013-8021. DOI:10.13271/j.mpb.016.008013 |
 2025, Vol. 33
2025, Vol. 33


