2. 中国农业大学生物学院, 北京 100193
2. College of Biological Sciences, China Agricultural University, Beijing 100193, China
硅(Si)作为土壤中较为丰富的非金属元素,在pH < 9.0的土壤中主要以单硅酸[Si(OH)4]的形式被植物的根系吸收[1–2]。虽然Si一直未被列为植物的必须元素,但其在植物生长发育过程中发挥的作用引起了科研工作者的关注。已有研究表明施加Si肥能够促进植物的生长和增强光合作用,从而提高作物的产量和品质[3–4]。此外,Si还能够提高植物对生物和非生物胁迫的耐受能力,已有文献报道Si能提高植物对病原体(如真菌)、盐、干旱、高温与低温、重金属等胁迫的抗性[5–10]。
Si在植物体中的含量约占植物干重的0.1%~10%[11],基于不同植物的Si积累差异最初提出了3个Si吸收的生理模型:外排、被动吸收和主动吸收[12]。Si如何穿越根系中的特化组织并转移到地上部一直是个有趣的问题,直到在高等植物水稻中鉴定到硅转运蛋白Lsi1和Lsi2[13–15],才逐渐解开植物Si转运机制和Si积累差异的原因。本文主要综述了近年来高等植物硅转运蛋白的研究成果和植物对Si的吸收转运机制,希望对后续植物硅转运蛋白的研究提供参考。
1 硅与植物Si作为一种有益元素,在植物中表现出的正效应已经被广泛证实。根据已有的报道,可将Si对植物的作用概括为植物生长发育、植物激素与相关信号通路、生物胁迫和非生物胁迫4个主要的方面[1, 16–23]。Si在植物生长发育过程中的作用:(1) 改善各种养分的吸收,提高N、P、K等元素的吸收, 抑制Cl–的吸收;(2) 提高植物的光合作用;(3) 提高作物的产量与品质。Si在植物激素与相关信号通路中的作用,如SA信号通路、JA信号通路、ET信号通路、ROS信号通路、NO信号通路。Si在生物胁迫中的作用:(1) 抗真菌造成的病害,如雀斑病、锈病、白粉病等;(2) 抗虫害,如蛀虫、大螟、根结线虫等。Si在非生物胁迫中的作用:(1) 减轻有毒元素的毒害,如减轻Al、Cd、Pb等重金属元素以及As和B等非金属元素的毒害;(2) 提高植物的抗盐胁迫能力;(3) 提高植物的干旱胁迫能力;(4) 减轻UV辐射的伤害;(5)提高植物应对温度胁迫的能力。
不同物种间Si含量存在差异,Si含量占植物干重的0.1%~10%。同种植物不同器官中Si含量也存在明显的差异,通常地上部 > 根系[24]。随着研究的深入,转运蛋白在Si的吸收转运过程中发挥重要作用,硅转运蛋白的分布、数量和功能影响植物Si的积累[25]。
2 植物硅转运蛋白最早在硅藻中发现具有Si吸收转运功能的蛋白SIT[26],这类蛋白具有10个跨膜结构域,有2个保守的Gly-Arg-Gln模体分布在跨膜结构域2和3、7和8之间[27]。然而后续的研究表明高等植物存在与硅藻不同的硅吸收转运系统,水稻(Oryza sativa)中鉴定出的硅转运蛋白OsLsi1和OsLsi2为后续的研究提供了参考,已经在多种植物中鉴定出硅转运蛋白[25]。根据硅转运蛋白对Si的转运功能主要分为2类,一类是硅内流转运蛋白,另一类是硅外流转运蛋白。下面将从硅转运蛋白的分子特征、分布特征和表达调控机制3个方面进行综述。
2.1 硅转运蛋白的分子特征 2.1.1 硅内流转运蛋白的分子特征硅内流转运蛋白均属于NOD26膜内在蛋白(nodulin-26-like intrinsic proteins,NIPs)家族的成员,这是一类由5个环(LoopA~LoopE)连接的6次跨膜蛋白,在Loop B和LoopE上分别存在1个Asn-Pro-Ala(NPA)模体,在2个NPA模体之间存在数量恒定的108个氨基酸[28]。
目前在水稻(OsLsi1、OsLsi6)[13, 29]、大麦(Hordeum vulgare, HvLsi1和HvLsi6)[30–31]、玉米(Zea mays, ZmLsi1和ZmLsi6)[32]、小麦(Triticum aestivum, TaLsi1)[33]、黑麦草(Lolium perenne, LpLsi1)[34]、毛竹(Phyllostachys edulis, PhLsi1、PeLsi1-1、PeLsi1-2、PeLsi6-1和PeLsi6-2)[35–36]、南瓜(Cucurbita moschata, CmLsi1)[37]、黄瓜(Cucumis sativus, CsLsi1)[38]、番茄(Lycopersicon esculentum, SILsi1)[39]、烟草(Nicotiana tabacum, NtLsi1)[40]、苹果(Malus domestica, MdLsi1)[41]、土豆(Solanum tuberosum, StLsi1)[42]、草莓(Fragaria× ananassa, FaLsi1)[43]、大豆(Glycine max, GmNIP2-1、GmNIP2-2)[44]、葡萄(Vitis vinifera, VvNIP2-1)[45]、甘蔗(Saccharum officinarum, SbLsi6)[46]、穇(Eleusine coracana, EcLsi1、EcLsi6)[47]中鉴定出硅内流转运蛋白,这些硅内流转运蛋白主要分为2类:Lsi1和Lsi6, 均为同源蛋白,依据ar/R选择性过滤器是由Gly、Ser、Gly、Arg (G、S、G、R)组成被归为NIPIII亚族[48]。其中Lsi1基因的CDS序列长度为849~894 bp, 编码283~298个氨基酸,Lsi6基因编码295~300个氨基酸。Saitoh等[49]在2021年成功解析了水稻OsLsi1的三维结构,OsLsi1与水通道蛋白一样为四聚体结构,但具有不同的跨膜螺旋方向,并且由5个特定的氨基酸(Thr65、Gly88、Ser207、Gly216、Arg222)构成的选择性过滤器使得Lsi1对硅酸具有较高的亲和性。同年,Vanden等[50]也通过X-ray晶体衍射技术解析了OsLsi1的晶体结构,并通过分子模拟技术模拟硅酸在跨膜扩散前与选择性过滤器相互作用的过程,结果与Saitoh等[49]的具有很高的一致性, 表明NIPIII家族独特的选择性过滤器三维空间结构使硅酸能够顺利通过。
然而,并非所有高等植物的硅内流转运蛋白均属于NIPIII亚族成员。蕨类植物木贼(Equisetum hyemale)的硅内流转运蛋白(EaNIP3;1、EaNIP3;3、EaNIP3;4)属于NIPII成员,选择性过滤器由Ser、Thr、Ala、Arg组成。此前还未曾报道过植物NIPII家族成员具有运输硅的能力,对其选择性过滤器的氨基酸组成分析表明最后1个氨基酸Arg在几乎所有植物的水通道蛋白中高度保守[51]。
2.1.2 硅外流转运蛋白的分子特征硅外流转运蛋白均具有Citrate transporter-like结构域,是一类需要能量驱动的转运蛋白[14],与硅内流转运蛋白没有任何相似性,属于未知的离子通道转运蛋白家族。硅外流转运蛋白也存在2类:Lsi2和Lsi3,目前已经在水稻(OsLsi2、OsLsi3)[14, 52–53]、大麦(HvLsi2)[31, 54]、玉米(ZmLsi2)[54]、南瓜(CmLsi2s)[55]、黄瓜(CmLsi2)[56]、土豆(StLsi2)[42]、草莓(FaLsi2)[43]、木贼(EaLsi2s)[57]、番茄(SILsi2s)[39]、穇(EcLsi2、EcLsi3)[47]、毛竹(PeLsi2、PeLsi3-1、PeLsi3-2)[36]中鉴定出硅外流转运蛋白,均为跨膜蛋白,具有8~12个跨膜结构域,由472~547个氨基酸组成。
Lsi2的跨膜结构域在植物中也高度保守,尤其是在TM6和TM7之间最大的细胞内环。在单子叶植物的TM6-TM7环相较于双子叶植物较短和较疏水,这种差异是否会影响Lsi2对Si的转运能力还有待验证[58]。目前对Lsi2和Lsi3的报道也仅仅是通过生物信息学的方法预测了蛋白的信息,蛋白的晶体结构还未曾报道,还需要研究者深入研究其结构特征。
2.2 硅转运蛋白的分布特征Lsi1 单子叶植物水稻、大麦、黑麦草中Lsi1基因只在根系中表达[13, 27, 31],而玉米、毛竹和双子叶植物南瓜、黄瓜、烟草中虽然在地上部也检测到Lsi1的表达,但根系中的表达水平要高于地上部[32, 36–38, 40]。OsLsi1在根的基部区域(距根尖 > 10 mm)的表达量要高于根尖区,HvLsi1在根的基部区域(距根尖 > 15 mm)的表达量也要高于根尖区域, 证实了Lsi1主要在根的成熟区发挥功能[30, 59]。借助免疫染色技术和GFP荧光技术,水稻Lsi1分布在根的外皮层和内皮层的远轴端;大麦和玉米Lsi1分布在根的外表皮和皮层细胞中远轴端[13, 30, 32];而黄瓜和番茄的Lsi1在根外表皮和皮层细胞中无极性分布,在黄瓜内表皮中Lsi1也分布在远轴端[38–39],这种Lsi1在根系中的分布差异可能与植物硅积累能力相关。从南瓜‘Shintosa’和‘Super-unryu’品种中均克隆出Lsi1基因,但2个Lsi1基因编码的蛋白质在第75位和242位的氨基酸存在差异,第242位的氨基酸由脯氨酸变成缬氨酸会使蛋白定位在内质网上而不是细胞膜上,从而使得Lsi1不能正常发挥功能,因此推断不同植物的Lsi1主要在根系中发挥硅内流的功能,其作为植物中最先接触到土壤中Si(OH)4的硅内流转运蛋白,在植物根系对硅的吸收中发挥重要的功能。
Lsi2 玉米、大麦、毛竹、水稻中Lsi2主要在根系中表达[14, 31, 36, 54],而黄瓜和南瓜的Lsi2基因则在根和茎中表达[55–56],大麦的Lsi2基因在生殖期的节点处也有表达[31]。与OsLsi1相似,OsLsi2也在根的成熟区有较高水平的表达[60]。水稻Lsi2定位在根的外表皮和内表皮细胞的近轴端[14],与OsLsi1的分布不同,而大麦、玉米、黄瓜和番茄的Lsi2均无极性分布的定位在内皮层细胞[36, 39, 56],推断Lsi2主要在根系中发挥硅外流的功能,其能够协助皮层细胞中的Si(OH)4穿越凯氏带,负责Si(OH)4在木质部的装载。
Lsi3 目前仅见水稻和毛竹中有Lsi3的报道, 其中水稻Lsi3的研究较为深入。在营养生长阶段, OsLsi3主要在根系中表达;在生殖阶段OsLsi3在Node I表达量较高;在根系中OsLsi3主要在根的成熟区域表达量较高[52–53]。Node I处的定位结果表明,OsLsi3分布在伸长维管束鞘和弥散维管束的薄壁细胞之间[52]。水稻根的组织和细胞定位结果表明, OsLsi3主要分布在中柱鞘中,不具有OsLsi2相似的极性分布特点,表明OsLsi3在中柱鞘中发挥对硅酸的外排作用[53]。我们在毛竹中也鉴定出与OsLsi3同源的蛋白PeLsi3-1和PeLsi3-2,其编码基因在根系和茎中均有表达[36]。而水稻根系中OsLsi3能使Si(OH)4高效在木质部装载,缺失该蛋白会导致水稻硅含量约下降30%[53],足以证明Lsi3在根系硅的转运过程中发挥不可忽视的作用。
Lsi6 目前仅对水稻、玉米、大麦、毛竹Lsi6基因的功能进行了研究。水稻OsLsi6、ZmLsi6、HvLsi6、PeLsi6-1和PeLsi6-2在根、茎、叶中均有表达,在生长阶段OsLsi6和HvLsi6主要在Node I有较高的表达[29, 31–32, 36]。OsLsi6和HvLsi6均在根尖区域有较高水平的表达,与Lsi1在根成熟区的表达情况相反[29, 31]。HvLsi6分布在根系中外皮层和内皮层细胞的远轴端,而OsLsi6和ZmLsi6在根系中均无极性分布,但在地上部的叶片和叶鞘中Lsi6均分布在木质部的薄壁细胞中[29, 31]。Lsi1和Lsi6虽然同为硅内流转运蛋白,但它们在根系中不同区域的表达情况不同,这种差异表达特点使得硅转运蛋白对Si的吸收分工更加明确和高效。敲除OsLsi6导致水稻圆锥花序硅的含量明显减少,证明Lsi6主要在地上部负责Si的卸载和分配[29]。
2.3 硅转运蛋白基因的表达调控机制近年来大多数的研究致力于植物全基因组层面鉴定硅转运蛋白并对相应蛋白的功能进行验证, 仅少数研究涉及硅转运蛋白基因的表达调控机制, 我们按照硅转运蛋白的类别对已有研究成果进行归纳。
Lsi1 外源Si对不同植物Lsi1基因的调控方式主要有3种:水稻、毛竹和黑麦草Lsi1基因的表达水平受到Si的下调[13, 34, 36];玉米、大麦和番茄Lsi1基因的表达水平并不受Si的调控[30, 32, 39];CsLsi1基因受到Si的上调[38]。抑制剂K252a和冈田酸并不能影响OsLsi1的转运能力,说明其不受磷酸化的调控,HgCl处理15 min能够显著的影响Lsi1的转运活性[13, 61]。运用氨基酸突变技术将烟草Lsi1第125位的脯氨酸突变成苯丙氨酸后,Si的转运活性有显著提升。我们通过单颗粒示踪技术研究了毛竹PeLsi1-1在质膜上的运动情况,K2SiO3的处理能够降低蛋白在质膜上的运动能力[36]。此外,对Lsi1基因表达的时间变化分析表明,OsLsi1和CSiT1、CSiT2的表达均具有很明显的生物节律,根系OsLsi1的表达水平在抽穗期表达量达到峰值,这与水稻硅高效吸收的阶段一致[59, 62]。研究表明ABA能够下调OsLsi1的表达,在对其启动子区域分析发现存在ABA响应元件,但是ABA如何调控基因的表达还不清楚[59]。通过启动子序列缺失分析的方法确定在OsLsi1基因上游-327~-292 bp区域能够响应Si,还发现在基因上游-1 082~-886 bp区域存在未知功能元件能抑制基因在距离根尖3 mm处的表达[63]。研究表明HMG1蛋白能够与OsLsi1启动子结合,正向调控该基因的表达使OsLsi1蛋白与ATP合成酶的亚基互作进一步调控SHMT来提高植物的抗寒能力,同时OsLsi1还通过下调14-3-3f的表达进一步调控相应途径提高植物的抗寒性[64]。
Lsi2 Lsi2是阴离子转运蛋白,其活性很大程度受到质子梯度的影响,使用DNP、CCCP、FCCP等解偶联剂和低温处理,Lsi2的活性均受到抑制[14]。除了番茄Lsi2s蛋白不具有功能并且表达不受外源硅的调控,其他物种Lsi2基因均受到外源硅的下调。OsLsi2同样受到ABA的下调,并且根系中的表达量在抽穗期达到峰值,与OsLsi1一同在根系中完成该时期硅的高效吸收[60]。对OsLsi2和OsLsi1的启动子区域进行分析发现有许多相同的顺式元件,2个基因是否共享相同的调控方式仍需要进一步去验证[63]。Lsi2起初作为硅外流转运蛋白从水稻中被鉴定出来,后续的研究表明OsLsi2还能将亚砷酸盐从外皮层转运中柱,可能亚砷酸盐和硅酸共享相似的转运途径[65]。最新的研究表明,一种R2R3型MYB转录因子OsARM1能够结合在OsLsi1和OsLsi2的AC-I元件,进而调节根系对AS的吸收,至于OsARM1能否调节根系对Si的吸收还有待验证[66]。
Lsi3 目前仅在高硅积累型植物水稻和毛竹中有Lsi3的研究。作为Lsi2的同源蛋白Lsi3对硅的外流能力同样与pH有关,低pH下OsLsi3的转运活力显著增强[53]。水稻和毛竹的Lsi3均受到Si的下调[36, 52],OsLsi3的启动子区域与OsLsi2没有任何相似性[53],说明Lsi3的调控机制与Lsi2不同。
Lsi6 目前仅在禾本科植物中有Lsi6的相关报道,OsLsi6、HvLsi6、ZmLsi6并不受Si的调控[29, 31–32], 而PeLsi6-1和PeLsi6-2受到Si的上调[36]。Lsi6是否还受到其他因素的调控,作为Lsi1的同源蛋白是否与Lsi1的调控方式存在相似之处,还有待研究。
3 硅转运蛋白介导的植物Si吸收转运系统Takahashi等[12]最早提出了植物对Si吸收的3个模型:主动吸收、被动吸收和外排,但当时并未发现硅转运蛋白在硅积累中的作用。2015年Ma等[25]描绘出禾本科植物水稻、大麦和玉米根系和地上部的Si吸收转运模式图。Kaur等[67]将双子叶植物和蕨类植物硅转运蛋白的研究进行了归纳,绘制了黄瓜根系的硅吸收转运模式图,综述了不同类型植物根系的Si吸收转运系统,但未提及地上部Si的吸收转运过程。Mandlik等[68]在2020年同样综述了水稻的硅吸收转运系统,并详细介绍了Si在植物不同部位的沉积,然而却没有将Si的沉积和Si的吸收转运过程联系起来。同年,Guar等[69]综述了Si参与植物的抗逆性和硅转运蛋白的研究结果,并未过多涉及植物Si的吸收转运系统。2021年,Mitani等[70]根据植物硅转运蛋白的研究结果,将根系中硅转运蛋白的分布和Takahashi等[12]提出的3个吸收模型相互关联,很好解释了高硅积累型植物、中等硅积累型植物和低硅积累型植物根系对Si的吸收差异。下文对已有的综述内容和最新的研究结果进行分析、整理归纳,并将植物地下部和地上部的Si吸收转运系统进行概述,希望能够完善植物硅转运蛋白参与的Si吸收机理模型。
3.1 根系Si吸收转运系统研究表明,硅转运蛋白Lsi1和Lsi2共同参与根系对土壤中Si的吸收转运过程,其中Lsi1具有双向转运功能,Lsi2受到能量的调控只具有单向转运的功能。然而,一些学者认为Lsi2作为“黑匣子”对根系中单硅酸的转运发挥关键的功能[58]。拟南芥作为典型的非硅积累型植物,其水通道蛋白家族中并未出现与Lsi1同源的蛋白,但却具有Lsi2同源的蛋白,将小麦Lsi1基因在拟南芥异源表达,硅积累能力有了明显的提升[33];将毛竹PeLsi1-1、PeLsi1-2、PeLsi2基因转化拟南芥,其中转PeLsi1-1和PeLsi1-2基因的拟南芥具有明显的硅积累能力,而转PeLsi2基因的拟南芥的硅积累能力并未有明显变化[36];水稻缺少OsLsi1和OsLsi2任何一个硅转运蛋白,硅的积累能力均受到严重的影响,进而影响水稻的正常发育[13–14]。所以Lsi1和Lsi2在硅积累型植物的根系中必不可少,两者协同完成根系对土壤中硅素的吸收。此外,凯氏带结构也在Si(OH)4的吸收转运中起到一定的作用,已有研究表明,凯氏带的结构缺失会导致内皮层上的硅转运蛋白的数量减少, 从而导致Si等元素的吸收受到严重的抑制[71]。
综合分析已发表的文献,我们对硅积累型植物根系对Si(OH)4的吸收过程总结如下:(1) 土壤中的Si(OH)4可以与水分一样自由的穿越根系的表皮; (2) 进入表皮的Si(OH)4可以通过质外体途径沿皮层细胞横向运输到内皮层,或者通过硅转运蛋白Lsi1介导的共质体途径从表皮运输到内皮层;(3) 内皮层具有凯氏带结构,Si(OH)4不能通过质外体途径穿越凯氏带,Si(OH)4只能通过跨细胞质膜的方式进入到内皮层细胞并被内皮层上分布的Lsi2外排出细胞顺利通过内皮层;(4) 从内皮层释放的Si(OH)4便可通过质外体途径运输到中柱鞘,进一步装载到木质部,并通过蒸腾作用转运到地上部。
鉴于单子叶植物和双子叶植物根系结构存在明显差异,融合现有文献资料和我们对毛竹的研究结果,构建了禾本科植物(图 1: A左侧)和双子叶植物(图 1: B左侧)根系Si吸收转运系统模型。图中3种模型代表 3类Si积累型植物,其中Lsi1和Lsi2在根系不同组织的分布差异显著影响植物根对Si(OH)4的吸收与转运能力。值得注意的是,2022年Huang等[53]报道OsLsi3在木质部能高效装载Si(OH)4, 我们的研究表明,Lsi3在毛竹根系中有较高水平的表达,这导致水稻和毛竹根系的硅吸收能力要强于目前已知的硅积累型植物。
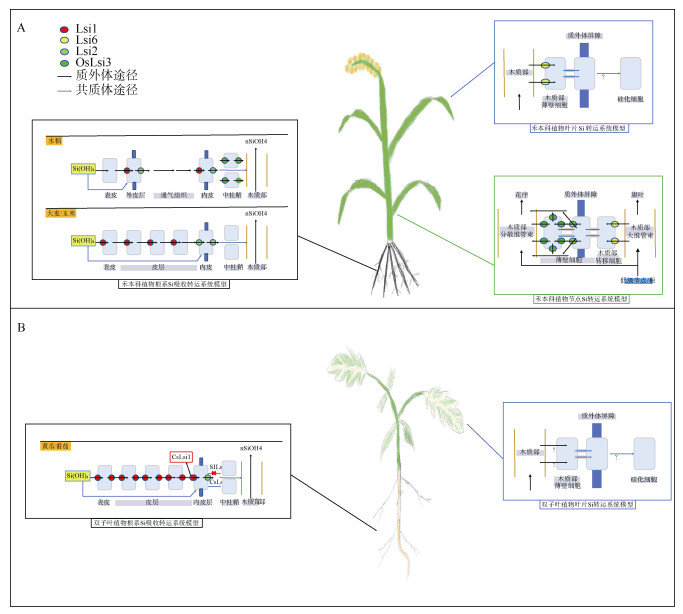
|
图 1 硅转运蛋白介导的植物硅吸收转运系统工作模型。A: 禾本科植物; B: 双子叶植物。 Fig. 1 Working model of Si transporter-mediated Si transport systems in plants. A: Gramineae; B: Dicotyledon. |
目前仅对禾本科植物地上部硅转运蛋白进行了较深入研究,我们总结出禾本科植物地上部硅转运系统工作模型(图 1: A右侧)。木质部装载的Si(OH)4会在禾本科植物的节处进行分配,大部分Si(OH)4会在硅转运蛋白的协助下或通过质外体途径优先转运到花序中。硅转运蛋白参与Si(OH)4从木质部大维管束到花序的运输过程:(1) 木质部大维管束的Si(OH)4被木质部转移细胞的Lsi6卸载并进入木质部转移细胞;(2) 通过薄壁细胞间的胞间连丝运输到木质部分散维管束;(3) Si(OH)4在木质部分散维管束重新装载并向上转运到花序。小部分Si(OH)4可以直接通过木质部大维管束转运到旗叶并在叶片中沉积:(1) Si(OH)4通过Lsi6的卸载进入薄壁细胞;(2) 薄壁细胞中Si(OH)4通过胞间连丝在细胞中运输并通过未知的转运途径转运到硅化细胞中。除水稻和大麦外,其他已经鉴定出硅转运蛋白的禾本科植物在地上部只检测到Lsi6,缺乏单向运输的Lsi2和高效Si(OH)4装载的转运蛋白Lsi3,这可能是影响地上部硅积累能力的重要原因。
而双子叶植物地上部硅转运蛋白的研究比较少,仅见硅转运蛋白基因Lsi1和Lsi2在地上部表达的报道,但这2个转运蛋白是否参与Si(OH)4在地上部的转运过程还不清楚。我们同样构建了双子叶植物叶片Si转运模型(图 1: B右侧),具体发挥转运功能的蛋白还未知,需要科研工作者来填补这一空白。
通过硅转运蛋白介导对Si(OH)4的转运, Si(OH)4被分配到地上部不同的组织和器官中。研究表明, 禾本科植物和木贼均具有硅化结构,其细胞壁上均具有一种混合交联的葡聚糖,推测细胞壁可能作为SiO2沉积的模板[72]。富含脯氨酸的蛋白(如SBP117)能够诱导硅酸的沉淀[73],在植硅体中释放的赖氨酸和酸性氨基酸加强了SiO2的沉积[74]。硅酸可以在细胞间隙中沉积,也可在特定的细胞中完成生物硅化作用,在叶片中硅的沉积可以形成哑铃型和泡状等形式的植硅体。此外,在植物体中还存在有机硅, 有机硅可以与细胞壁上的半纤维素交联,这样的有机结构提高了细胞壁的机械性能并且有利于细胞膜的稳定[75–76]。
4 展望Si作为土壤中含量丰富的非金属元素,其是否作为植物的必需元素一直饱受争议。然而,Si在植物生长发育及应答各种生物及非生物胁迫中发挥着重要的作用,尤其是对禾本科植物的产量至关重要[3–10, 16–23]。Si的吸收转运过程与硅转运蛋白的功能密切相关,各种前沿生物技术的快速发展及应用,可以更好地帮助科研工作者鉴定并阐明硅转运蛋白的功能。尽管如此,目前对植物硅转运蛋白的研究还存在许多困惑,需要进行更多的探索。提出该研究领域中一些值得深入思考的问题供研究者参考:(1) 目前仅是简单的分析了Si对转运蛋白基因的调控方式,是否存在多种因素共同参与调控硅转运蛋白基因的表达?(2) 单子叶植物和双子叶植物中的硅吸收转运蛋白均属于NIPIII亚族成员,但蕨类植物中NIPII亚族的部分成员也具有硅吸收能力,是否在植物体中还存在其他具有硅吸收或外排功能的蛋白?(3) 目前仅研究了水稻中地上部的硅转运系统,其他硅积累型植物地上部的硅转运系统又是怎样的?(4) 在不同硅积累型植物根系中硅转运蛋白的分布具有明显差异,这样的分布特点是由什么因素决定的?(5) 仍有硅积累型植物还未获得全基因组数据,硅转运蛋白的数量和功能还需要进一步研究。
| [1] |
TAYADE R, GHIMIRE A, KHAN W, et al. Silicon as a smart fertilizer for sustainability and crop improvement[J]. Biomolecules, 2022, 12(8): 1027. DOI:10.3390/biom12081027 |
| [2] |
EPSTEIN E. The anomaly of silicon in plant biology[J]. Proc Natl Acad Sci USA, 1994, 91(1): 11-17. DOI:10.1073/pnas.91.1.11 |
| [3] |
XU C X, LIU Z P, LIU Y L. The physiological function of silicon in plants[J]. Plant Physiol Commun, 2004, 40(6): 753-757. 徐呈祥, 刘兆普, 刘友良. 硅在植物中的生理功能[J]. 植物生理学通讯, 2004, 40(6): 753-757. DOI:10.13592/j.cnki.ppj.2004.06.036 |
| [4] |
GAO D, CAI K Z, CHEN J N, et al. Silicon enhances photochemical efficiency and adjusts mineral nutrient absorption in Magnaporthe oryzae infected rice plants[J]. Acta Physiol Plant, 2011, 33(3): 675-682. DOI:10.1007/s11738-010-0588-5 |
| [5] |
CAI K Z, GAO D, CHEN J N, et al. Probing the mechanisms of silicon-mediated pathogen resistance[J]. Plant Signal Behav, 2009, 4(1): 1-3. DOI:10.4161/psb.4.1.7280 |
| [6] |
SHI Y, ZHANG Y, YAO H J, et al. Silicon improves seed germination and alleviates oxidative stress of bud seedlings in tomato under water deficit stress[J]. Plant Physiol Biochem, 2014, 78: 27-36. DOI:10.1016/j.plaphy.2014.02.009 |
| [7] |
ZHU Y X, XU X B, HU Y H, et al. Silicon improves salt tolerance by increasing root water uptake in Cucumis sativus L.[J]. Plant Cell Rep, 2015, 34(9): 1629-1646. DOI:10.1007/s00299-015-1814-9 |
| [8] |
AZEEM S, LI Z, ZHENG H P, et al. Quantitative proteomics study on Lsi1 in regulation of rice (Oryza sativa L.) cold resistance[J]. Plant Growth Regul, 2016, 78(3): 307-323. DOI:10.1007/s10725-015-0094-2 |
| [9] |
SOUNDARARAJAN P, SIVANESAN I, JANA S, et al. Influence of silicon supplementation on the growth and tolerance to high temperature in Salvia splendens[J]. Hort Environ Biotechnol, 2014, 55(4): 271-279. |
| [10] |
KIM Y H, KHAN A L, KIM D H, et al. Silicon mitigates heavy metal stress by regulating p-type heavy metal ATPases, Oryza sativa low silicon genes, and endogenous phytohormones[J]. BMC Plant Biol, 2014, 14(1): 13. DOI:10.1186/1471-2229-14-13 |
| [11] |
MA J F, TAKAHASHI E. Soil, Fertilizer and Plant Silicon Research in Japan[M]. Amsterdam: Elsevier Science, 2002, 12: 257-274.
|
| [12] |
TAKAHASHI E, MA J F, MIYAKE Y. The possibility of silicon as an essential element for higher plants[J]. Comments Agric Food Chem, 1990, 2(2): 99-122. |
| [13] |
MA J F, TAMAI K, YAMAJI N, et al. A silicon transporter in rice[J]. Nature, 2006, 440(7084): 688-691. DOI:10.1038/nature04590 |
| [14] |
MA J F, YAMAJI N, MITANI N, et al. An efflux transporter of silicon in rice[J]. Nature, 2007, 448(7150): 209-212. DOI:10.1038/nature05964 |
| [15] |
MA J F, YAMAJI N. Silicon uptake and accumulation in higher plants[J]. Trends Plant Sci, 2006, 11(8): 392-397. DOI:10.1016/j.tplants.2006.06.007 |
| [16] |
LUYCKX M, HAUSMAN J F, LUTTS S, et al. Silicon and plants: Current knowledge and technological perspectives[J]. Front Plant Sci, 2017, 8: 411. DOI:10.3389/fpls.2017.00411 |
| [17] |
MUKARRAM M, PETRIK P, MUSHTAQ Z, et al. Silicon nanoparticles in higher plants: Uptake, action, stress tolerance, and crosstalk with phytohormones, antioxidants, and other signaling molecules[J]. Environ Pollut, 2022, 310: 119855. DOI:10.1016/j.envpol.2022.119855 |
| [18] |
COSKUN D, DESHMUKH R, SONAH H, et al. The controversies of silicon's role in plant biology[J]. New Phytol, 2019, 221(1): 67-85. DOI:10.1111/nph.15343 |
| [19] |
AHIRE M L, MUNDADA P S, NIKAM T D, et al. Multifaceted roles of silicon in mitigating environmental stresses in plants[J]. Plant Physiol Biochem, 2021, 169: 291-310. DOI:10.1016/j.plaphy.2021.11.010 |
| [20] |
DEBONA D, RODRIGUES F A, DATNOFF L E. Silicon's role in abiotic and biotic plant stresses[J]. Annu Rev Phytopathol, 2017, 55(1): 85-107. DOI:10.1146/annurev-phyto-080516-035312 |
| [21] |
SHANMUGAIAH V, GAUBA A, HARI S K, et al. Effect of silicon micronutrient on plant's cellular signaling cascades in stimulating plant growth by mitigating the environmental stressors[J]. Plant Growth Regul, 2023, 100(2): 391-408. DOI:10.1007/s10725-023-00982-6 |
| [22] |
KHAN I, AWAN S A, RIZWAN M, et al. Silicon: An essential element for plant nutrition and phytohormones signaling mechanism under stressful conditions[J]. Plant Growth Regul, 2023, 100(2): 301-319. DOI:10.1007/s10725-022-00872-3 |
| [23] |
SIDDIQUI M H, MUKHERJEE S, AL-MUNQEDHI B M A, et al. Salicylic acid and silicon impart resilience to lanthanum toxicity in Brassica juncea L. seedlings[J]. Plant Growth Regul, 2023, 100(2): 453-466. DOI:10.1007/s10725-021-00787-5 |
| [24] |
GONG H J, CHEN K M, WANG S M, et al. Advances in silicon nutrition of plants[J]. Acta Bot Boreali-Occid Sin, 2004, 24(12): 2385-2392. 宫海军, 陈坤明, 王锁民, 等. 植物硅营养的研究进展[J]. 西北植物学报, 2004, 24(12): 2385-2392. DOI:10.3321/j.issn:1000-4025.2004.12.034 |
| [25] |
MA J F, YAMAJI N. A cooperative system of silicon transport in plants[J]. Trends Plant Sci, 2015, 20(7): 435-442. DOI:10.1016/j.tplants.2015.04.007 |
| [26] |
HILDEBRAND M, VOLCANI B E, GASSMANN W, et al. A gene family of silicon transporters[J]. Nature, 1997, 385(6618): 688-689. DOI:10.1038/385688b0 |
| [27] |
MARRON A O, ALSTON M J, HEAVENS D, et al. A family of diatom-like silicon transporters in the siliceous loricate choanoflagellates[J]. Proc R Soc B Biol Sci, 2013, 280(1756): 20122543. DOI:10.1098/rspb.2012.2543 |
| [28] |
POMMERRENIG B, DIEHN T A, BIENERT G P. Metalloido-porins: Essentiality of nodulin 26-like intrinsic proteins in metalloid transport[J]. Plant Sci, 2015, 238: 212-227. DOI:10.1016/j.plantsci.2015.06.002 |
| [29] |
YAMAJI N, MITATNI N, MA J F. A transporter regulating silicon distribution in rice shoots[J]. Plant Cell, 2008, 20(5): 1381-1389. DOI:10.1105/tpc.108.059311 |
| [30] |
CHIBA Y, MITANI N, YAMAJI N, et al. HvLsi1 is a silicon influx transporter in barley[J]. Plant J, 2009, 57(5): 810-818. DOI:10.1111/j.1365-313X.2008.03728.x |
| [31] |
YAMAJI N, CHIBA Y, MITANI-UENO N, et al. Functional characterrization of a silicon transporter gene implicated in silicon distribution in barley[J]. Plant Physiol, 2012, 160(3): 1491-1497. DOI:10.1104/pp.112.204578 |
| [32] |
MITANI N, YAMAJI N, MA J F. Identification of maize silicon influx transporters[J]. Plant Cell Physiol, 2009, 50(1): 5-12. DOI:10.1093/pcp/pcn110 |
| [33] |
MONTPETIT J, VIVANCOS J, MITANI-UENO N, et al. Cloning, functional characterization and heterologous expression of TaLsi1, a wheat silicon transporter gene[J]. Plant Mol Biol, 2012, 79(1/2): 35-46. DOI:10.1007/s11103-012-9892-3 |
| [34] |
PONTIGO S, LARAMA G, PARRA-ALMUNA L, et al. Physiological and molecular insights involved in silicon uptake and transport in ryegrass[J]. Plant Physiol Biochem, 2021, 163: 308-316. DOI:10.1016/j.plaphy.2021.04.013 |
| [35] |
MA H, WANG K, WU M D, et al. ORF cloning and bioinformatics analysis of silicon transporter PhLsi1 in Phyllostachys edulis[J]. J Hebei Norm Univ (Nat Sci), 2015, 39(4): 345-351. 马欢, 王凯, 吴妙丹, 等. 毛竹硅转运基因PhLsi1的ORF克隆及生物信息学分析[J]. 河北师范大学学报(自然科学版), 2015, 39(4): 345-351. DOI:10.13763/j.cnki.jhebnu.nse.2015.04.012 |
| [36] |
GENG X, GE B H, LIU Y J, et al. Genome-wide identification and functional analysis of silicon transporter family genes in moso bamboo (Phyllostachys edulis)[J]. Int J Biol Macromol, 2022, 223: 1705-1719. DOI:10.1016/j.ijbiomac.2022.10.099 |
| [37] |
MITANI N, YAMAJI N, AGO Y, et al. Isolation and functional characterization of an influx silicon transporter in two pumpkin cultivars contrasting in silicon accumulation[J]. Plant J, 2011, 66(2): 231-240. DOI:10.1111/j.1365-313X.2011.04483.x |
| [38] |
SUN H, GUO J, DUAN Y K, et al. Isolation and functional characterization of CsLsi1, a silicon transporter gene in Cucumis sativus[J]. Physiol Plant, 2017, 159(2): 201-214. DOI:10.1111/ppl.12515 |
| [39] |
SUN H, DUAN Y K, MITANI-UENO N, et al. Tomato roots have a functional silicon influx transporter but not a functional silicon efflux transporter[J]. Plant Cell Environ, 2020, 43(3): 732-744. DOI:10.1111/pce.13679 |
| [40] |
COSKUN D, DESHMUKH R, SONAH H, et al. Si permeability of a deficient Lsi1 aquaporin in tobacco can be enhanced through a conserved residue substitution[J]. Plant Direct, 2019, 3(8): e00163. DOI:10.1002/pld3.163 |
| [41] |
LIU H L, YANG L L, XIN M M. Cloning and bioinformatic analysis of MdLsi1 gene in apple (Malus domestica Borkh.)[J]. N Hort, 2019(9): 27-33. 刘海莉, 杨蕾蕾, 辛苗苗, 等. 苹果MdLsi1基因的克隆及生物信息学分析[J]. 北方园艺, 2019(9): 27-33. DOI:10.11937/bfyy.20183232 |
| [42] |
VULAVALA V K R, ELBAUM R, YERMIYAHU U, et al. Silicon fertilization of potato: Expression of putative transporters and tuber skin quality[J]. Planta, 2016, 243(1): 217-229. DOI:10.1007/s00425-015-2401-6 |
| [43] |
OUELLETTE S, GOYETTE M H, LABBÉ C, et al. Silicon transporters and effects of silicon amendments in strawberry under high tunnel and field conditions[J]. Front Plant Sci, 2017, 8: 949. DOI:10.3389/fpls.2017.00949 |
| [44] |
DESHMUKH R K, VIVANCOS J, GUÉRIN V, et al. Identification and functional characterization of silicon transporters in soybean using comparative genomics of major intrinsic proteins in Arabidopsis and rice[J]. Plant Mol Biol, 2013, 83(4/5): 303-315. DOI:10.1007/s11103-013-0087-3 |
| [45] |
NORONHA H, SILVA A, MITANI-UENO N, et al. The grapevine NIP2;1 aquaporin is a silicon channel[J]. J Exp Bot, 2020, 71(21): 6789-6798. DOI:10.1093/jxb/eraa294 |
| [46] |
CEN G L, SUN T T, CHEN Y L, et al. Characterization of silicon transporter gene family in Saccharum and functional analysis of the ShLsi6 gene in biotic stress[J]. Gene, 2022, 822: 146331. DOI:10.1016/j.gene.2022.146331 |
| [47] |
MUNDADA P S, AHIRE M L, UMDALE S D, et al. Characterization of influx and efflux silicon transporters and understanding their role in the osmotic stress tolerance in finger millet [Eleusine coracana (L.) Gaertn. ][J]. Plant Physiol Biochem, 2021, 162: 677-689. DOI:10.1016/j.plaphy.2021.03.033 |
| [48] |
YAMAJI N, MA J F. Metalloid transporters and their regulation in plants[J]. Plant Physiol, 2021, 187(4): 1929-1939. DOI:10.1093/plphys/kiab326 |
| [49] |
SAITOH Y, MITANI-UENO N, SAITO K, et al. Structural basis for high selectivity of a rice silicon channel Lsi1[J]. Nat Commun, 2021, 12(1): 6236. DOI:10.1038/s41467-021-26535-x |
| [50] |
VAN DEN BERG B, PEDEBOS C, BOLLA J R, et al. Structural basis for silicic acid uptake by higher plants[J]. J Mol Biol, 2021, 433(21): 167226. DOI:10.1016/j.jmb.2021.167226 |
| [51] |
GRÉGOIRE C, RÉMUS-BOREL W, VIVANCOS J, et al. Discovery of a multigene family of aquaporin silicon transporters in the primitive plant Equisetum arvense[J]. Plant J, 2012, 72(2): 320-330. DOI:10.1111/j.1365-313X.2012.05082.x |
| [52] |
YAMAJI N, SAKURAI G, MITANI-UENO N, et al. Orchestration of three transporters and distinct vascular structures in node for intervascular transfer of silicon in rice[J]. Proc Natl Acad Sci USA, 2015, 112(36): 11401-11406. DOI:10.1073/pnas.1508987112 |
| [53] |
HUANG S, YAMAJI N, SAKURAI G, et al. A pericycle-localized silicon transporter for efficient xylem loading in rice[J]. New Phytol, 2022, 234(1): 197-208. DOI:10.1111/nph.17959 |
| [54] |
MITANI N, CHIBA Y, YAMAJI N, et al. Identification and characterization of maize and barley Lsi2-like silicon efflux transporters reveals a distinct silicon uptake system from that in rice[J]. Plant Cell, 2009, 21(7): 2133-2142. DOI:10.1105/tpc.109.067884 |
| [55] |
MITANI-UENO N, YAMAJI N, MA J F. Silicon efflux transporters isolated from two pumpkin cultivars contrasting in Si uptake[J]. Plant Signal Behav, 2011, 6(7): 991-994. DOI:10.4161/psb.6.7.15462 |
| [56] |
SUN H, DUAN Y K, QI X C, et al. Isolation and functional characterization of CsLsi2, a cucumber silicon efflux transporter gene[J]. Ann Bot, 2018, 122(4): 641-648. DOI:10.1093/aob/mcy103 |
| [57] |
VIVANCOS J, DESHMUKH R, GRÉGOIRE C, et al. Identification and characterization of silicon efflux transporters in horsetail (Equisetum arvense)[J]. J Plant Physiol, 2016, 200: 82-89. DOI:10.1016/j.jplph.2016.06.011 |
| [58] |
COSKUN D, DESHMUKH R, SHIVARAJ S M, et al. Lsi2: A black box in plant silicon transport[J]. Plant Soil, 2021, 466(1/2): 1-20. DOI:10.1007/s11104-021-05061-1 |
| [59] |
YAMAJI N, MA J F. Spatial distribution and temporal variation of the rice silicon transporter Lsi1[J]. Plant Physiol, 2007, 143(3): 1306-1313. DOI:10.1104/pp.106.093005 |
| [60] |
YAMAJI N, MA J F. Further characterization of a rice silicon efflux transporter, Lsi2[J]. Soil Sci Plant Nutr, 2011, 57(2): 259-264. DOI:10.1080/00380768.2011.565480 |
| [61] |
MITANI N, YAMAJI N, MA J F. Characterization of substrate specificity of a rice silicon transporter, Lsi1[J]. Pflugers Arch Europ J Phsiol, 2008, 456(4): 679-686. DOI:10.1007/s00424-007-0408-y |
| [62] |
WANG H S, YU C, FAN P P, et al. Identification of two cucumber putative silicon transporter genes in Cucumis sativus[J]. J Plant Growth Regul, 2015, 34(2): 332-338. DOI:10.1007/s00344-014-9466-5 |
| [63] |
MITANI-UENO N, YAMAJI N, MA J F. High silicon accumulation in the shoot is required for down-regulating the expression of Si transporter genes in rice[J]. Plant Cell Physiol, 2016, 57(12): 2510-2518. DOI:10.1093/pcp/pcw163 |
| [64] |
LI Z, KHAN M U, YAN X, et al. Deciphering the molecular mechanisms of chilling tolerance in Lsi1-overexpressing rice[J]. Int J Mol Sci, 2022, 23(9): 4667. DOI:10.3390/ijms23094667 |
| [65] |
MA J F, YAMAJI N, MITANI N, et al. Transporters of arsenite in rice and their role in arsenic accumulation in rice grain[J]. Proc Natl Acad Sci USA, 2008, 105(29): 9931-9935. DOI:10.1073/pnas.0802361105 |
| [66] |
WANG F Z, CHEN M X, YU L J, et al. OsARM1, an R2R3 MYB transcription factor, is involved in regulation of the response to arsenic stress in rice[J]. Front Plant Sci, 2017, 8: 1868. DOI:10.3389/fpls.2017.01868 |
| [67] |
KAUR H, GREGER M. A review on Si uptake and transport system[J]. Plants (Basel), 2019, 8(4): 81. DOI:10.3390/plants8040081 |
| [68] |
MANDLIK R, THAKRAL V, RATURI G, et al. Significance of silicon uptake, transport, and deposition in plants[J]. J Exp Bot, 2020, 71(21): 6703-6718. DOI:10.1093/jxb/eraa301 |
| [69] |
GAUR S, KUMAR J, KUMAR D, et al. Fascinating impact of silicon and silicon transporters in plants: A review[J]. Ecotoxicol Environ Saf, 2020, 202: 110885. DOI:10.1016/j.ecoenv.2020.110885 |
| [70] |
MITANI-UENO N, MA J F. Linking transport system of silicon with its accumulation in different plant species[J]. Soil Sci Plant Nutr, 2021, 67(1): 10-17. DOI:10.1080/00380768.2020.1845972 |
| [71] |
WANG Z G, ZHANG B L, CHEN Z W, et al. Three OsMYB36 members redundantly regulate Casparian strip formation at the root endodermis[J]. Plant Cell, 2022, 34(8): 2948-2968. DOI:10.1093/plcell/koac140 |
| [72] |
FRY S C, NESSELRODE B H W A, MILLER J G, et al. Mixed-linkage (1→3, 1→4)-β-d-glucan is a major hemicellulose of Equisetum (horsetail) cell walls[J]. New Phytol, 2008, 179(1): 104-115. DOI:10.1111/j.1469-8137.2008.02435.x |
| [73] |
ELBAUM R, MELAMED-BESSUDO C, TUROSS N, et al. New methods to isolate organic materials from silicified phytoliths reveal fragmented glycoproteins but no DNA[J]. Quat Int, 2009, 193(1/2): 11-19. DOI:10.1016/j.quaint.2007.07.006 |
| [74] |
HARRISON C C. Evidence for intramineral macromolecules containing protein from plant silicas[J]. Phytochemistry, 1996, 41(1): 37-42. DOI:10.1016/0031-9422(95)00576-5 |
| [75] |
GŁAZOWSKA S, BALDWIN L, MRAVEC J, et al. The impact of silicon on cell wall composition and enzymatic saccharification of Brachypodium distachyon[J]. Biotechnol Biofuels, 2018, 11(1): 171. DOI:10.1186/s13068-018-1166-0 |
| [76] |
PU J B, WANG L J, ZHANG W J, et al. Organically-bound silicon enhances resistance to enzymatic degradation and nanomechanical properties of rice plant cell walls[J]. Carbohydr Polym, 2021, 266: 118057. DOI:10.1016/j.carbpol.2021.118057 |
 2024, Vol. 32
2024, Vol. 32


