钙离子(Ca2+)作为植物体内的一种第二信使, 在时空上的浓度变化形成钙信号。钙信号在振幅、频率和定位上具有特异性,是连接体内外刺激与细胞响应及基因表达的关键信号之一[1]。
作为一类最重要的Ca2+感受器,钙调素(calmo-dulin, CaM)是真核生物特有、高度保守且广泛存在的Ca2+结合蛋白,具有4个能与Ca2+结合的EF-hand[2–3]。当Ca2+与CaM结合时,CaM分子发生构象变化, 继而与靶标结合而传递钙信号[1]。能与CaM结合的靶标称为钙调素结合蛋白(CaM-binding protein, CaMBP), 根据与CaM结合时是否需要Ca2+,可以将其分为Ca2+依赖型和Ca2+不依赖型[5]。据Poovaiah等[6]估计,拟南芥(Arabidopsis thaliana)中至少有500个CaMBP。
在植物中,CaM也是一类极为重要的第二信使, 参与生长发育和对外界刺激响应的调节[6]。拟南芥中共有7个CaM基因,编码4种经典CaM亚型(CaM1/4、CaM2/3/5、CaM6和CaM7)[7];不同蛋白间只有少数几个氨基酸残基不同[8]。CaM处于高度选择压力下来保持高水平的保守型,可能是植物体需要同时转录多个基因来保持高的基因表达水平, 或者是CaM家族具有各自不同的特定功能[9]。因此,确定不同CaM基因的结构及功能的异同, 对于阐明CaM家族的进化意义和同种型基因之间功能关系有重要意义[8]。
植物细胞中存在大量由不同数目内含子间隔而形成的断裂基因,一个基因在不同时空或在不同内外刺激时产生不同mRNA,进而产生不同蛋白的方式称为选择性剪接(alternative splicing, AS),可导致基因功能的多样性[10]。拟南芥中的AS类型有内含子保留(约40%)、选择性5′或3′剪接位点(约23%)、外显子跳过(约8%)及少量其他类型[11–12]。AS的功能大致分为:(1) 增加蛋白的多样性;(2) 改变蛋白的修饰位点;(3) 干扰性小肽的负调节效应;(4) 调节转录因子的活性[13]。由拟南芥数据库(https://www.arabidopsis.org/)可知,AtCaM5的核苷酸序列存在2个内含子,有2种不同的剪接方式,第2个内含子中含有终止密码子。如果第2个内含子保留,则会使翻译提前终止,产生149个氨基酸残基的CaM5.1; 如果第2个内含子被剪切,则产生含有181个氨基酸残基的CaM5.3。
IQM家族是一个含有IQ基序的CaMBP家族[14]。我们前期研究表明,拟南芥钙调素结合蛋白IQM3 (IQ-motif containing protein 3)可以在酵母细胞中与包括CaM5.1在内的CaM亚型成员结合,但是不能与CaM5.3结合。CaM5的选择性剪接产生2种类型的蛋白,推测他们具有不同的功能,因此, 进一步分析CaM5.3的结构和蛋白质结合活性的关系十分必要。
1 材料和方法 1.1 材料本研究所用CDS由拟南芥野生型Col的RNA反转录cDNA扩增得到,pGBKT7质粒、pGADT7质粒、大肠杆菌DH5α、酵母菌AH109均为实验室保存;逆转录酶、高保真Taq酶、胶回收试剂盒、质粒提取试剂盒均购自TaKaRa宝生物工程(大连)公司,同源重组酶购自南京诺唯赞公司,引物合成和测序服务由上海生工生物公司完成。
1.2 方法 1.2.1 载体构建首先,在拟南芥数据库中,根据基因编号查询CDS,设计相关引物(表 1)。其次,以拟南芥cDNA为模版,先用高保真Taq酶进行扩增,再用同源重组酶将扩增产物与经EcoR I线性化载体进行同源重组,分别得到pGBKT7-IQM3和pGADT7-CaM1-7。最后,将重组质粒转化大肠杆菌DH5α,挑取阳性克隆,送生工生物公司完成测序,提取质粒备用。
| 表 1 本研究所用克隆引物序列 Table 1 Cloning primer sequences used in this research |
CaM5.1-CTDCML10载体的构建:先用引物9和10扩增CaM5.1的CDS,再用引物17和18扩增CML10的CTD,然后用引物9和10,对上述扩增产物的混合物进行重叠延伸PCR扩增,最后用同源重组将产物克隆到pGADT7载体。
1.2.2 IQM3蛋白毒性和转录自激活性分析利用醋酸锂(LiAc)法制备酵母AH109感受态细胞备用。将pGBKT7-IQM3单独转化酵母感受态细胞,涂布于SD/-Trp培养基上以分析IQM3的毒性;将pGBKT7-IQM3和pGADT7同时转化酵母感受态细胞,涂布于SD/-Leu/-Trp培养基上,随后挑单菌落涂布于SD/-His/-Leu/Trp/-Ade营养缺陷培养基以分析IQM3的转录自激活性分析。
1.2.3 酵母双杂交试验将pGBKT7-IQM3分别与pGADT7-CaM1.1/2.1/3.1/4.1/5.1/5.3/6.1/7.1/CaM5.1-CTDCML10同时转化酵母感受态细胞,涂布于SD/-Leu/-Trp营养缺陷培养基,随后挑单菌落涂布于SD/-His/-Leu/Trp/-Ade/+X-α-gal营养缺陷培养基,30 ℃倒置培养4~5 d。实验重复3次。
1.2.4 生物信息学分析利用Snapgene (4.2.4)软件中的“Align protein sequence”-Clustal Omega方法对CaM1.1/2.1/3.1/4.1/5.1/5.3/6.1/7.1的氨基酸序列进行比对,序列按照CaM家族进化树亲缘关系排序(1/4、6、7、2/3/5)。
因为在已发表的蛋白数据库PDB (protein data bank) (https://www.rcsb.org/)中未找到CaM5.3的空间结构,所以在预测的蛋白数据库Alpha Fold Protein Structure Database (https://alphafold.ebi.ac.uk/)查找, 结果表明,CaM5.1和CaM5.3的Uniprot ID分别为Q682T9和F4IVN6。
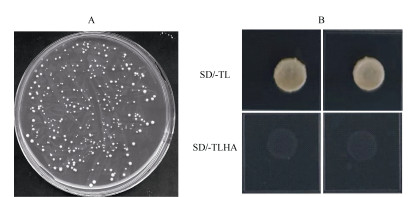
2 结果和分析 2.1 IQM3蛋白毒性和转录自激活性分析要利用某蛋白进行酵母双杂交实验,首先必须确定其是否具有毒性和转录自激活活性。因此,构建pGBKT7-IQM3载体,进行IQM3蛋白毒性及自激活活性分析。图 1: A可见,pGBKT7-IQM3的转基因酵母能在SD/-His中生长,说明IQM3蛋白没有毒性。图 1: B可见,空载体pGADT7和pGBKT7-IQM3的转基因酵母不能在SD/-His/-Leu/Trp/-Ade/上生长,提示IQM3没有自激活活性。因此,pGBKT7-IQM3可用于酵母双杂交分析。
|
图 1 IQM3毒性(A)和自激活分析(B)。SD/-Trp: 缺Trp的培养基;SD/-TLHA/: 缺Trp、Leu、His和Ade的培养基。下同 Fig. 1 Toxicity detection (A) and self-activation (B) of IQM3 protein. SD/-Trp: -Trp medium; SD/-TLHA/: -Trp-Leu-His-Ade medium. The same below |
拟南芥IQM家族的6个成员都含有1个IQ基序[15],在酵母和植物细胞中IQM1通过IQ基序与CaM5.1结合[16]。为了确定该家族的另一成员IQM3是否也具有CaM结合活性,进行IQM3与CaM家
族不同成员的酵母双杂交试验。结果表明,在SD/-Leu/-Trp培养基中,所有的组合均能形成菌落(图 2),说明共转化成功;在SD/-His/-Leu/Trp/-Ade/+X-α-gal培养基中,除pGBKT7分别与pGADT7-CaM1.1/2.1/3.1/4.1/5.3/6.1/7.1/CaM5.3的组合(未展示)和pGBKT7-IQM3/pGADT7-CaM5.3组合没有形成菌落外,其他组合形成了菌落且变蓝色,说明IQM3能在酵母中与CaM1.1/2.1/3.1/4.1/6.1/7.1/结合。

|
图 2 IQM3与CaM家族成员的酵母双杂交分析。SD/-TL: 缺Trp和Leu的培养基;SD/-TLHA/+X-α-Gal:缺Trp、Leu、His和Ade,含X-α-Gal的培养基。1~7分别为pGBKT7-IQM3与PGADT7-CaM1、CaM2、CaM3、CaM4、CaM5.3、CaM6、CaM7。下同 Fig. 2 Yeast two-hybrid analysis of IQM3 and CaM family members. SD/-TL: -Trp-Leu medium; SD/-TLHA/X-α-Gal: -Trp-Leu-His-Ade/+X-α-Gal medium. 1-7 are pGBKT7-IQM3 combined with PGADT7-CaM1, CaM2, CaM3, CaM4, CaM5.3, CaM6, CaM7, respectively. The same below |
为了探究CaM5.3不能和IQM3结合的原因, 进行了相关生物信息学分析。在拟南芥数据库中获取CaM5的2个剪接体所编码的氨基酸序列:CaM5.1和CaM5.3 (未发现命名为CaM5.2的剪接体),与从该数据库获取CaM家族其他成员的氨基酸序列进行比对(图 3)。结果表明,CaM5.3在第4个EF-hand末端还有1个由32个氨基酸残基组成的C-末端序列(C-terminal domain, CTD),其余序列与CaM2.1、CaM3.1和CaM5.1完全相同。因此,CaM5.3的CTD可能是阻碍CaM5.3与IQM3结合的原因。

|
图 3 拟南芥CaM家族氨基酸序列的同源性比对。红框: EF-hand保守区; *: 高度保守区; ∶: 相似序列。 Fig. 3 Homology alignment of amino acid sequences of CaM family in Arabidopsis thaliana. Red box: EF-hand conserved region; *: Highly conserved sequences; ∶: Similar sequences. |
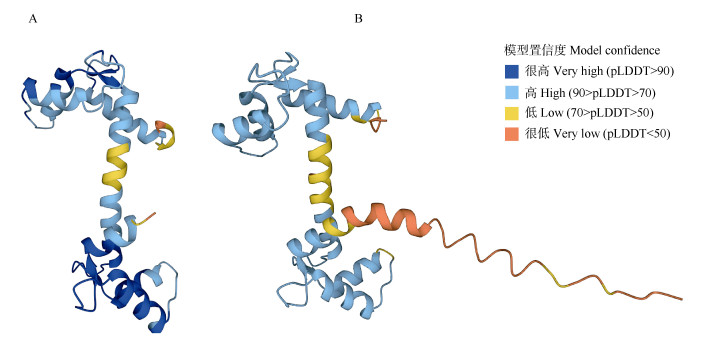
为了进一步分析CaM5.3的特异性,有必要了解其空间结构,但在最主要收集已发表的蛋白质结构数据库PDB (https://www.rcsb.org/)中未找到该蛋白的空间结构。因此,利用Alpha Fold Protein Structure Database (https://alphafold.ebi.ac.uk/)进行预测。从图 4可见,预测的CaM5.1的构象与PDB中报道的该蛋白的构象相似,整个蛋白呈哑铃型,其N-端及相邻的2个EF-hand、C-端及相邻的2个EF-hand分别位于哑铃的两“铃”,中央的α-螺旋形成两铃的“柄”;CaM5.3与CaM5.1相比,C-端多出了CTD。推测这个CTD的存在可能影响该蛋白的构象,进而影响与IQM3的结合。
|
图 4 CaM5.1 (A)与CaM5.3 (B)的三维结构预测图。pLDDT: 预测的lDDT-Cα值,每个残基的局部置信度,pLDDT越高,置信度越高,pLDDT低表示该区域可能是独立的非结构域。 Fig. 4 Predicted 3D structures of CaM5.1 (A) and CaM5.3 (B). pLDDT: Predicted value of lDDT-Cα, local confidence for each residue, the higher pLDDT the higher confidence, and low pLDDT indicates that the region may be an independent non-domain. |
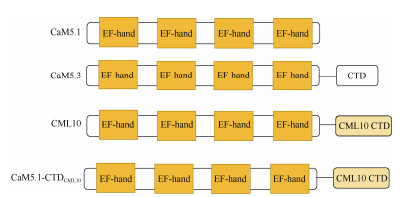
CML10是一个与CaM家族同源性较高的CML家族成员,其结构与CaM5.3相似,除具有4个EF-hand外,其C-末端也有1个CTD,由45个氨基酸残基组成(图 5),该序列影响CML10与磷酸甘露糖变位酶(Phosphomannomutase, PMM)的结合[17]。CML10的CTD与CaM5.3的CTD同源性较低,其可以用于替换CaM5.3的CTD,验证CaM5.3的CTD是阻碍CaM5.3与IQM3在酵母中结合的原因。因此,将CML10的CTD连接到CaM5.1的CDS的C-末端(图 5),构建pGADT7-CaM5.1-CTDCML10载体, 与IQM3进行酵母双杂交分析(图 6)。
|
图 5 CaM5.1、CaM5.3、CML10、CaM5.1-CTDCML10蛋白的基序组成 Fig. 5 Motif composition of CaM5.1, CaM5.3, CML10 and CaM5.1-CTDCML10 |
|
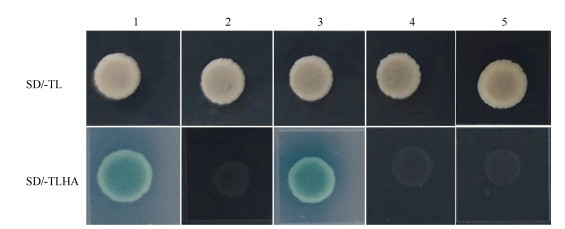
图 6 CaM5.1、CaM5.3和CaM5.1-CTDCML10分别与IQM3的酵母双杂交。1: pGBKT7-53/pGADT7-T,阳性对照;2: pGBKT7/pGADT7,阴性对照;3: pGBKT7-IQM3/pGADT-CaM5.1; 4: pGBKT7-IQM3/pGADT-CaM5.3; 5: pGBKT7-IQM3/pGADT-CaM5.1-CTDCML10。 Fig. 6 Yeast two-hybrid of IQM3 with CaM5.1, CaM5.3 and CaM5.1-CTDCML10. 1: pGBKT7-53/pGADT7-T, positive control; 2: pGBKT7/pGADT7, negative control; 3: pGBKT7-IQM3/pGADT-CaM5.1; 4: pGBKT7-IQM3/pGADT-CaM5.3; 5: pGBKT7-IQM3/pGADT-CaM5.1-CTDCML10. |
在SD/-TLHA/X-α-Gal培养基中,阳性对照能正常生长且变蓝色(图 6: 1),阴性对照pGBKT7分别与pGADT7-CaM5.1/5.3/5.1-CTDCML10的组合均不能形成菌落(未展示);IQM3仅能在酵母中与CaM5.1发生相互作用(图 6: 3),不能与CaM5.3 (图 6: 4)和CaM5.1-CTDCML10发生相互作用(图 6: 5)。这表明在CaM5.1的C-端增加CML10的CTD会阻碍所得重组蛋白与IQM3在酵母中的结合,提示CaM5.3不能与IQM3结合的原因与CaM5的CTD有关。
3 讨论和结论本研究表明,拟南芥CaM5的2个不同剪接体CaM5.1和CaM5.3具有不同的生化功能,在酵母中,CaM5.1能与IQM3结合,而CaM5.3不能与IQM3结合。生物信息学分析表明,CaM5.3比CaM5.1多出1个由35个氨基酸残基组成的CTD,可能阻碍CaM5.3与IQM3的结合。在CaM5.1的C-末端接上CML10的CTD干扰了所得重组蛋白与IQM3的结合,证实CaM5.3的CTD阻碍了其与IQM3的结合。可见,拟南芥CaM5的可变剪接方式影响其蛋白结合活性。
当CaM与Ca2+结合,会使CaM中EF-hand的2个α螺旋角度变为垂直,进而引起CaM分子表面特性发生改变,形成易于CaMBP结合的疏水表面和疏水穴[18]。当EF-hand没有与Ca2+结合时,CaM的N-端会维持关闭状态,不能与CaMBP结合;而C-端会在半开和关闭的状态间变化,从而还能与CaMBP结合[19–20]。左娜等[21]归纳总结CaM与CaMBP的相互作用主要是依靠疏水作用力,而非静电作用力。因此,CaM5.3不能与IQM3发生蛋白质间相互作用的主要原因可能是CaM5.3末端CTD在空间结构上阻挡了CaM5.3C-末端结构域的疏水表面或疏水作用力。而IQM3是一个钙不依赖的CaMBP[15], 因此推测,在没有Ca2+的情况下,CaM5.3的N-末端结构域维持关闭状态,C-末端的CTD阻挡了IQM3与CaM5.3的结合。CaM5.3的CTD改变了CaM5.3的靶蛋白的种类,使CaM5.3具有不同于常规CaM的生物功能。
尽管本研究从生化水平上揭示了CaM5.1和CaM5.3这2种剪接体具有不同的蛋白结合活性, 但是CaM5基因的可变剪接的生物学功能未知,进一步研究可聚焦于CaM5.1和CaM5.3在植物体内的时空表达、细胞定位以及与CaM5.1和CaM5.3特异结合蛋白及其信号通路的分析。
| [1] |
MEDVEDEV S S. Principles of calcium signal generation and transduction in plant cells[J]. Russ J Plant Physiol, 2018, 65(6): 771-783. DOI:10.1134/S1021443718060109 |
| [2] |
YUN C H, BAI J, SUN D Y, et al. Structure of potato calmodulin PCM6: The first report of the three-dimensional structure of a plant calmodulin[J]. Acta Crystallogr D Biol Cryst, 2004, 60(7): 1214-1219. DOI:10.1107/S0907444904009771 |
| [3] |
ISHIDA H, RAINALDI M, VOGEL H J. Structural studies of soybean calmodulin isoform 4 bound to the calmodulin-binding domain of tobacco mitogen-activated protein kinase phosphatase-1 provide insights into a sequential target binding mode[J]. J Biol Chem, 2009, 284(41): 28292-28305. DOI:10.1074/jbc.M109.025080 |
| [4] |
ANDREWS C, XU Y T, KIRBERGER M, et al. Structural aspects and prediction of calmodulin-binding proteins[J]. Int J Mol Sci, 2020, 22(1): 308. DOI:10.3390/ijms22010308 |
| [5] |
O'DAY D H. CaMBOT: Profiling and characterizing calmodulin-binding proteins[J]. Cell Signal, 2003, 15(4): 347-354. DOI:10.1016/S0898-6568(02)00116-X |
| [6] |
POOVAIAH B W, DU L Q, WANG H Z, et al. Recent advances in calcium/calmodulin-mediated signaling with an emphasis on plant-microbe interactions[J]. Plant Physiol, 2013, 163(2): 531-542. DOI:10.1104/pp.113.220780 |
| [7] |
MCCORMACK E, BRAAM J. Calmodulins and related potential calcium sensors of Arabidopsis[J]. New Phytol, 2003, 159(3): 585-598. DOI:10.1046/j.1469-8137.2003.00845.x |
| [8] |
LANDONI M, DE FRANCESCO A, GALBIATI M, et al. A loss-of-function mutation in Calmodulin 2 gene affects pollen germination in Arabidopsis thaliana[J]. Plant Mol Biol, 2010, 74(3): 235-247. DOI:10.1007/s11103-010-9669-5 |
| [9] |
MCCORMACK E, TSAI Y C, BRAAM J. Handling calcium signaling: Arabidopsis CaMs and CMLs[J]. Trends Plant Sci, 2005, 10(8): 383-389. DOI:10.1016/j.tplants.2005.07.001 |
| [10] |
ZOU Z M, ZOU H X, ZHANG H, et al. Studies on alternative splicing variants of rice OsHDT703 gene[J]. Acta Agric Boreali-Sin, 2019, 34(4): 9-15. 邹志明, 邹海霞, 张焕, 等. 水稻OsHDT703基因的可变剪接体分析[J]. 华北农学报, 2019, 34(4): 9-15. DOI:10.7668/hbnxb.201751587 |
| [11] |
ECKARDT N A. The plant cell reviews alternative splicing[J]. Plant Cell, 2013, 25(10): 3639. DOI:10.1105/tpc.113.251013 |
| [12] |
REDDY A S N, MARQUEZ Y, KALYNA M, et al. Complexity of the alternative splicing landscape in plants[J]. Plant Cell, 2013, 25(10): 3657-3683. DOI:10.1105/tpc.113.117523 |
| [13] |
HAO X F, WU Z J, MA T, et al. Alternative splicing of plant genes: Full of change, sail with wind[J]. Chin Bull Bot, 2022, 57(5): 661-672. 郝雪峰, 毋张晶, 马甜, 等. 植物基因选择性剪接: 断舍离合, 达权知变[J]. 植物学报, 2022, 57(5): 661-672. DOI:10.11983/CBB22041 |
| [14] |
TIAN C E, ZHOU Y P. Research progress in plant IQ motif-containing calmodulin-binding proteins[J]. Chin Bull Bot, 2013, 48(4): 447-460. 田长恩, 周玉萍. 植物具IQ基序的钙调素结合蛋白的研究进展[J]. 植物学报, 2013, 48(4): 447-460. DOI:10.3724/SP.J.1259.2013.00447 |
| [15] |
ZHOU Y P, CHEN Y Z, YAMAMOTO K T, et al. Sequence and expression analysis of the Arabidopsis IQM family[J]. Acta Physiol Plant, 2010, 32(1): 191-198. DOI:10.1007/s11738-009-0398-9 |
| [16] |
ZHOU Y P, DUAN J, FUJIBE T, et al. AtIQM1, a novel calmodulin-binding protein, is involved in stomatal movement in Arabidopsis[J]. Plant Mol Biol, 2012, 79(4): 333-346. DOI:10.1007/s11103-012-9915-0 |
| [17] |
CHO K M, NGUYEN H T K, KIM S Y, et al. CML 10, a variant of calmodulin, modulates ascorbic acid synthesis[J]. New Phytol, 2016, 209(2): 664-678. DOI:10.1111/nph.13612 |
| [18] |
ZHANG M J, TANAKA T, IKURA M. Calcium-induced conformational transition revealed by the solution structure of apo calmodulin[J]. Nat Struct Biol, 1995, 2(9): 758-767. DOI:10.1038/nsb0995-758 |
| [19] |
FENG Y D, WEI Q. Diversity of the modes for calmodulin binding to its targets[J]. Prog Chem, 2012, 24(10): 2028-2039. 冯业丹, 魏群. 钙调素与靶蛋白结合模式的多样性[J]. 化学进展, 2012, 24(10): 2028-2039. |
| [20] |
WU C T, FAN T, LÜ T X, et al. Recent advances in calmodulin binding protein involved in plant responses to adversity stresses[J]. J Trop Subtrop Bot, 2022, 30(6): 823-834. 吴春婷, 范甜, 吕天晓, 等. 钙调素结合蛋白参与调控植物逆境胁迫的研究进展[J]. 热带亚热带植物学报, 2022, 30(6): 823-834. DOI:10.11926/jtsb.4711 |
| [21] |
ZUO N, CHEN J, LÜ Y G. Advance progress in plant calmodulin and calmodulin-binding proteins structure biology[J]. Cereals Oils, 2016, 29(9): 1-5. 左娜, 陈洁, 吕莹果. 植物钙调素及其结合蛋白的结构生物学进展[J]. 粮食与油脂, 2016, 29(9): 1-5. DOI:10.3969/j.issn.1008-9578.2016.09.001 |
 2024, Vol. 32
2024, Vol. 32


