2. 华南国家植物园, 广州 510650;
3. 中国科学院大学, 北京 100049
2. South China National Botanical Garden, Guangzhou 510650, China;
3. University of Chinese Academy of Sciences, Beijing 100049, China
种子是裸子植物和被子植物的特殊繁殖体。种子萌发是种子植物生命周期中的重要发育事件,它意味着植物开始了新的生命周期,决定了植物何时进入自然或农业生态系统[1]。在自然条件下,为抵御不良生长环境,种子会通过改变自身代谢水平调控休眠过程,在合适的时间以及环境条件下启动种子萌发这一生理过程。种子的早萌和胎萌对植物后期结实的产量及质量有很大影响。因此,种子萌发与作物收成及作物经济效益关系十分密切,对农业生产有着重要意义。
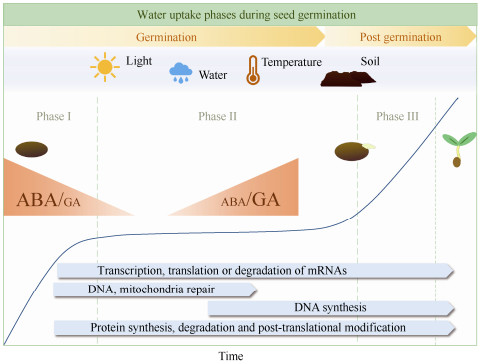
种子萌发根据吸水过程主要可分为3个阶段, 包括吸水期、滞缓期和重新吸水期[2]。种子萌发过程中涉及多种生物学变化,包括DNA修复,线粒体合成,mRNA合成,蛋白合成、降解以及翻译后修饰等[3–4]。种子的萌发过程受到外界环境信号如光照[5]、温度[6]、水分[7]和土壤硝酸盐[8],以及内源激素信号[5–8]如赤霉素(GA)、脱落酸(ABA)、乙烯和油菜素内酯等水平的影响[9–12](图 1)。
2 光信号转导途径光照是驱动植物碳代谢和地球生命活动的重要环境因素,也是植物生长发育过程中的重要环境调控因子。由于植物在自然界中固着生活,从而进化出多种生长方式及生理反应,以应对不断变化的环境条件[13]。光形态建成和暗形态建成是植物生长发育的两个重要过程。不同的光暗条件、光照强度、光质等因素极大地影响着植物的光形态建成、光合作用、碳水化合物的代谢、以及其他生理生化指标。高等植物利用光受体感受自然环境中光的强度、光质、入射光方向以及光周期变化等信号,并利用这些信息来调控植物的生长和发育过程,包括种子萌发、幼苗生长、去黄化、向光性、叶片生长、避荫反应、开花周期等[14]。
2.1 光受体植物已经进化出多种光受体蛋白来接收周围光信号,包括感知波长为600~750 nm的红光(R)与远红光(FR)信号的光敏色素(phytochromes, PHYs), 感知波长为320~500 nm的蓝光(B)与紫外光A (UV-A)信号的隐花色素(cryptochromes, CRYs)、向光素(phototropins, PHOTs)、ZTL/FKF1/LKP2、AUREO (aureochrome, AUREO),以及感知波长为280~320 nm紫外光B (UV-B)的UVRESISTANCE LOCUS8 (UVR8)[15–17]。
尽管植物的光感受器种类繁多,但它们的功能遵循着几个共同的机制。对于光敏色素、隐花色素以及向光素,其活性形式依赖于自磷酸化的二聚体的形成。当吸收较短波长的光,如蓝光(B)或红光(R)时,光受体被激活;而当吸收较长波长光,如绿光(G)或远红光(FR)时,光受体则失活[18–20]。此外,这些光受体通过选择性地与转录因子或E3泛素连接酶相互作用来介导光信号,调节转录因子的稳定性,从而重构整个转录过程[21]。
2.2 光敏色素介导的光信号途径光敏色素(PHYs)是植物中参与光调控生长发育的重要因子,对种子萌发、幼苗去黄化、向重力反应、避荫反应、叶片生长、开花周期以及向光性等生理过程具有调控作用[22]。光敏色素全蛋白由可溶性的光敏色素脱辅基和生色团构成。光敏色素以二聚体存在,且在体内有2种差异明显但可通过光发生可逆转变的形式,即在黑暗或者远红光下,光敏色素以无生物活性的Pr形式存在;当植物接收红光时,光敏色素会转变为具有生物活性的Pfr形式[23]。
在模式植物拟南芥(Arabidopsis thaliana)中,有5种光敏色素蛋白:PHYA、PHYB、PHYC、PHYD和PHYE。其中,PHYA主要在黑暗中生长的幼苗中富集,而在照射红光或者白光后其水平快速下降[24]。PHYB-PHYE在光下稳定,且在光下生长的植物中PHYB是主要存在的光敏色素[25]。在萌动的种子中,PHYB的积累较PHYA更为明显,因此PHYB在光调控的种子萌发过程中起到主导作用[26]。同时,PHYB和PHYA在不同的光照条件下可发生Pr形式到Pfr形式的可逆转变的特性,使其成为高等植物依赖于R/FR调控种子萌发的开关。研究还表明,在种子吸胀早期,FR会抑制PHYB依赖的萌发过程,随后会启动PHYA依赖的萌发途径[27]。
3 光调控种子萌发的转录调控机制前人根据萌发对光照反应的不同,将种子区分为需光种子(或感光种子)、忌光种子和中性种子[28]。典型的需光种子有莴苣(Lactuca sativa)、烟草(Nico-tiana tabacum)、番茄(Lycopersicon esculentum)、生菜(Lactuca sativa var. ramosa)、木麻黄(Casuarina equi-setifolia)和模式植物拟南芥等。通常需光种子的粒型较小(1~200 µg),因而又被称为小种子[29]。1952年,Borthwick等报道红光可促进莴苣种子萌发,远红光则抑制种子萌发[30]。随后的研究表明,光敏色素作为主要的光受体调节了植物的种子萌发过程。通常认为,光敏色素的最终输出途径是通过调控植物体内ABA和GA的相对含量,从而调控植物的种子萌发。
早期研究表明,光敏色素的信号转导涉及1个bHLH转录因子——光敏色素互作蛋白PIF1。PIF1作用于PHYA和PHYB的下游,可通过直接或间接的方式调控植物体内ABA和GA的合成和信号转导途径调控种子萌发。随后的研究表明,光敏色素还可直接调控下游生长素信号与细胞壁松弛和软化等相关基因的表达[31]。在种子萌发过程中,生长发育途径转变与伴随着基因的转录激活与染色质结构变化。近年来,随着表观遗传学的学科发展,越来越多的研究表明,表观遗传修饰作用广泛参与了光调控的植物种子萌发过程。
3.1 PIF1转录因子介导的转录调控作用在光信号转导途径中,PIF1作为主效的负调控因子参与了光敏色素介导的拟南芥种子萌发过程[32]。在黑暗或者远红光条件下,种子中的PIF1蛋白大量累积并抑制萌发相关基因的表达;红光照射后,有生物活性的PHYA和PHYB与PIF1发生蛋白相互作用,诱导PIF1发生泛素化修饰,并通过泛素/26S蛋白酶途径降解。PIF1对种子萌发的抑制作用被解除后,种子萌发得以起始[32–33]。
PIF1可通过直接调控植物激素信号途径与细胞壁代谢途径相关基因的表达调控种子萌发[34]。在远红光光照条件下,PIF1在细胞核中积累并激活DAG1基因的转录,进而抑制GA3ox1的表达和阻断GA的生物合成,最终抑制种子萌发[35]。SOMNUS (SOM)是一种CCCH型锌指蛋白,是光敏色素介导种子萌发途径中的负调控因子[36]。PIF1通过直接与SOM的启动子结合来激活其转录。种子中累积的SOM蛋白通过诱导ABA合成基因ABA1、NCED6和NCED9的表达,并抑制ABA分解代谢基因CYP707A2的表达,导致ABA水平升高;同时,SOM通过抑制GA合成基因GA3ox1、GA3ox2的表达,诱导GA分解代谢基因GA2ox2的表达来抑制GA的生物合成[36]。PIF1对靶基因表达的转录调控作用涉及多个转录因子或协同调控因子。最近的研究表明,PIF1与小RNA分子miR408的启动子结合,抑制miR408的积累,并调节对萌发有抑制作用的植物花青素(Plantacya-nin, PCY)的丰度,从而调控种子萌发[37]。同时, 转录因子Long hypocotyl in far-red1 (HFR1)作为PIF1的转录辅助调节因子,通过阻止PIF1与下游萌发相关基因的结合来阻断PIF1的转录活性,进而促进种子的萌发过程[38]。
PIF1的稳定性和转录活性由多种途径共同调控。研究表明De-Etiolated 1 (DET1)作用于HFR1和PIF1上游,降解HFR1蛋白,并增强PIF1的稳定性。通过实验验证和数据建模,表明PHYB-DET1-HFR1-PIF1和PHYB-DET1-Protease-PIF1形成2条独立的信号通路,分别参与了光介导的种子萌发过程[39]。在PHYB-DET1-HFR1-PIF1信号通路中,DET1与HFR1直接互作,并通过组成COP10-DET1-DDB1-CUL4 E3泛素连接酶复合体降解HFR1,进而释放PIF1的转录活性从而抑制种子萌发;在PHYB-DET1-Protease-PIF1信号通路中,DET1-COP10与PIF1发生蛋白互作,抑制蛋白酶体对PIF1的降解从而抑制种子萌发[39]。最近研究表明,RVE1和PIF1直接结合在彼此的启动子上,相互激活表达,形成1个转录反馈环,并通过增强彼此的DNA结合能力和转录活性,从而调控种子萌发相关基因的表达[40]。AP2/ERF是植物中较大的转录因子家族,其成员在植物各个生物过程中发挥调节功能。研究发现,光敏色素互作蛋白ERF55/ ERF58直接结合到PIF1和SOM的启动子区域,通过激活其转录,间接的调控ABA/GA代谢相关基因的表达[41]。
研究表明,活化的PHYB与SPA1发生直接相互作用来抑制COP1-SPA1复合物的活性,而COP1-SPAs E3泛素连接酶在光照下优先与磷酸化的PIF1形成复合物从而参与PIF1的光诱导降解[42–43]。拟南芥F-BOX蛋白Cold temperature germinating 10 (CTG10)与PIF1相互作用形成反馈回路,并调节其在植物中的丰度。在黑暗条件下,CTG10与PIF1在细胞核中共存并积累;种子转入照光时,CTG10促进PIF1的降解,从而促进种子萌发和后续幼苗发育的完成[44]。此外,Photoperiodic control of hypocotyl 1 (PCH1)和PCH1-LIKE在体内促进PHYB与PIF1的相互作用,从而促进PIF1的光诱导降解,同时还会抑制PIF1的DNA结合能力,负向调控PIF1靶基因的表达[45] (图 2)。
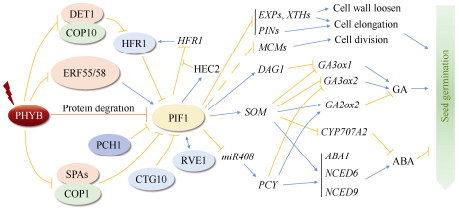
|
图 2 光敏色素互作蛋白PIF1介导的转录调控作用[32–45] Fig. 2 Phytochrome-interacting factor PIF1 mediated transcriptional regulation[32–45] |
表观遗传学是研究DNA序列不发生改变但引起可遗传的基因表达变化的学科。表观遗传方式主要包括DNA甲基化、组蛋白乙酰化、组蛋白甲基化和染色质重塑等[46]。越来越多的研究表明,多种表观遗传修饰在光介导的种子萌发过程中发挥了重要的调控作用。
研究表明,拟南芥组蛋白去乙酰化酶HDA6和光敏色素PHYB共同调控光诱导的染色质紧缩[47]。而光敏色素PHYA的转录调控与特殊的组蛋白标记相关,包括H3K9/14ac、H3K27ac、H3K4me3、H4K5ac、H4K8ac、H4K12ac和H4K16ac,并且组蛋白去乙酰化酶HDA19参与其中[48]。这说明表观遗传调控因子可通过影响光响应基因的染色质状态参与植物生长发育过程。
已有的研究表明,组蛋白去乙酰化酶HDA15作为负调控因子参与了PHYB介导的种子萌发过程。在拟南芥中HDA15缺失促进种子萌发,而超表达HDA15则抑制种子萌发。HDA15与PIF1在黑暗条件发生蛋白互作,通过降低组蛋白H3的乙酰化(H3ac)水平抑制了多种激素信号途径和细胞过程相关基因表达,从而抑制了PHYB依赖的种子萌发过程[49]。同时,组蛋白精氨酸去甲基化酶JMJ20和JMJ22通过促进GA生物合成基因GA3ox1和GA3ox2的表达促进了PHYB依赖的种子萌发过程[50]。在红光诱导的种子萌发过程中,光活化的PHYB-PIL5-SOM信号途径解除了SOM对JMJ20和JMJ22的转录抑制,诱导JMJ20和JMJ22表达,从而去除GA3ox1和GA3ox2基因位点的组蛋白精氨酸的二甲基化(H4R3me2),促进GA的生物合成和种子萌发[50]。
研究表明,组蛋白H3K4/H3K36甲基转移酶基因EARLY FLOWERING IN SHORT DAYS (EFS)作为负调控因子参与了光调控的种子萌发过程。EFS直接结合到PIF1基因座位上,通过增加结合位点上的H3K36me2和H3K36me3修饰水平,促进PIF1基因表达从而抑制种子的萌发[51]。此外,组蛋白甲基化酶SU(VAR)3-9 HOMOLOGUE 5(SUVH5)作为正调控因子参与光依赖的种子萌发过程。SUVH5功能的缺失导致了种子萌发率下降并且引起24.6%光响应转录组的改变,SUVH5还通过H3K9二甲基化作用(H3K9me2)抑制吸胀种子中ABA信号途径基因和萌发抑制因子DOG类基因表达[52]。综上所述, 不同类型的表观遗传因子通过组蛋白乙酰化和甲基化修饰作用改变染色质的结构,进而调控种子萌发相关基因的表达(图 3)。

|
图 3 表观遗传因子在光调控的种子萌发中的作用[49–52] Fig. 3 Roles of epigenetic factors in light-mediated seed germination[49–52] |
萌发是种子植物开启生命周期至关重要的阶段。而对于农业生产,适时的种子萌发是保证粮食产量和品质的关键农艺过程。红光促进而远红光抑制种子萌发的这一特殊生物学转换机制为精确控制种子萌发提供了多种思路。
首先,在农业生产中,除了常规利用赤霉素处理打破休眠促进种子萌发,也可以选择利用红光对需光种子,如生菜、番茄、胡萝卜(Daucus carota var. sativus)和莴苣等进行照射处理诱导萌发,可以有效的节约成本。对于粮食的贮藏,除了考虑环境中的湿度和氧气浓度,配合进行远红光照射,可以有效的抑制种子萌发,保证粮食的品质。其次,基于光信号传导网络的研究基础,可以通过基因工程技术改变光敏色素下游萌发相关基因,如萌发主效因子PIF1和HFR1的蛋白转录活性与表达水平,从而调控不同类型植物种子对光的敏感性和萌发能力。此外, 利用现有发掘的转录因子和表观遗传因子,结合农作物遗传位点差异分析,可以选育出对光照敏感差异的品种,如大豆(Glycine max)、黄瓜(Cucumis sativus)、玉米(Zea mays)等,并最终通过杂交育种或基因编辑的方式应用于农业育种工作中。这些工作对于有效减少种子胎萌以及改善种子难萌发的性状,均具有重要的应用前景。
| [1] |
WEITBRECHT K, MÜLLER K, LEUBNER-METZGER G. First off the mark: Tarly seed germination[J]. J Exp Bot, 2011, 62(10): 3289-3309. DOI:10.1093/jxb/err030 |
| [2] |
HAN C, YANG P F. Studies on the molecular mechanisms of seed germination[J]. Proteomics, 2015, 15(10): 1671-1679. DOI:10.1002/pmic.201400375 |
| [3] |
SANO N, RAJJOU L, NORTH H M. Lost in translation: Physiological roles of stored mRNAs in seed germination[J]. Plants (Basel), 2020, 9(3): 347. DOI:10.3390/plants9030347 |
| [4] |
PASZKIEWICZ G, GUALBERTO J M, BENAMAR A, et al. Arabidopsis seed mitochondria are bioenergetically active immediately upon imbibition and specialize via biogenesis in preparation for autotrophic growth[J]. Plant Cell, 2017, 29(1): 109-128. DOI:10.1105/tpc.16.00700 |
| [5] |
ZEIDLER M. Analysis of phytochrome-dependent seed germination in Arabidopsis[J]. Methods Mol Biol, 2022, 2494: 117-124. DOI:10.1007/978-1-0716-2297-1_8 |
| [6] |
IWASAKI M, PENFIELD S, LOPEZ-MOLINA L. Parental and environmental control of seed dormancy in Arabidopsis thaliana[J]. Annu Rev Plant Biol, 2022, 73(1): 355-378. DOI:10.1146/annurevarplant-102820-090750 |
| [7] |
FOOTITT S, CLEWES R, FEENEY M, et al. Aquaporins influence seed dormancy and germination in response to stress[J]. Plant Cell Environ, 2019, 42(8): 2325-2339. DOI:10.1111/pce.13561 |
| [8] |
FOOTITT S, HUANG Z Y, CLAY H A, et al. Temperature, light and nitrate sensing coordinate Arabidopsis seed dormancy cycling, resulting in winter and summer annual phenotypes[J]. Plant J, 2013, 74(6): 1003-1015. DOI:10.1111/tpj.12186 |
| [9] |
ZHAO H Y, ZHANG Y M, ZHENG Y. Integration of ABA, GA, and light signaling in seed germination through the regulation of ABI5[J]. Front Plant Sci, 2022, 13: 1000803. DOI:10.3389/fpls.2022.1000803 |
| [10] |
DEBEAUJON I, KOORNNEEF M. Gibberellin requirement for Arabidopsis seed germination is determined both by testa characteristics and embryonic abscisic acid[J]. Plant Physiol, 2000, 122(2): 415-424. DOI:10.1104/pp.122.2.415 |
| [11] |
KOORNNEEF M, BENTSINK L, HILHORST H. Seed dormancy and germination[J]. Curr Opin Plant Biol, 2002, 5(1): 33-36. DOI:10.1016/S1369-5266(01)00219-9 |
| [12] |
LEUBNER-METZGER G. Brassinosteroids and gibberellins promote tobacco seed germination by distinct pathways[J]. Planta, 2001, 213(5): 758-763. DOI:10.1007/s004250100542 |
| [13] |
SULLIVAN J A, DENG X W. From seed to seed: The role of photoreceptors in Arabidopsis development[J]. Dev Biol, 2003, 260(2): 289-297. DOI:10.1016/S0012-1606(03)00212-4 |
| [14] |
KAMI C, LORRAIN S, HORNITSCHEK P, et al. Light-regulated plant growth and development[J]. Curr Top Dev Biol, 2010, 91: 29-66. DOI:10.1016/S0070-2153(10)91002-8 |
| [15] |
BRIGGS W R, CHRISTIE J M. Phototropins 1 and 2: Versatile plant blue-light receptors[J]. Trends Plant Sci, 2002, 7(5): 204-210. DOI:10.1016/S1360-1385(02)02245-8 |
| [16] |
CASHMORE A R, JARILLO J A, WU Y J, et al. Cryptochromes: Blue light receptors for plants and animals[J]. Science, 1999, 284(5415): 760-765. DOI:10.1126/science.284.5415.760 |
| [17] |
SUETSUGU N, WADA M. Evolution of three LOV blue light receptor families in green plants and photosynthetic stramenopiles: Phototropin, ZTL/FKF1/LKP2 and aureochrome[J]. Plant Cell Physiol, 2013, 54(1): 8-23. DOI:10.1093/pcp/pcs165 |
| [18] |
VOITSEKHOVSKAJA O V. Phytochromes and other (photo)receptors of information in plants[J]. Russ J Plant Physiol, 2019, 66(3): 351-364. DOI:10.1134/S1021443719030154 |
| [19] |
LI Z W, JIANG G X, LIU X C, et al. Histone demethylase SlJMJ6 promotes fruit ripening by removing H3K27 methylation of ripeningrelated genes in tomato[J]. New Phytol, 2020, 227(4): 1138-1156. DOI:10.1111/nph.16590 |
| [20] |
CHENG M C, KATHARE P K, PAIK I, et al. Phytochrome signaling networks[J]. Annu Rev Plant Biol, 2021, 72: 217-244. DOI:10.1146/annurev-arplant-080620-024221 |
| [21] |
JING Y J, LIN R C. Transcriptional regulatory network of the light signaling pathways[J]. New Phytol, 2020, 227(3): 683-697. DOI:10.1111/nph.16602 |
| [22] |
FRANKLIN K A, QUAIL P H. Phytochrome functions in Arabidopsis development[J]. J Exp Bot, 2010, 61(1): 11-24. |
| [23] |
QUAIL P H. An emerging molecular map of the phytochromes[J]. Plant Cell Environ, 1997, 20(6): 657-665. DOI:10.1046/j.1365-3040.1997.d01-108.x |
| [24] |
CLOUGH R C, VIERSTRA R D. Phytochrome degradation[J]. Plant Cell Environ, 1997, 20(6): 713-721. DOI:10.1046/j.1365-3040.1997.d01-107.x |
| [25] |
SHARROCK R A, CLACK T. Patterns of expression and normalized levels of the five Arabidopsis phytochromes[J]. Plant Physiol, 2002, 130(1): 442-456. DOI:10.1104/pp.005389 |
| [26] |
SHINOMURA T, NAGATANI A, CHORY J, et al. The induction of seed germination in Arabidopsis thaliana is regulated principally by phytochrome B and secondarily by phytochrome A[J]. Plant Physiol, 1994, 104(2): 363-371. DOI:10.1104/pp.104.2.363 |
| [27] |
LEE K P, PISKUREWICZ U, TUREČKOVÁ V, et al. Spatially and genetically distinct control of seed germination by phytochromes A and B[J]. Genes Dev, 2012, 26(17): 1984-1996. DOI:10.1101/gad.194266.112 |
| [28] |
MÉRAI Z, GRAEBER K, WILHELMSSON P, et al. Aethionema arabicum: A novel model plant to study the light control of seed germination[J]. J Exp Bot, 2019, 70(12): 3313-3328. DOI:10.1093/jxb/erz146 |
| [29] |
KOUTSOVOULOU K, DAWS M I, THANOS C A. Campanulaceae: A family with small seeds that require light for germination[J]. Ann Bot, 2014, 113(1): 135-143. DOI:10.1093/aob/mct250 |
| [30] |
BORTHWICK H A, HENDRICKS S B, PARKER M W, et al. A reversible photoreaction controlling seed germination[J]. Proc Natl Acad Sci USA, 1952, 38(8): 662-666. DOI:10.1073/pnas.38.8.662 |
| [31] |
XU X S, PAIK I, ZHU L, et al. Illuminating progress in phytochromemediated light signaling pathways[J]. Trends Plant Sci, 2015, 20(10): 641-650. DOI:10.1016/j.tplants.2015.06.010 |
| [32] |
OH E, KIM J, PARK E, et al. PIL5, a phytochrome-interacting basic helix-loop-helix protein, is a key negative regulator of seed germination in Arabidopsis thaliana[J]. Plant Cell, 2004, 16(11): 3045-3058. DOI:10.1105/tpc.104.025163 |
| [33] |
OH E, YAMAGUCHI S, KAMIYA Y, et al. Light activates the degradation of PIL5 protein to promote seed germination through gibberellin in Arabidopsis[J]. Plant J, 2006, 47(1): 124-139. DOI:10.1111/j.1365-313X.2006.02773.x |
| [34] |
OH E, KANG H, YAMAGUCHI S, et al. Genome-wide analysis of genes targeted by PHYTOCHROME INTERACTING FACTOR 3-LIKE5 during seed germination in Arabidopsis[J]. Plant Cell, 2009, 21(2): 403-419. DOI:10.1105/tpc.108.064691 |
| [35] |
DE WIT M, GALVÃO V C, FANKHAUSER C. Light-mediated hormonal regulation of plant growth and development[J]. Annu Rev Plant Biol, 2016, 67(1): 513-537. DOI:10.1146/annurev-arplant-043015-112252 |
| [36] |
KIM D H, YAMAGUCHI S, LIM S, et al. SOMNUS, a CCCH-type zinc finger protein in Arabidopsis, negatively regulates light-dependent seed germination downstream of PIL5[J]. Plant Cell, 2008, 20(5): 1260-1277. DOI:10.1105/tpc.108.058859 |
| [37] |
JIANG A L, GUO Z L, PAN J W, et al. The PIF1-miR408-PLANTACYANIN repression cascade regulates light-dependent seed germination[J]. Plant Cell, 2021, 33(5): 1506-1529. DOI:10.1093/plcell/koab060 |
| [38] |
SHI H, ZHONG S W, MO X R, et al. HFR1 sequesters PIF1 to govern the transcriptional network underlying light-initiated seed germination in Arabidopsis[J]. Plant Cell, 2013, 25(10): 3770-3784. DOI:10.1105/tpc.113.117424 |
| [39] |
SHI H, WANG X, MO X R, et al. Arabidopsis DET1 degrades HFR1 but stabilizes PIF1 to precisely regulate seed germination[J]. Proc Natl Acad Sci USA, 2015, 112(12): 3817-3822. DOI:10.1073/pnas.1502405112 |
| [40] |
YANG L W, JIANG Z M, JING Y J, et al. PIF1 and RVE1 form a transcriptional feedback loop to control light-mediated seed germination in Arabidopsis[J]. J Integr Plant Bioly, 2020, 62(9): 1372-1384. DOI:10.1111/jipb.12938 |
| [41] |
LI Z L, SHEERIN D J, VON ROEPENACK-LAHAYE E, et al. The phytochrome interacting proteins ERF55 and ERF58 repress lightinduced seed germination in Arabidopsis thaliana[J]. Nat Commun, 2022, 13: 1656. DOI:10.1038/s41467-022-29315-3 |
| [42] |
LU X D, ZHOU C M, XU P B, et al. Red-light-dependent interaction of phyB with SPA1 promotes COP1-SPA1 dissociation and photomorphogenic development in Arabidopsis[J]. Mol Plant, 2015, 8(3): 467-478. DOI:10.1016/j.molp.2014.11.025 |
| [43] |
ZHU L, BU Q Y, XU X S, et al. CUL4 forms an E3 ligase with COP1 and SPA to promote light-induced degradation of PIF1[J]. Nat Commun, 2015, 6: 7245. DOI:10.1038/ncomms8245 |
| [44] |
MAJEE M, KUMAR S, KATHARE P K, et al. KELCH F-BOX protein positively influences Arabidopsis seed germination by targeting PHYTOCHROME-INTERACTING FACTOR1[J]. Proc Natl Acad Sci USA, 2018, 115(17): E4120-E4129. DOI:10.1073/pnas.1711919115 |
| [45] |
CHENG M C, ENDERLE B, KATHARE P K, et al. PCH1 and PCHL directly interact with PIF1, promote its degradation, and inhibit its transcriptional function during photomorphogenesis[J]. Mol Plant, 2020, 13(3): 499-514. DOI:10.1016/j.molp.2020.02.003 |
| [46] |
LIU X C, YANG S G, ZHAO M L, et al. Transcriptional repression by histone deacetylases in plants[J]. Mol Plant, 2014, 7(5): 764-772. DOI:10.1093/mp/ssu033 |
| [47] |
TESSADORI F, VAN ZANTEN M, PAVLOVA P, et al. Phytochrome B and histone deacetylase 6 control light-induced chromatin compaction in Arabidopsis thaliana[J]. PLoS Genet, 2009, 5(9): e1000638. DOI:10.1371/journal.pgen.1000638 |
| [48] |
JANG I C, CHUNG P J, HEMMES H, et al. Rapid and reversible light-mediated chromatin modifications of Arabidopsis phytochrome A locus[J]. Plant Cell, 2011, 23(2): 459-470. DOI:10.1105/tpc.110.080481 |
| [49] |
GU D C, CHEN C Y, ZHAO M L, et al. Identification of HDA15-PIF1 as a key repression module directing the transcriptional network of seed germination in the dark[J]. Nucl Acid Res, 2017, 45(12): 7137-7150. DOI:10.1093/nar/gkx283 |
| [50] |
CHO J N, RYU J Y, JEONG Y M, et al. Control of seed germination by light-induced histone arginine demethylation activity[J]. Dev Cell, 2012, 22(4): 736-748. DOI:10.1016/j.devcel.2012.01.024 |
| [51] |
LEE N, KANG H, LEE D, et al. A histone methyltransferase inhibits seed germination by increasing PIF1 mRNA expression in imbibed seeds[J]. Plant J, 2014, 78(2): 282-293. DOI:10.1111/tpj.12467 |
| [52] |
GU D C, JI R J, HE C M, et al. Arabidopsis histone methyltransferase SUVH5 is a positive regulator of light-mediated seed germination[J]. Front Plant Sci, 2019, 10: 841. DOI:10.3389/fpls.2019.00841 |
 2024, Vol. 32
2024, Vol. 32