温度影响植物的生长发育,低温胁迫是植物生长发育中最关键的非生物胁迫之一。低温会直接影响植物的光合系统,使植物的生长发育受到抑制, 产生冻害和冷害[1],严重时甚至死亡。叶片是植物最重要的光合器官,同时也是感知外界环境变化较为敏感的器官,能够及时对低温环境做出响应。随温度降低,冬油菜(Brassica rapa)的叶面积、气孔导度、光合速率、蒸腾速率等均逐渐下降[2]。在低温下,菠菜(Spinacia oleracea)叶片的类黄酮总量增加,增强了植株的抗逆性并使糖分积累,叶片中的类黄酮含量与抗寒性的强弱有显著相关性[3]。枳(Citrus trifoliata)实生苗的根系和叶片中的黄烷酮的含量随着低温胁迫时间的延长而增加,从而增强抗氧化胁迫的能力[4]。因此,类黄酮在植物应对低温胁迫方面发挥着重要作用。
类黄酮是植物中一类重要的次生代谢产物,其生物合成途径前期需要查尔酮合酶(CHS)、查尔酮异构酶(CHI)和黄烷酮-3-羟化酶(F3H)等3个酶家族,后期需要二氢黄酮醇-4-还原酶(DFR)、花青素合成酶(ANS),无色花青素还原酶(ANR)和花青素还原酶(LAR)等4个家族参与,经过一系列反应生成不同种类的类黄酮[5]。研究表明,随茉莉花(Brunfelsia acuminata)花瓣的颜色变浅,CHS的表达量降低[6];通过沉默非洲菊(Gerbera hybrida)中的GCHS1,花瓣颜色由粉色变为纯白色[7];通过过表达牡丹(Paeonia suffruticosa) CHI可以改变烟草(Nicotiana tabacum)花瓣的着色[8]。除了参与花色的形成,类黄酮合成相关酶基因也参与响应逆境胁迫。低温胁迫下泡核桃(Juglans sigillata)中的CHS显著上调表达[9];芹菜(Apium graveolens)叶柄中的CHI和CHS酶活性在低温下显著升高[10];红花草莓(Fragaria×Potentilla) CHI的表达会随着温度的升高而受到抑制[11];低温下拟南芥(Arabidopsis thaliana)中CHS、CHI和F3H显著上调,诱导了花青素的积累[12]。过表达番茄(Lycopersicon esculentum)的F3H能增强烟草的耐冷性[13]。
竹子是重要的森林资源,具有生长快速的特点,其光合作用、初生增粗和笋的木质化等已开展了广泛研究[14–16]。毛竹(Phyllostachys edulis)是我国栽培面积最大的竹种,已从叶片组织结构、代谢组和转录组方面对其响应低温进行了分析,找到了类黄酮代谢途径的差异表达基因[17–18]。另外,研究表明毛竹中PeCHS可能受不同激素的调控[19]。对七彩红竹(Indosasa hispida ‘Rainbow’)的研究表明, IhCHS1和IhCHI1在幼嫩红秆中显著表达[20–21]。因此,对竹子类黄酮代谢及其酶基因的研究尚不够深入。本研究以毛竹叶片为材料,研究不同生长时期及低温胁迫条件下类黄酮含量的变化趋势,通过生物信息学的方法鉴定毛竹类黄酮早期生物合成的酶基因,明确其分子特征与系统进化关系,并通过qPCR验证关键酶基因的表达模式,以期为深入研究基因的功能提供参考依据。
1 材料和方法 1.1 材料将毛竹(Phyllostachys edulis)种子播种于草炭: 蛭石=3:2的基质中,置于约25 ℃的人工气候室(光16 h/暗8 h,相对湿度约80%)中培养。选择株高和长势一致的3个月大小的毛竹实生苗,一部分用于采集不同生长时期的叶片,包括卷叶(S1)、半卷叶(S2)、展叶(S3)和老叶(S4);另一部分随机分为4组,每组4盆,置于4 ℃培养箱中模拟低温胁迫处理。根据叶片类黄酮的含量变化,选择含量最高时期的叶片作为低温处理材料,分别在低温处理0 (对照)、4和8 h后采集叶片,液氮速冻后置于-80 ℃冰箱中用于后续实验。
1.2 类黄酮含量测定类黄酮的基本结构是A和C环、B和C环2个共轭体系,用芦丁标准品绘制标准曲线。首先制备芦丁标准品母液(1.0 mg/mL),用60%乙醇稀释为0、0.2、0.4、0.6、0.8、1.0 mg/mL共5个浓度梯度, 在510 nm处测定吸光值,以芦丁浓度为横坐标, 吸光值为纵坐标绘制标准曲线。采用亚硝酸钠-硝酸铝比色法提取叶片类黄酮并测定吸光度后,根据标准曲线计算类黄酮含量[22]。类黄酮含量(mg/g FW)= (ΔA+0.0049)×V÷(1.6277×W)×D, 式中, V为提取液体积(1.5 mL);W为样品质量(g);D为稀释倍数, 未稀释即为1。
1.3 基因查找与生物信息学分析利用毛竹最新基因组数据[23],以水稻(Oryza sativa)和拟南芥的3个类黄酮生物合成酶家族(CHS、CHI和F3H)的基因序列为诱饵,利用TBtools v1.092软件[24]内置的BLASN和BLASTX进行同源序列比对,阈值E < 10–20。基于NCBI的CDD在线软件进行保守结构域分析,保留具有完整类黄酮早期生物合成酶基因保守结构域的序列。并对3个类黄酮早期生物合成酶基因家族成员命名。
利用ProtParam (http://web.Expasy.org/protparam/)对类黄酮早期生物合成酶的理化性质进行分析。运用TBtools软件以及毛竹基因组注释文件对毛竹类黄酮早期生物合成酶基因的结构进行分析;使用Plant-mPLoc server在线软件(http://www.csbio.sjtu.edu.cn/bioinf/plant-multi/)进行亚细胞定位的预测[25]。使用PlantCARE (http://bioinformatics.Psb.Ugent.be/webtools/plantcare/html/)对其启动子序列(2 000 bp)中所含调控元件进行分析预测[26]。
为探究毛竹类黄酮合成酶基因的潜在功能及不同物种间的进化关系,从Phytozome V.12数据库(https://phytozome.jgi.doe.gov/)获得水稻、拟南芥、玉米(Zea mays)和高粱(Sorghum bicolor)等物种类黄酮早期生物合成酶的氨基酸序列,利用MEGA 7.0,基于p-distance模型采用邻接法对毛竹和其他物种的序列进行比对分析,生成系统进化树[27],校验参数为1 000次重复,氨基酸替换模型选用No. of differences[28]。
1.4 组织表达模式分析从NCBI Short Read Archive下载毛竹组织(鞭、笋、根、叶片、芽和鞘)的26个转录组数据(登录号:SRX2408703~SRX2408728)[23],筛选其中类黄酮早期合成酶基因的FPKM (fragments per kilobase of exon model per million mapped fragments),并用Log2(FPKM+1)的值作为基因的表达量,用TBtools软件进行表达谱热图绘制,可视化分析各基因的表达情况。
1.5 叶片中的定量表达分析选择关键的毛竹类黄酮早期生物合成酶基因, 根据其基因序列,使用Primer Premier 5.0软件设计特异定量引物(表 1)。用植物RNA提取试剂盒(天漠, TR205-50, 中国)提取RNA,用反转录试剂盒反转录为cDNA (TaKaRa, RR037A, 日本)。qPCR试验在qTOWER2.2系统上进行,体系参照Roche Light Cycler® 480 SYBR Green Ⅰ Master kit试剂盒(Roche, 4707516001, 德国),扩增程序:95 ℃预变性10 min,然后95 ℃变性10 s,60 ℃解链10 s,40个循环。选择PeTIP41作为内参基因[29],相对表达量采用2–ΔΔCT法[30]计算。
| 表 1 实时定量PCR引物 Table 1 Primers used in qPCR |
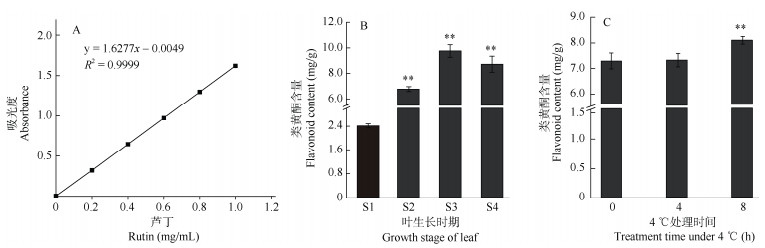
用芦丁标准品建立标准曲线,回归方程为y= 1.6277x-0.0049,相关系数为R2=0.999 9,表明芦丁在一定的范围内具有良好的线性关系(图 1: A)。根据芦丁标准曲线,分别得到不同生长时期和低温胁迫下毛竹叶片的类黄酮含量。在叶片的4个生长时期,类黄酮的含量存在显著差异,随叶片的生长呈先增加后降低的趋势,且在S3时达到最高(图 1: B)。低温处理下毛竹S3叶片的类黄酮含量随胁迫时间的延长而增加,在8 h时显著高于对照,差异达极显著水平(图 1: C),这表明毛竹叶片的生长时期和低温均会影响类黄酮含量。
|
图 1 毛竹叶片的类黄酮含量。A: 芦丁标准曲线; B: 不同生长时期; C: 低温处理; S1: 卷叶期; S2: 半卷叶期; S3: 展叶期; S4: 老叶期; **: P < 0.01。 Fig. 1 Flavonoid content in leaves of Phyllostachys edulis. A: Rutin standard curve; B: Different growth stages; C: Low temperature treatment; S1: Rolled leaf stage; S2: Half-rolled leaf stage; S3: Leaf-spreading stage; S4: Old leaf stage; **: P < 0.01. |
毛竹叶片类黄酮的形成受到其生物合成酶基因表达的影响,因此对毛竹中类黄酮早期生物合成酶基因进行了鉴定。经过在毛竹基因组数据库中比对分析,共获得29个编码具有完整保守结构域的类黄酮早期生物合成酶的基因,分别属于CHS、CHI和F3H家族,分别命名为PeCHS1~PeCHS20、PeCHI1~PeCHI8和PeF3H1。基因结构分析表明,大部分PeCHSs (14个)含有1个内含子,PeCHS10的内含子数量最多(3个),PeCHS4、PeCHS5、PeCHS8、PeCHS14和PeCHS16均含有2个内含子;PeCHIs内含子数量差异很大,有2~13个,其中PeCHI3最多(13个),PeCHI7最少(2个),PeF3H1有2个内含子(图 2)。

|
图 2 PeCHSs、PeCHIs和PeF3H1的基因结构 Fig. 2 Gene structure of PeCHSs, PeCHIs and PeF3H1 |
启动子序列分析结果表明,PeCHSs、PeCHIs和PeF3H1的启动子序列中包含多种非生物胁迫响应元件,如低温的LTR、干旱的MBS和缺氧的ARE等(图 3: A)。PeCHSs和PeCHIs的启动子中均有STRE (逆境响应防御),其中PeCHI4和PeCHI6的启动子最多(9个);多数PeCHSs和PeCHIs启动子中含有低温响应防御元件(MYB-like sequence)、逆境伤害相关(WUN-motif)和真菌诱导(W box)等;4个PeCHIs和PeF3H1的启动子中都含有低温响应元件(LTR);PeCHSs和PeF3H1的启动子中均含有逆境响应防御元件(TC-rich repeats)。此外,在这些序列中还鉴定到其他调控元件,如MYB转录因子的结合元件,与激素相关的响应元件等(图 3: B)。这说明类黄酮早期生物合成基因的转录可能会受到低温及其他各种非生物胁迫的影响。

|
图 3 PeCHSs、PeCHIs和PeF3H1启动子中的顺式作用元件(A)和应答元件(B) Fig. 3 Cis-acting elements (A) and responsive elements (B) in the promoters of PeCHSs, PeCHIs and PeF3H1 |
PeCHSs编码蛋白长度为368~461 aa,分子量为39.94~50.37 kDa,理论等电点为5.54~9.46,亚细胞定位预测在叶绿体和细胞质中。PeCHIs编码蛋白长度为164~400 aa,分子量为18.75~44.92 kDa, 理论等电点为4.83~9.26,预测亚细胞定位均在叶绿体。PeF3H1编码蛋白长度为363 aa,分子量为40.23 kDa,理论等电点为5.38,预测亚细胞定位在细胞质(表 2)。基因结构和蛋白理化性质的差异可能是导致基因功能不同的主要原因。
| 表 2 PeCHSs、PeCHIs和PeF3H1的基本特征 Table 2 Basic characteristics of PeCHSs, PeCHIs and PeF3H1 |
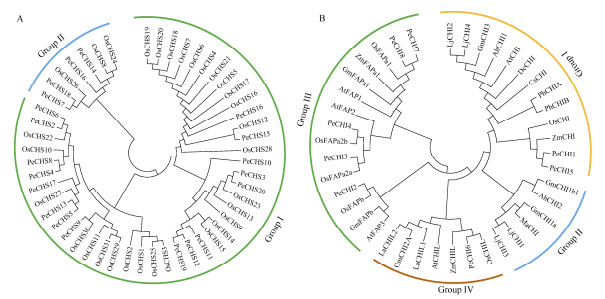
根据水稻中类黄酮早期生物合成CHS的分类构建,毛竹20个PeCHSs聚类在2个组,分别含有2个和18个成员,其中分支Ⅰ中PeCHS14和PeCHS16分别与已知功能的OsCHS24和OsCHS8高度同源,推测它们可能具有合成查尔酮的功能(图 4: A);分支Ⅱ中PeCHSs与OsCHSs各成员存在不同程度的同源性,但功能尚不确定。通过构建毛竹与其他物种CHI的进化分类,PeCHIs在Group Ⅰ、Group Ⅲ和Group Ⅳ 3个组中(图 4: B),PeCHI1和PeCHI5与ZmCHI聚类在Group Ⅰ分支,推测它们具有合成柚皮素的功能。在F3H的进化树中,毛竹PeF3H1与水稻OsF3H同源性最高,推测它们可能具有相似的功能。
|
图 4 基于不同种植物的CHS (A)和CHI (B)家族成员构建的系统发育树。Pe: 毛竹; Os: 水稻; Zm: 玉米; At: 拟南芥; Gm: 大豆; La: 羽扇豆; Lj: 百脉根; Ma: 紫苜蓿; Ah: 落花生; Ph: 矮牵牛; Dc: 康乃馨; Cs: 茶树。 Fig. 4 Phylogenetic tree based on CHS (A) and CHI (B) family in different species. Pe: Phyllostachys edulis; Os: Oryza sativa; Zm: Zea mays; At: Arabidopsis thaliana; Gm: Glycine max; La: Lupinus angustifolius; Lj: Lotus japonicus; Ma: Medicago sativa; Ah: Arachis hypogaea; Ph: Petunia hybrida; Dc: Dianthus caryophyllus; Cs: Camellia sinensis. |
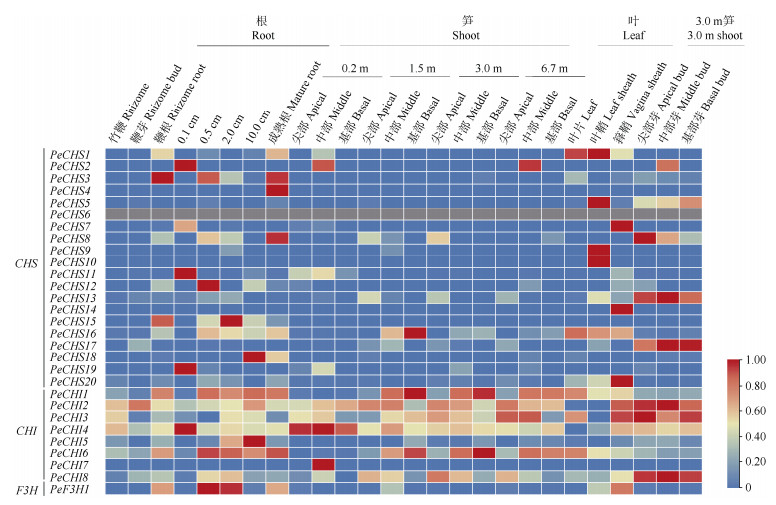
组织表达分析在一定程度上能够验证基因的功能,根据毛竹26个组织的转录组数据分析了类黄酮早期合成酶基因的转录水平。3个基因家族的各个成员在同一组织的不同生长时期以及不同组织中的表达模式存在差异(图 5)。PeCHSs在毛竹的根、笋和叶的部分组织中能检测到表达,更倾向在根和叶中表达,如PeCHS9、PeCHS10、PeCHS14和PeCHS20仅在叶中高表达,PeCHS3、PeCHS4、PeCHS15和PeCHS18仅在根中高表达;大部分PeCHSs基因在笋中(除PeCHS2和PeCHS16外)的表达水平较低;而PeCHS6在26个组织中均没有检测到。PeCHIs (除PeCHI5和PeCHI7外)在各个组织中均有表达,它们可能与毛竹整个生长发育过程密切相关。PeF3H1倾向在根和箨片中表达。
|
图 5 PeCHSs、PeCHIs和PeF3H1在毛竹26个组织中的表达模式 Fig. 5 Expression pattern of PeCHSs, PeCHIs and PeF3H1 in twenty-six tissues of Phyllostachys edulis |
植物在不同生长时期和低温胁迫下的类黄酮含量会发生显著变化,以适应外界环境。根据系统进化、启动子元件和转录组数据分析,筛选获得6个代表基因(PeCHS1、PeCHS13、PeCHS16、PeCHI1、PeCHI6和PeF3H1)进行qPCR分析。结果表明,6个基因随着叶片的生长表达量变化差异较大,其中PeCHS1呈现先升高后降低的趋势,与类黄酮含量的趋势相同,在S3达到最高峰,表达量是S1的687.7倍;PeCHS13、PeCHI1和PeF3H1的表达量显著下调;PeCHS16呈现先下调后上调的表达趋势,在S4显著升高,是S1的41.58倍;而PeCHI6的表达则呈现波动变化的趋势,在S2和S3变化不显著,在S4时,其表达量显著上调,是S1的4.57倍(图 6: A)。经过低温胁迫后,这6个基因均呈现逐渐上升的表达趋势,且均在8 h达到峰值, 与类黄酮含量表达趋势一致(图 6: B)。因此,推测他们在毛竹叶片中的类黄酮合成和抵御低温胁迫中发挥着关键作用。

|
图 6 PeCHSs、PeCHIs和PeF3H1在不同时期(A)和低温处理(B)叶片中的相对表达量。*: P < 0.05; **: P < 0.01。 Fig. 6 Relative expression of PeCHSs, PeCHIs and PeF3H1 in leaves at different growth stages (A) and under low temperature (B). *: P < 0.05; **: P < 0.01. |
类黄酮化合物广泛存在于植物体内,是一类重要的次生代谢产物,在植物遭受低温、干旱等非生物胁迫时,类黄酮合成的相关基因被诱导表达,通过清除活性氧在植物的生长发育和逆境胁迫中发挥重要功能。毛竹广泛分布于我国长江流域及南方各省区,具有重要的经济价值,但是温度制约了毛竹生长和地理分布,尤其是冬季低温和冰冻雨雪灾害的影响尤为严重。因此,提高毛竹抵御低温胁迫的能力是其育种的重要目标之一。本研究通过检测毛竹叶片中类黄酮含量,包括不同生长时期和4 ℃低温处理下的功能叶片,发现类黄酮含量发生了明显变化,表明在毛竹生长发育和低温条件下类黄酮可能发挥着重要作用。通过分析类黄酮早期生物合成途径中涉及的酶基因家族成员发现,毛竹CHS家族成员与其它物种相似,不同分支的家族成员可能具有不同的功能。虽然PeCHS13和OsCHS27聚类在一起,但水稻中OsCHS27缺乏2个保守的氨基酸而不具有催化合成类黄酮的功能[31],因此PeCHSs的具体功能需要进一步研究。PeCHIs家族的成员多于水稻和拟南芥,这与该家族基因发生过复制和扩张有关[23]。Group Ⅰ中PeCHI1和PeCHI5与玉米和水稻中具有合成类黄酮功能的CHI在同一组;Group Ⅲ中的CHI属于脂肪酸结合蛋白(FAPs), PeCHI2、PeCHI3、PeCHI4、PeCHI7和PeCHI8可能影响毛竹细胞中脂肪酸的生物合成及储存[32]; Group Ⅳ作为类黄酮产生的增强剂,PeCHI6可能间接促进类黄酮的生物合成[33]。F3H家族在毛竹中只有1个基因,这与F3H以单拷贝形式存在,在进化上保守[34]一致。
研究表明,类黄酮生物合成酶基因的表达具有发育时期特异性。在大豆中,GmCHS1倾向于在绿叶中表达,而GmCHS2倾向于在子叶中表达[35]。葡萄的CHI在幼叶、幼根、果实和种子中均有表达[36]。银杏(Ginkgo biloba)叶片幼叶时期,虽然分生组织活跃,类黄酮合成快,但由于叶片自身的合成能力有限,因此类黄酮的含量较低;在展叶时期,叶片自身的合成能力强,类黄酮含量较高[37]。在毛竹卷叶中的类黄酮含量最低,功能叶片中的最高,不同时期叶片中仅PeCHS1的表达与类黄酮含量一致,其它类黄酮早期生物合成基因的表达变化差异明显但没有规律,PeCHS13和PeCHI1在卷叶时期表达量最高,PeCHS16和PeCHI6在老叶时期表达量最高,这可能与叶片处于不同生长时期以及类黄酮生物合成途径存在分支有关。
当受到低温胁迫时,植物体内通过上调表达类黄酮合成酶基因促进其积累来增强植物对低温的耐受性。对低温处理的桃树(Prunus persica)叶片进行转录组分析表明,类黄酮合成相关基因显著上调[38]; 低温促进烟草叶片和苦荞麦(Fagopyrum tataricum)中类黄酮相关基因上调表达,从而提高类黄酮的含量[39–40]。本研究对低温处理毛竹叶中类黄酮含量和基因表达的分析也获得了类似的结果,类黄酮含量及其合成酶基因(3个PeCHSs、2个PeCHIs和PeF3H1)的表达随着低温处理时间的延长而逐渐增加,且在8 h时达到峰值。然而,除PeCHS1外, 其他类黄酮早期生物合成的关键酶基因在不同生长阶段叶片中的表达与类黄酮含量变化并不一致,其原因可能与叶片生长发育的复杂调控有关,有待于进一步研究。
本研究在毛竹中共鉴定到3个酶基因家族的29个成员,分别为20个查尔酮合酶基因(PeCHSs)、8个查尔酮异构酶基因(PeCHIs)和1个黄酮-3-烃化酶基因(PeF3H1)。在基因结构、进化分析、理化性质、顺式作用元件和转录组数据等生物学分析的基础上,通过低温下叶片中类黄酮含量的变化和不同基因的表达差异,初步确定了PeCHS1、PeCHS13、PeCHS16、PeCHI1、PeCHI6和PeF3H1可能是毛竹类黄酮早期生物合成的关键酶基因,为进一步解析类黄酮早期合成关键酶基因的功能和抗逆基因资源挖掘提供了参考依据。
| [1] |
DING Y H, WANG M, XIE J, et al. Response to low temperature stress in plants and advances in research methods[J]. Jiangsu Agric Sci, 2019, 47(14): 31-36. 丁红映, 王明, 谢洁, 等. 植物低温胁迫响应及研究方法进展[J]. 江苏农业科学, 2019, 47(14): 31-36. DOI:10.15889/j.issn.1002-1302.2019.14.007 |
| [2] |
LI P, MA L, XU F, et al. Comparison of morphological and physiological responses of two different species of northern winter rapeseed before overwintering[J]. Agric Res Arid Areas, 2022, 40(5): 42-51. 李鹏, 马骊, 徐芳, 等. 越冬前北方不同类型强冬性冬油菜形态及生理响应研究[J]. 干旱地区农业研究, 2022, 40(5): 42-51. DOI:10.7606/j.issn.1000-7601.2022.05.05 |
| [3] |
WATANABE M, AYUGASE J. Effect of low temperature on flavornoids, oxygen radical absorbance capacity values and major components of winter sweet spinach (Spinacia oleracea L[J]. J Sci Food Agric, 2015, 95(10): 2095-2104. DOI:10.1002/jsfa.6925 |
| [4] |
YAN X. The effects of environmental stresses on the compont and gene expression of flovoinds in Trifoliate orange [D]. Wuhan: Huazhong Agricultural University, 2011. 晏校. 逆境胁迫对枳实生苗类黄酮组分含量及关键酶基因表达量的影响[D]. 武汉: 华中农业大学, 2011. |
| [5] |
XU W J, GRAIN D, BOBET S, et al. Complexity and robustness of the flavonoid transcriptional regulatory network revealed by comprehensive analyses of MYB-bHLH-WDR complexes and their targets in Arabidopsis seed[J]. New Phytol, 2014, 202(1): 132-144. DOI:10.1111/nph.12620 |
| [6] |
LI M, CAO Y T, YE S R, et al. Isolation of CHS gene from Brunfelsia acuminata flowers and its regulation in anthocyanin biosynthesis[J]. Molecules, 2016, 22(1): 44. DOI:10.3390/molecules22010044 |
| [7] |
HELARIUTTA Y, ELOMAA P, KOTILAINEN M, et al. Chalcone synthase-like genes active during corolla development are differentially expressed and encode enzymes with different catalytic properties in Gerbera hybrida (Asteraceae)[J]. Plant Mol Biol, 1995, 28(1): 47-60. DOI:10.1007/BF00042037 |
| [8] |
ZHOU L, WANG Y, REN L, et al. Overexpression of Ps-CHI1, a homologue of the chalcone isomerase gene from tree peony (Paeonia suffruticosa), reduces the intensity of flower pigmentation in transgenic tobacco[J]. Plant Cell Tiss Organ Cult, 2014, 116(3): 285-295. DOI:10.1007/s11240-013-0403-2 |
| [9] |
WANG Y, XIAO L Y, MA T, et al. Cloning and functional analysis of chalcone synthase gene in cold-induced Juglans sigillata[J]. Mol Plant Breed, 2018, 16(2): 386-391. 王毅, 肖良俊, 马婷, 等. 低温诱导泡核桃中查尔酮合成酶基因克隆及功能分析[J]. 分子植物育种, 2018, 16(2): 386-391. DOI:10.13271/j.mpb.016.000386 |
| [10] |
ZHANG M. Effects of temperature, drought and UV-B irradiation time on celery growth and flavonoids accumulation [D]. Nanjing: Nanjing Agricultural University, 2017. 张萌. 温度、干旱和UV-B照射时间对芹菜生长及黄酮类物质积累的影响[D]. 南京: 南京农业大学, 2017. |
| [11] |
LIANG J C. Response of flower color to temperature and analysis of flower color related gene expression in red-flowered strawberry [D]. Shenyang: Shenyang Agricultural University, 2018. 梁吉昌. 红花草莓花色对温度的响应及花色相关基因表达分析[D]. 沈阳: 沈阳农业大学, 2018. |
| [12] |
ZHANG Y Q, ZHENG S, LIU Z J, et al. Both HY5 and HYH are necessary regulators for low temperature-induced anthocyanin accumulation in Arabidopsis seedlings[J]. J Plant Physiol, 2011, 168(4): 367-374. DOI:10.1016/j.jplph.2010.07.025 |
| [13] |
JIA H F, JIU S, ZHANG C, et al. Abscisic acid and sucrose regulate tomato and strawberry fruit ripening through the abscisic acid-stressripening transcription factor[J]. Plant Biotechnol J, 2016, 14(10): 2045-2065. DOI:10.1111/pbi.12563 |
| [14] |
ZHAO H S, LOU Y F, SUN H Y, et al. Transcriptome and comparative gene expression analysis of Phyllostachys edulis in response to high light[J]. BMC Plant Biol, 2016, 16: 34. DOI:10.1186/s12870-016-0720-9 |
| [15] |
WEI Q, JIAO C, GUO L, et al. Exploring key cellular processes and candidate genes regulating the primary thickening growth of moso underground shoots[J]. New Phytol, 2017, 214(1): 81-96. DOI:10.1111/nph.14284 |
| [16] |
YANG K B, LI L C, LOU Y F, et al. A regulatory network driving shoot lignification in rapidly growing bamboo[J]. Plant Physiol, 2021, 187(2): 900-916. DOI:10.1093/plphys/kiab289 |
| [17] |
WANG H Y, GUO L, ZHA R F, et al. Histological, metabolomic, and transcriptomic analyses reveal mechanisms of cold acclimation of moso bamboo (Phyllostachys edulis) leaf[J]. Tree Physiol, 2022, 20: tpac064. DOI:10.1093/treephys/tpac064 |
| [18] |
HUANG Z, ZHU P L, ZHONG X J, et al. Transcriptome analysis of Moso bamboo (Phyllostachys edulis) reveals candidate genes involved in response to dehydration and cold stresses[J]. Front Plant Sci, 2022, 13: 960302. DOI:10.3389/fpls.2022.960302 |
| [19] |
ZHANG N, BAO H, CUI B L, et al. Genomic analysis of chalcone synthase gene family of Phyllostachys edulis[J]. Mol Plant Breed, 2022, 20(3): 817-825. 章妮, 暴涵, 崔博亮, 等. 毛竹查尔酮合成酶基因家族全基因组分析[J]. 分子植物育种, 2022, 20(3): 817-825. DOI:10.13271/j.mpb.020.000817 |
| [20] |
ZHOU X, WANG C C, BI W, et al. Cloning and sequence analysis of chalcone synthase gene IhCHS1 from Indosasa hispida cv. Rainbow[J]. J Bamboo Res, 2014, 33(3): 11-16. 周旭, 王晨晨, 毕玮, 等. 七彩红竹查尔酮合成酶IhCHS1基因的克隆与分析[J]. 竹子研究汇刊, 2014, 33(3): 11-16. DOI:10.3969/j.issn.1000-6567.2014.03.002 |
| [21] |
WANG C C, BI W, WANG J, et al. Cloning and expression analysis of IhCHI1 from Indosasa hispida cv. Rainbow[J]. J W China For Sci, 2014, 43(2): 85-90. 王晨晨, 毕玮, 王娟, 等. 七彩红竹查尔酮异构酶基因IhCHI1的克隆与表达分析[J]. 西部林业科学, 2014, 43(2): 85-90. DOI:10.16473/j.cnki.xblykx1972.2014.02.018 |
| [22] |
XIA G H, LI X H, WANG X Y. Effect of different processing on the antioxidant activity of flavonoids from Polygonatum odoratum[J]. Food Sci Technol, 2018, 43(3): 181-185. 夏光辉, 李新华, 王晓雅. 不同加工处理对玉竹黄酮抗氧化活性的影响研究[J]. 食品科技, 2018, 43(3): 181-185. DOI:10.13684/j.cnki.spkj.2018.03.034 |
| [23] |
PENG Z H, LU Y, LI L B, et al. The draft genome of the fast-growing non-timber forest species moso bamboo (Phyllostachys heterocycla)[J]. Nat Genet, 2013, 45(4): 456-461. DOI:10.1038/ng.2569 |
| [24] |
CHEN C J, CHEN H, ZHANG Y, et al. TBtools: An integrative toolkit developed for interactive analyses of big biological data[J]. Mol Plant, 2020, 13(8): 1194-1202. DOI:10.1016/j.molp.2020.06.009 |
| [25] |
CHOU K C, SHEN H B. Cell-PLoc 2.0: An improved package of webservers for predicting subcellular localization of proteins in various organisms[J]. Nat Sci, 2010, 2(10): 1090-1103. DOI:10.4236/ns.2010.210136 |
| [26] |
LESCOT M, DÉHAIS P, THIJS G, et al. PlantCARE, a database of plant cis-acting regulatory elements and a portal to tools for in silico analysis of promoter sequences[J]. Nucl Acids Res, 2002, 30(1): 325-327. DOI:10.1093/nar/30.1.325 |
| [27] |
KUMAR S, STECHER G, TAMURA K. MEGA7: Molecular evolutionary genetics analysis version 7.0 for bigger datasets[J]. Mol Biol Evol, 2016, 33(7): 1870-1874. DOI:10.1093/molbev/msw054 |
| [28] |
JONES D T, TAYLOR W R, THORNTON J M. The rapid generation of mutation data matrices from protein sequences[J]. Comput Appl Biosci, 1992, 8(3): 275-282. DOI:10.1093/bioinformatics/8.3.275 |
| [29] |
FAN C J, MA J M, GUO Q R, et al. Selection of reference genes for quantitative real-time PCR in bamboo (Phyllostachys edulis)[J]. PLoS One, 2013, 8(2): e56573. DOI:10.1371/journal.pone.0056573 |
| [30] |
LIVAK K J, SCHMITTGEN T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2−ΔΔCT Method[J]. Methods, 2001, 25(4): 402-408. DOI:10.1006/meth.2001.1262 |
| [31] |
PARK H L, YOO Y, BHOO S H, et al. Two chalcone synthase isozymes participate redundantly in UV-induced sakuranetin synthesis in rice[J]. Int J Mol Sci, 2020, 21(11): 3777. DOI:10.3390/ijms21113777 |
| [32] |
NGAKI M N, LOUIE G V, PHILIPPE R N, et al. Evolution of the chalcone-isomerase fold from fatty-acid binding to stereospecific catalysis[J]. Nature, 2012, 485(7399): 530-533. DOI:10.1038/nature11009 |
| [33] |
MORITA Y, TAKAGI K, FUKUCHI-MIZUTANI M, et al. A chalcone isomerase-like protein enhances flavonoid production and flower pigmentation[J]. Plant J, 2014, 78(2): 294-304. DOI:10.1111/tpj.12469 |
| [34] |
PELLETIER M K, SHIRLEY B W. Analysis of flavanone 3-hydro-xylase in Arabidopsis seedlings: Coordinate regulation with chalcone synthase and chalcone isomerase[J]. Plant Physiol, 1996, 111(1): 339-345. DOI:10.1104/pp.111.1.339 |
| [35] |
ANGURAJ VADIVEL A K, KRYSIAK K, TIAN G, et al. Genome-wide identification and localization of chalcone synthase family in soybean (Glycine max (L.) Merr)[J]. BMC Plant Biol, 2018, 18(1): 325. DOI:10.1186/s12870-018-1569-x |
| [36] |
ZHOU J, YAO Q H, PENG R H, et al. Cloning and expression analysis of CHI of Kyoho grape by semi-quantity RT-PCR[J]. Acta Bot Boreali-Occid Sin, 2009, 29(9): 1723-1729. 周军, 姚泉洪, 彭日荷, 等. 巨峰葡萄查尔酮异构酶基因克隆及表达分析[J]. 西北植物学报, 2009, 29(9): 1723-1729. DOI:10.3321/j.issn:1000-4025.2009.09.001 |
| [37] |
VOGT T, GUL P G. Accumulation of flavonoids during leaf development in Cistus laurifolius[J]. Phytochemistry, 1994, 36(3): 591-597. DOI:10.1016/S0031-9422(00)89780-0 |
| [38] |
ZHOU P, LIN Z K, GUO R, et al. Effects of low temperature treatment on gene expression and flavonoids biosynthesis metabolism in peach (Prunus persica) leaves[J]. J Agric Biotechnol, 2021, 29(7): 1283-1294. 周平, 林志楷, 郭瑞, 等. 低温处理对桃树叶片基因表达及类黄酮合成代谢的影响[J]. 农业生物技术学报, 2021, 29(7): 1283-1294. DOI:10.3969/j.issn.1674-7968.2021.07.005 |
| [39] |
ZHANG G, PANG T, WANG S S, et al. Regulation of different growth temperatures on flavonoids content and their metabolic pathways in tobacco leaves[J]. Genom Appl Biol, 2016, 35(12): 3527-3537. 张刚, 逄涛, 王莎莎, 等. 不同生长温度对烟草叶片黄酮类化合物含量及其代谢途径的调控[J]. 基因组学与应用生物学, 2016, 35(12): 3527-3537. DOI:10.13417/j.gab.035.003527 |
| [40] |
SONG Y J, FENG J C, LIU D M, et al. Different phenylalanine pathway responses to cold stress based on metabolomics and transcripttomics in tartary buckwheat landraces[J]. J Agric Food Chem, 2022, 70(2): 687-698. DOI:10.1021/acs.jafc.1c06915 |
 2024, Vol. 32
2024, Vol. 32



