2. 东北林业大学生命科学学院, 哈尔滨 150040
2. College of Life Science, Northeast Forestry University, Harbin 150040, China
扩展蛋白(expansin, EXP)是植物中重要的蛋白之一,最早在黄瓜(Cucumis sativus)的根尖细胞壁中被发现[1],是一类能够使植物细胞壁松弛的活性蛋白,在植物生长发育和逆境抗性过程中发挥着重要的作用[2]。在拟南芥(Arabidopsis thaliana)和毛果杨(Populus trichocarpa)中各有36个EXP成员[3–4],葡萄(Vitis vinifera)中有29个[5],水稻(Oryza sativa)中有58个[3]。根据结构的差异,扩展蛋白可分为4个亚家族,分别为扩展蛋白A、B亚家族(EXPA、EXPB)和类扩展蛋白A、B亚家族(EXLA、EXLB)[6]。其中EXPA主要参与植物的根毛发生与伸长[7]、种子萌发[8–9]、果实软化[10–11]、木质部的形成[12]、纤维发育[13]、茎和节间的伸长[14]、叶片发育[15]等重要发育过程,同时也参与响应植物非生物胁迫的过程, 如耐盐性[16]、抗旱性[17]、抗重金属[18]等;EXPB则主要参与花粉管伸长[19–22]等。
桉树(Eucalyptus)是世界三大速生树种之一,具有生长速度快、轮伐周期短、适应性强以及木材质量较好等优点,广泛应用于制浆造纸、木材加工、医疗和香料等产业,因此具有十分重要的经济价值以及生态价值[23]。其大量优良基因的功能尚待挖掘,巨桉(E. grandis)基因组测序的完成为开展相关研究提供了基础[24],目前在巨桉中共发现35个EXP家族成员[25],其中EXPA亚家族成员24个,但其在桉树生长发育或者响应胁迫中的具体功能尚未见报道。
前期对桉树不同节间进行转录组测序,在初生生长到次生生长的转换过程中EgrEXPA8和EgrEXPA10存在差异表达,尤其是在次生生长阶段表达量显著下降,说明其可能参与到桉树的次生生长或者木质部发育的负调控。本研究以巨桉无性系GL1为材料, 克隆了2个扩展蛋白基因EgrEXPA8和EgrEXPA10,对其基因结构及编码的蛋白结构和理化性质等进行了分析,采用qRT-PCR技术分析它们的组织表达模式及对不同胁迫的响应,并初步对EgrEXPA8和EgrEXPA10基因功能进行分析,为其功能挖掘和分子调控机制研究提供参考依据。
1 材料和方法 1.1 材料使用巨桉(Eucalyptus grandis)无性系GL1组培苗为材料,选取生长健壮的生根苗移植到中国林业科学研究院热带林业研究所温室培养,6个月后苗木生长至约1.5 m,分别取根、茎尖、木质部、韧皮部、幼叶、成熟叶,同时选取从顶端起第1、3、5、7、9、11节间作为初生生长到次生生长转换的材料。
生根的组培苗移植至温室后,经改良霍格兰营养液水培生长至约20 cm高,选取生长健壮、根系良好的水培苗,分别用0.1 mmol/L茉莉酸甲酯(MeJA)、0.1 mmol/L水杨酸(SA)进行喷施、200 mmol/L NaCl溶液灌根,并在处理后0、1、6、24和168 h收集叶片。将水培苗转移至去除磷(磷酸二氢钾)或硼元素(硼酸)的改良霍格兰营养液中培养,分别在6、24、48、96和21 d后收集叶片。每处理5株苗,每处理3个重复,取样统一采集顶端向下第4~6片叶片。所有样品采集后迅速置于液氮中速冻,随后立即转入超低温冰箱-80 ℃保存,用于RNA提取。
1.2 菌株与质粒克隆载体pEASY-T1 Simple Cloning Vector购于全式金生物技术(北京)有限公司,DH5α大肠杆菌感受态细胞购自擎科生物科技(北京)有限公司,植物RNA提取试剂盒购于艾德莱生物科技(北京)有限公司,DNA回收试剂盒购于天根生化科技(北京)有限公司,反转录试剂购于美国英杰生命技术有限公司(Invitrogen),荧光定量PCR试剂TB Green® Premix Ex TaqTM购于大连宝日医生物技术有限公司(TaKaRa)。
1.3 RNA的提取与cDNA的合成按照Aidlab EASYspin植物RNA快速提取试剂盒的说明书进行RNA提取,然后经过DNase Ⅰ消化基因组DNA,得到纯化的总RNA。采用琼脂糖凝胶电泳以及NanoDrop one (购于美国赛默飞世尔科技公司)检测RNA质量和浓度,分别取1 μg检测合格的总RNA,采用SuperscriptⅢ反转录试剂盒进行cDNA合成。
1.4 基因克隆与测序从Phytozome 13 (https://phytozome-next.jgi.doe.gov/)数据库中分别下载EgrEXPA8和EgrEXPA10基因序列。利用Primer 5.0软件设计巨桉EgrEXPA8和EgrEXPA10基因的特异性克隆引物(表 1),由擎科生物科技(北京)有限公司合成。以稀释10倍的cDNA为模板,采用Q5 High-Fidelity PCR Kit (NEB)进行PCR反应,采用20 μL反应体系,反应程序为:98 ℃预变性30 s;98 ℃变性10 s,68 ℃/70 ℃ (EgrEXPA8/EgrEXPA10)退火20 s,72 ℃延伸45 s,35个循环;72 ℃延伸2 min。利用1.5%的琼脂糖凝胶电泳检测PCR产物,并使用天根(北京)胶回收试剂盒回收位置正确且条带明亮的产物,随后连接到pEASY®-T1CloningKit载体上并转入DH5α感受态细胞,在抗性平板上挑取平滑的菌落摇菌进行菌液PCR验证,选取正确的样本菌液送睿博兴科生物技术(北京)有限公司进行测序。
| 表 1 引物序列 Table 1 Primer sequence |
利用ProtParam (https://web.expasy.org/protparam/)在线分析氨基酸序列的理化性质,包括理论等电点、氨基酸长度以及蛋白分子量等;利用ProtScale (https://web.expasy.org/protscale/)在线工具分析蛋白质的疏水性和亲水性;利用SOPMA (https://npsaprabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=/NPSA/npsa_sopma.html)和SWISS-MODEL (https://swissmodel.expasy.org/interactive)在线工具进行蛋白质二级结构和三级结构分析;利用NCBI的Conserved domains database (CDD)数据库(https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)对蛋白进行结构域预测;在拟南芥信息数据库(https://www.Arabidopsis.org/index.jsp)中检索并下载了所有EXPA蛋白的氨基酸序列,利用Clustal程序对桉树扩展蛋白EgrEXPA8、EgrEXPA10与拟南芥扩展蛋白EXPA亚家族所有成员进行多序列比对分析,使用MEGA 6.0软件,采用邻接法(neighbor-joining method, NJ)构建系统进化树;同时利用NCBI BlASTP (https://blast.ncbi.nlm.nih.gov/Blast.cgi)在线查找在拟南芥、杨树、水稻、葡萄、大豆(Glycine max)和黄瓜中的同源序列,并通过PFAM和SMART对不同植物的同源序列进行验证,下载各成员的蛋白序列,并利用DNAMAN软件进行蛋白质同源序列分析,利用MEGA 6.0内置的Clustal W程序进行多重序列比对,并采用邻接法构建系统进化树。
1.6 EgrEXPA8和EgrEXPA10的差异表达分析将巨桉RNA样品进行反转录,以稀释10倍的cDNA样品为模板,根据克隆的EgrEXPA8和EgrEXPA10测序结果,利用Primer Premier 5.0软件分别设计特异性定量引物,由北京擎科生物科技有限公司合成(表 1),以EgrEF2为内参基因。使用Roche公司的Light Cycler 96荧光定量分析仪以及大连宝日医生物技术有限公司(TaKaRa)的SYBR Premix Ex Taq Ⅱ试剂盒。实时定量PCR的反应体系为20 μL,包括1 μL cDNA、10 μL SYBR Green Ⅰ Master、10 μmol/L的正反向引物各0.8和7.4 μL,用ddH2O补足。反应程序为95 ℃ 30 s,95 ℃ 5 s,60 ℃ 34 s, 共40个循环。溶解曲线分析程序采用95 ℃ 15 s,60 ℃ 1 min,95 ℃ 15 s。每个样品3次生物学重复以及3次技术重复,根据2–ΔΔCT法[26]计算目的基因的相对表达量。
1.7 数据统计分析采用SPSS22分析软件对数据进行单因素方差分析(One-Way ANOVA)和Duncan多重比较(P=0.05), 然后利用GraphPad Prism 8进行制图。
2 结果和分析 2.1 EgrEXPA8和EgrEXPA10基因克隆提取巨桉叶片的RNA,反转录为cDNA,以此为模板,根据扩展蛋白基因Eucgr.J00120.1和Eucgr. I01954.1序列设计引物(表 1),进行PCR扩增,克隆目的基因。扩增产物经电泳检测,2个扩展蛋白片段长度均约为750 bp (图 1)。回收目的片段,连接到T载体上并进行测序,其长度分别为750和735 bp,编码249和244个氨基酸,命名为EgrEXPA8 (Eucgr.J00120.1)和EgrEXPA10 (Eucgr.I01954.1)。
|
图 1 巨桉EgrEXPA8和EgrEXPA10基因的PCR扩增电泳图。M: DL 2000 DNA Marker; 1: EgrEXPA10; 2: EgrEXPA8。 Fig. 1 PCR amplification of EgrEXPA8 and EgrEXPA10 gene in Eucalyptus grandis. M: DL 2000 DNA Marker; 1: EgrEXPA10; 2: EgrEXPA8. |
采用ProtParam对EgrEXPA8和EgrEXPA10蛋白理化性质进行分析。EgrEXPA8蛋白的理论分子量为26.52 kD,等电点(pI)为7.53,脂溶性指数为61.20。
EgrEXPA10蛋白的理论分子量与EgrEXPA8近似,为26.29 kD,等电点为9.1,脂溶性指数为69.10。此外,EgrEXPA8蛋白不稳定性指数为31.20,而EgrEXPA10达40.32,表明其稳定性低于EgrEXPA8。这也表明这2个蛋白可能具有不同的功能。
ProtScale预测巨桉EgrEXPA8和EgrEXPA10蛋白为亲水性蛋白。SOPMA和SWISS-MODEL工具同源建模法预测表明,EgrEXPA8和EgrEXPA10蛋白的二级和三级结构主要以延伸链和无规卷曲为主(图 2)。结构域预测表明EgrEXPA8和EgrEXPA10蛋白均含有2个保守结构域DPBB_1和Pollen_allerg_1。

|
图 2 巨桉EgrEXPA8 (A, C)和EgrEXPA10 (B, D)的二级(A, B)和三级(C, D)结构模型 Fig. 2 Secondary (A, B) and tertiary (C, D) structures model of EgrEXPA8 (A, C) and EgrEXPA10 (B, D) of Eucalyptus grandis |
根据Sampedro等[27]的分类方法,27个EXPA蛋白可分为10个类群,EgrEXPA10属于第I类群,与拟南芥的AtEXPA5亲缘关系最近,EgrEXPA8属于第Ⅲ类群,与拟南芥的AtEXPA8亲缘关系最近(图 3: A)。同源序列分析表明,EgrEXPA8与其他植物扩展蛋白的同源性为73.83%~82.81%,其中与葡萄VvEXPA8的同源性最高。从构建的进化树可见,巨桉EgrEXPA8与葡萄、大豆、杨树、拟南芥聚在同一支上(图 3: B),亲缘关系较近。EgrEXPA10与其他植物扩展蛋白的同源性为67.43%~82.38%,与葡萄VvEXPA10的同源性最高,巨桉EgrEXPA10与黄瓜CsEXPA5的亲缘关系最近,且与葡萄、大豆、杨树、拟南芥聚在同一支上(图 3: C)。

|
图 3 系统进化树。A: EgrEXPA8、EgrEXPA10和拟南芥α-expansins; B: EgrEXPA8与其他植物α-expansins; C: EgrEXPA10与其他植物α-expansins; Egr: 巨桉; Pt: 毛果杨; Gm: 大豆; Vv: 葡萄; At: 拟南芥; Os: 水稻; Cs: 黄瓜。 Fig. 3 Phylogenetic tree. A: EgrEXPA8 and EgrEXPA10 with α-expansins in Arabidopsis thaliana; B: EgrEXPA8 and α-expansins in other plants; C: EgrEXPA10 and α-expansins in other plants. Egr: Eucalyptus grandis; Pt: Populus trichocarpa; Vv: Vitis vinifera; At: Arabidopsis thaliana; Os: Oryza sativa; Cs: Cucumis sativus. |
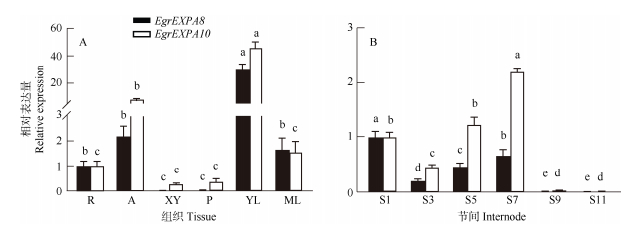
利用实时荧光定量PCR技术检测EgrEXPA8和EgrEXPA10在不同组织和不同节间的表达模式。结果表明,EgrEXPA8和EgrEXPA10在根、茎尖、木质部、韧皮部、幼叶、成熟叶中呈现相似的表达特征,均在幼叶和茎尖表达量最高,而在木质部和韧皮部中相对较低(图 4: A)。EgrEXPA8和EgrEXPA10均在茎的9和11节间的表达量显著降低,说明EgrEXPA8和EgrEXPA10可能参与桉树初生生长阶段细胞壁的维持,也可能参与桉树负调控次生生长。同时,EgrEXPA8和EgrEXPA10在茎节间的表达略微不同, 随着节间的次生发育越来越成熟, EgrEXPA8均被抑制表达,EgrEXPA10在中间节间上调表达,而在未木质化或者木质化程度较高的两端节间被抑制表达,其中在茎的节间7表达量最高(图 4: B)。
|
图 4 巨桉EgrEXPA8和EgrEXPA10的表达模式。R: 根; A: 茎尖; XY: 木质部; P: 韧皮部; YL: 幼叶; ML: 成熟叶。 Fig. 4 Expression patterns of EgrEXPA8 and EgrEXPA10 in Eucalyptus grandis. R: Root; A: Apical; XY: Xylem; P: Phloem; YL: Young leaf; ML: Mature leaf. |
EgrEXPA8能快速响应盐胁迫,盐处理1 h的表达量急剧下降,随处理时间的延长,表达量继续下降。EgrEXPA10的表达量在盐处理后也呈下降的趋势,与EgrEXPA8不同的是,其在处理6 h才出现显著降低,处理168 h的表达量呈上升趋势(图 5: A)。EgrEXP8和EgrEXPA10的表达对茉莉酸甲酯表现出相似的响应方式,均被抑制表达(图 5: B)。在水杨酸处理下,EgrEXPA8和EgrEXPA10的表达呈现出不同的趋势,EgrEXPA8的表达量迅速显著提高, 且长期喷施处理仍保持着较高水平, EgrEXPA10只在处理168 h时比对照显著提高(图 5: C)。长期缺硼条件下, EgrEXPA8和EgrEXPA10的表达量均显著提高,均在处理504 h后达最高,EgrEXPA8在处理48 h内均被抑制表达,而EgrEXPA10在处理24 h后逐渐上调表达(图 6: A)。缺磷条件下,EgrEXPA8的表达量呈下降-升高-下降的趋势,在处理48 h后达到峰值, 约为对照的1.6倍;EgrEXPA10的表达量在处理24 h后显著提高,且在处理48 h后达到峰值,约为对照的8~9倍,随后其表达量有所下调,但仍比对照提高(图 6: B)。

|
图 5 不同胁迫下巨桉EgrEXPA8和EgrEXPA10的表达模式 Fig. 5 Expression patterns of EgrEXPA8 and EgrEXPA10 in Eucalyptus grandis under different stresses |

|
图 6 缺硼和缺磷下巨桉EgrEXPA8和EgrEXPA10的表达模式 Fig. 6 Expression patterns of EgrEXPA8 and EgrEXPA10 in Eucalyptus grandis under boron and phosphorus deficiency |
这表明巨桉在不同的胁迫和激素处理下,EgrEXPA8和EgrEXPA10的调控途径及调控机制存在差异性,同时也说明EgrEXPA8和EgrEXPA10在巨桉响应逆境胁迫中具有重要的调控作用。
3 结论和讨论扩展蛋白是植物细胞壁的重要组成部分,主要参与细胞扩张以及一系列发生细胞壁修饰的发育过程以及植物抗病的重要生理过程[28]。本研究通过转录组数据筛选得到2个在茎不同节间差异表达的基因,并克隆得到cDNA序列,利用生物信息学分析发现这2个基因均含有DPBB_1结构域和Pollen_ allerg_1结构域,均为扩展蛋白基因且属于EXPA亚家族成员。采用qRT-PCR技术分析EgrEXPA8和EgrEXPA10基因在不同组织部位、不同节间以及不同胁迫条件下的表达模式,以期初步探索它们在桉树生长调控、逆境胁迫过程中的作用。
由于植物扩展蛋白以酶的催化方式使细胞壁组分松弛、细胞伸展,因此不同的扩展蛋白基因在植物体内的发育阶段具有不同的表达模式,且与细胞生长和组织分化一致[29]。qRT-PCR结果表明, EgrEXPA8和EgrEXPA10基因均在分生组织或者细胞分裂旺盛的部位如嫩叶和茎尖中表达量较高,推测其主要在细胞分裂中起重要作用。另外, EgrEXPA8和EgrEXPA10在茎尖和初生生长占主要作用的上部节间表达量较高,而在次生生长占主导的下部节间表达量急剧降低,推断其可能主要调控初生生长或者负调控次生生长,这与前期转录组的表达趋势一致。已有研究表明外源喷施油菜素内酯(brassinosteroid, BR)可以提高拟南芥AtEXPA5的表达,且在det2-1和bri1-301突变体中AtEXPA5的表达量减少, 而在bzr1-1D功能获得型突变体中表达量增强[30]。近期研究表明BR在木本植物次生生长过程中主要通过促进纤维细胞的增加和减少木质素,同时促进木质部细胞的膨大起作用[31–33]。这表明EgrEXPA8和EgrEXPA10可能参与了BR调控次生木质部发育过程。通过构建EgrEXPA8和EgrEXPA10表达载体,以探索其在桉树生长调控中的作用尤其是木材形成中的作用。
植物生长调节剂(如水杨酸等)是生物胁迫及非生物胁迫反应的重要信号分子,能诱导植物产生持续抗性,从而减缓植物对多种逆境胁迫的伤害。本研究结果表明,EgrEXPA8和EgrEXPA10基因对盐胁迫、水杨酸与茉莉酸甲酯、缺磷、缺硼等逆境的响应方式类似,推测同一亚家族的扩展蛋白对相同胁迫或植物激素可能有相似的响应模式。在盐处理下,巨桉叶片内EgrEXPA8和EgrEXPA10基因的表达量均显著降低,这与地黄(Rehmannia glutinosa)扩展蛋白基因RgEXPA10的表达趋势一致[34]。小麦(Triticum aestivum)扩展蛋白TaEXPB23在茉莉酸甲酯处理后的mRNA水平提高[35],而本研究中EgrEXPA8和EgrEXPA10基因表达在茉莉酸甲酯处理下均被抑制,可能是由于EgrEXPA8和EgrEXPA10基因与TaEXPB23为不同的亚家族,其响应方式不同, 也可能是由于物种不同的原因。硼高效品种甘蓝型油菜(Brassica napus)在缺硼胁迫下根系中的BnaEXPA6、幼叶中的BnaEXPA5及老叶中的BnaEXPA3、BnaEXPA5、BnaEXPA8和BnaEXPA16基因的表达水平均显著高于硼低效品种[36]。巨桉EgrEXPA8和EgrEXPA10基因在长期缺硼胁迫下均显著上调,且EgrEXPA10较EgrEXPA8响应更迅速,说明这2个扩展蛋白可能在桉树缺硼胁迫中发挥着重要作用, 为将来研究桉树中硼响应的机制研究奠定基础。
本研究从巨桉中克隆了2个扩展蛋白基因EgrEXPA8和EgrEXPA10,可能参与桉树初生细胞壁的维持及次生生长,在盐胁迫、茉莉酸甲酯处理下均被抑制表达,在水杨酸处理、缺硼、缺磷处理下均被上调表达,在非生物胁迫响应中起调控作用。
| [1] |
MCQUEEN-MASON S, DURACHKO D M, COSGROVE D J. Two endogenous proteins that induce cell wall extension in plants[J]. Plant Cell, 1992, 4(11): 1425-1433. DOI:10.1105/tpc.4.11.1425 |
| [2] |
COSGROVE D J. Plant expansins: Diversity and interactions with plant cell walls[J]. Curr Opin Plant Biol, 2015, 25: 162-172. DOI:10.1016/j.pbi.2015.05.014 |
| [3] |
LEE Y, CHOI D, KENDE H. Expansins: Ever-expanding numbers and functions[J]. Curr Opin Plant Biol, 2001, 4(6): 527-532. DOI:10.1016/s1369-5266(00)00211-9 |
| [4] |
LI H Y, SHI Y, DING Y N, et al. Bioinformatics analysis of expansin gene family in poplar genome[J]. J Beijing For Univ, 2014, 36(2): 59-67. 李昊阳, 施杨, 丁亚娜, 等. 杨树扩展蛋白基因家族的生物信息学分析[J]. 北京林业大学学报, 2014, 36(2): 59-67. DOI:10.13332/j.cnki.jbfu.2014.02.014 |
| [5] |
DAL SANTO S, VANNOZZI A, TORNIELLI G B, et al. Genomewide analysis of the expansin gene superfamily reveals grapevine-specific structural and functional characteristics[J]. PLoS One, 2013, 8(4): e62206. DOI:10.1371/journal.pone.0062206 |
| [6] |
KENDE H, BRADFORD K, BRUMMELL D, et al. Nomenclature for members of the expansin superfamily of genes and proteins[J]. Plant Mol Biol, 2004, 55(3): 311-314. DOI:10.1007/s11103-004-0158-6 |
| [7] |
YU Z M, KANG B, HE X W, et al. Root hair-specific expansins modulate root hair elongation in rice[J]. Plant J, 2011, 66(5): 725-734. DOI:10.1111/j.1365-313x.2011.04533.x |
| [8] |
YAN A, WU M J, YAN L M, et al. AtEXP2 is involved in seed germination and abiotic stress response in Arabidopsis[J]. PLoS One, 2014, 9(1): e85208. DOI:10.1371/journal.pone.0085208 |
| [9] |
MORRIS K, LINKIES A, MüLLER K, et al. Regulation of seed germination in the close Arabidopsis relative Lepidium sativum: A global tissue-specific transcript analysis[J]. Plant Physiol, 2011, 155(4): 1851-1870. DOI:10.1104/pp.110.169706 |
| [10] |
ZHAO W W, FENG L, HU S B, et al. Cloning and expression analysis of CpEXPA2 gene related to softening of papaya fruit[J]. J Fruit Sci, 2018, 35(7): 785-793. 赵湾湾, 冯力, 胡绍彬, 等. 番木瓜果实软化相关CpEXPA2基因的克隆与表达分析[J]. 果树学报, 2018, 35(7): 785-793. DOI:10.13925/j.cnki.gsxb.20180101 |
| [11] |
HAYAMA H, ITO A, MORIGUCHI T, et al. Identification of a new expansin gene closely associated with peach fruit softening[J]. Postharv Biol Technol, 2003, 29(1): 1-10. DOI:10.1016/s0925-5214(02)00216-8 |
| [12] |
GRAY-MITSUMUNE M, MELLEROWICZ E J, ABE H, et al. Expansins abundant in secondary xylem belong to subgroup A of the α-expansin gene family[J]. Plant Physiol, 2004, 135(3): 1552-1564. DOI:10.1104/pp.104.039321 |
| [13] |
BAJWA K S, SHAHID A A, RAO A Q, et al. Stable transformation and expression of GhEXPA8 fiber expansin gene to improve fiber length and micronaire value in cotton[J]. Front Plant Sci, 2015, 6: 838. DOI:10.3389/fpls.2015.00838 |
| [14] |
KULUEV B, AVALBAEV A, MIKHAYLOVA E, et al. Expression profiles and hormonal regulation of tobacco expansin genes and their involvement in abiotic stress response[J]. Plant Physiol, 2016, 206: 1-12. DOI:10.1016/j.jplph.2016.09.001 |
| [15] |
GOH H H, SLOAN J, DORCA-FORNELL C, et al. Inducible repression of multiple expansin genes leads to growth suppression during leaf development[J]. Plant Physiol, 2012, 159(4): 1759-1770. DOI:10.1104/pp.112.200881 |
| [16] |
CHEN Y H, HAN Y Y, KONG X Z, et al. Ectopic expression of wheat expansin gene TaEXPA2 improved the salt tolerance of transgenic tobacco by regulating Na+/K+ and antioxidant competence[J]. Physiol Plant, 2017, 159(2): 161-177. DOI:10.1111/ppl.12492 |
| [17] |
ZHAO M R, HAN Y Y, FENG Y N, et al. Expansins are involved in cell growth mediated by abscisic acid and indole-3-acetic acid under drought stress in wheat[J]. Plant Cell Rep, 2012, 31(4): 671-685. DOI:10.1007/s00299-011-1185-9 |
| [18] |
ZHANG H, DING Y N, ZHI J K, et al. Over-expression of the poplar expansin gene PtoEXPA12 in tobacco plants enhanced cadmium accumulation[J]. Int J Biol Macromol, 2018, 116: 676-682. DOI:10.1016/j.ijbiomac.2018.05.053 |
| [19] |
MOLLET J C, LEROUX C, DARDELLE F, et al. Cell wall composition, biosynthesis and remodeling during pollen tube growth[J]. Plants, 2013, 2(1): 107-147. DOI:10.3390/plants2010107 |
| [20] |
LI L C, BEDINGER P A, VOLK C, et al. Purification and characterization of four β-expansins (Zea m 1 isoforms) from maize pollen[J]. Plant Physiol, 2003, 132(4): 2073-2085. DOI:10.1104/pp.103.020024 |
| [21] |
TABUCHI A, LI L C, COSGROVE D J. Matrix solubilization and cell wall weakening by β-expansin (group-1 allergen) from maize pollen[J]. Plant J, 2011, 68(3): 546-559. DOI:10.1111/j.1365-313x.2011.04705.x |
| [22] |
RUSSELL S D, BHALLA P L, SINGH M B. Transcriptome-based examination of putative pollen allergens of rice (Oryza sativa ssp. japonica)[J]. Mol Plant, 2008, 1(5): 751-759. DOI:10.1093/mp/ssn036 |
| [23] |
XIE Y J. Research progress on Eucalyptus breeding and its strategy in China[J]. World For Res, 2011, 24(4): 50-54. 谢耀坚. 中国桉树育种研究进展及宏观策略[J]. 世界林业研究, 2011, 24(4): 50-54. DOI:10.13348/j.cnki.sjlyyj.2011.04.015 |
| [24] |
MYBURG A A, GRATTAPAGLIA D, TUSKAN G A, et al. The genome of Eucalyptus grandis[J]. Nature, 2014, 510(7505): 356-362. DOI:10.1038/nature13308 |
| [25] |
ZHAN N, XIE Y J, CHEN H P, et al. Bioinformatics analysis of EXP gene family in Eucalyptus grandis[J]. For Res, 2018, 31(6): 39-46. 詹妮, 谢耀坚, 陈鸿鹏, 等. 巨桉EXP基因家族的生物信息学分析[J]. 林业科学研究, 2018, 31(6): 39-46. DOI:10.13275/j.cnki.lykxyj.2018.06.006 |
| [26] |
LIVAK K J, SCHMITTGEN T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT method[J]. Methods, 2001, 25(4): 402-408. DOI:10.1006/meth.2001.1262 |
| [27] |
SAMPEDRO J, COSGROVE D J. The expansin superfamily[J]. Genome Biol, 2005, 6(12): 242. DOI:10.1186/gb-2005-6-12-242 |
| [28] |
COSGROVE D J. Loosening of plant cell walls by expansins[J]. Nature, 2000, 407(6802): 321-326. DOI:10.1038/35030000 |
| [29] |
CHO H T, COSGROVE D J. Regulation of root hair initiation and expansin gene expression in Arabidopsis[J]. Plant Cell, 2002, 14(12): 3237-3253. DOI:10.1105/tpc.006437 |
| [30] |
PARK C H, KIM T W, SON S H, et al. Brassinosteroids control AtEXPA5 gene expression in Arabidopsis thaliana[J]. Phytochemistry, 2010, 71(4): 380-387. DOI:10.1016/j.phytochem.2009.11.003 |
| [31] |
YAN H F. The Brassinosteroid's effect on the secondary growth of Eucalyptus grandis and preliminary function analysis of the related genes [D]. Beijing: Chinese Academy of Forestry, 2015. 闫慧芳. 油菜素内酯对巨桉次生生长影响及转导途径中相关基因功能初步分析[D]. 北京: 中国林业科学研究院, 2015. |
| [32] |
ZHENG L W, MA J J, ZHANG L Z, et al. Revealing critical mechanisms of BR-mediated apple nursery tree growth using iTRAQ-based proteomic analysis[J]. J Proteomics, 2018, 173: 139-154. DOI:10.1016/j.jprot.2017.12.007 |
| [33] |
PRAMOD S, ANJU M, RAJESH H, et al. Effect of exogenously applied 24-epibrassinolide and brassinazole on xylogenesis and micro-distribution of cell wall polymers in Leucaena leucocephala (Lam) De Wit[J]. J Plant Growth Regul, 2021, 41(1): 404-416. DOI:10.1007/s00344-021-10313-6 |
| [34] |
WANG F Q, ZHOU Y, HUANG Y, et al. Cloning and expression analysis of the expansin gene RgEXPA10 in Rehmannia glutinosa[J]. Acta Pharm Sin, 2015, 50(2): 233-240. 王丰青, 周艳, 黄勇, 等. 地黄扩展蛋白基因RgEXPA10的克隆与表达分析[J]. 药学学报, 2015, 50(2): 233-240. DOI:10.16438/j.0513-4870.2015.02.004 |
| [35] |
HAN Y Y, LI A X, LI F, et al. Characterization of a wheat (Triticum aestivum L.) expansin gene, TaEXPB23, involved in the abiotic stress response and phytohormone regulation[J]. Plant Physiol Biochem, 2012, 54: 49-58. DOI:10.1016/j.plaphy.2012.02.007 |
| [36] |
WU T, ZENG N, LI W, et al. Genome-wide identification of the expansin gene family and differences in transcriptional responses to boron deficiency in Brassica napus L.[J]. Plant Sci J, 2021, 39(1): 59-75. 吴涛, 曾妮, 李巍, 等. 甘蓝型油菜扩展蛋白家族的全基因组鉴定及其对缺硼胁迫响应的差异分析[J]. 植物科学学报, 2021, 39(1): 59-75. DOI:10.11913/PSJ.2095-0837.2021.10059 |
 2023, Vol. 31
2023, Vol. 31



