油茶(Camellia oleifera)是重要的木本油料树种,茶籽中提取的茶油被誉为“东方橄榄油”,不饱和脂肪酸含量可高达90%,且富含维生素E,具有极高的营养价值和经济价值[1]。油茶植株生长发育、果实发育和种子萌发等生长发育过程,受植物体内的生长素、赤霉素、脱落酸、细胞分裂素等多种植物激素的调控。近年来研究表明,作为一种重要植物激素,赤霉素在油茶等植物的花芽分化、枝条生长、种籽品质与萌发等多方面都具有影响[2–6]。
赤霉素(gibberellic acid, GA)在种子萌发、花芽分化、侧枝伸长和生长发育中具有重要作用,其信号转导分子调控过程相对明确;DELLA蛋白作为赤霉素信号传导途径的负调控因子,对赤霉素信号转导发挥抑制作用[7–9]。DELLA蛋白不具有DNA结合结构域,当没有活性GA刺激存在时,DELLA蛋白通过与转录因子结合,抑制或激活转录因子的DNA结合活性,从而抑制或激活靶基因表达,调控下游GA响应基因的表达,阻遏植物的生长发育;而GA可通过激活GA受体GID1与DELLA蛋白结合形成GID1-GA-DELLA复合体,诱导DELLA蛋白的泛素化降解,从而达到去阻遏的效果[10]。DELLA蛋白属于GRAS家族,也是GRAS家族研究最广泛的亚群部落之一[11],已在拟南芥(Arabidopsis thaliana)[12]、茶树(C. sinensis)[13]、番茄(Lycopersicon esculenttum)[14]、水稻(Oryza sativa)[11]、大麦(Hordeum vulgare)[15]等许多植物中鉴定到,但对油茶中DELLA基因家族的分离鉴定和进化分析还未见报道。DELLA蛋白不仅作为GA信号转导途径中的关键调控因子,也是赤霉素与生长素、脱落酸、独脚金内酯等多个信号通路之间的相互作用节点[16]。
本研究通过同源克隆分离鉴定了油茶的5个DELLA基因,对其编码的蛋白进行理化性质、氨基酸序列比对及系统进化等生物信息学分析,并对其表达模式、对植物激素和非生物胁迫的响应活性进行研究,为油茶DELLA蛋白的功能解析和DELLA介导的生长发育调控研究奠定基础。
1 材料和方法 1.1 材料本研究选取2 a生的‘长林4号’油茶(Camellia oleifera)为材料,采集幼嫩叶片、花、茎、果实和芽,放于液氮中速冻,在-80 ℃冰箱保存,用于总RNA的提取。分别用50 μmol/L赤霉素(GA3)、独脚金内酯(SL)、玉米素(KT)、脱落酸(ABA)、茉莉酸(JA)和10 μmol/L 2, 4-D进行喷施,以0.01%乙醇水溶液喷施作为对照。非生物胁迫包括脱水干旱、300 mmol/L NaCl和4 ℃低温处理, 分别于处理0、3、6、9、12、24 h后采集幼芽,置于液氮中速冻,在–80 ℃冰箱保存,用于荧光定量PCR分析。每处理3个重复。
1.2 总RNA提取与cDNA合成样品在研钵中液氮低温研磨,按照植物RNA快速提取试剂盒说明书(RNA Easy Fast Plant Tissue Kit, 天根)提取RNA,然后用琼脂糖凝胶电泳检测,RNA提取质量符合标准后,使用MLV逆转录酶试剂(Promega)逆转录合成cDNA,5倍稀释后储存于–20℃冰箱备用。
1.3 DELLA基因克隆利用Phytozome网站(https://phytozome-next.jgi.doe.gov/)检索拟南芥、番茄、水稻、茶树中的DELLA基因数据,根据序列比对分析,选定基因克隆区域,利用Primer Premier 5软件设计基因克隆引物(表 1)。以油茶cDNA为模板,使用高保真DNA聚合酶(Prime STAR Max polymerase, TaKaRa)扩增目的基因。PCR反应体系和程序按照DNA聚合酶操作说明实施。PCR产物经琼脂糖凝胶电泳检测后,转移至pMD19-T载体,将阳性DH5α菌株送至测序公司进行Sanger测序。
| 表 1 所用引物 Table 1 Primers in the test |
利用在线软件ProtParam (https://web.expasy.org/protparam/)预测基因编码区长度、蛋白的氨基酸序列长度、分子量、分子式、等电点、疏水指数和脂溶系数等;利用在线软件NetPhos 3.1 Server (https://services.healthtech.dtu.dk/service.php?NetPhos-3.1)对DELLA蛋白磷酸化位点进行预测;在植物基因组网站Phytozome中获取拟南芥、番茄、水稻及茶树等物种的DELLA同源基因数据,使用DNAMAN软件对DELLA家族成员的氨基酸序列进行比对,利用在线工具MEME (https://meme-suite.org/meme/tools/meme)对DELLA的保守基序进行预测。利用MEGA 7.0软件对不同植物的DELLA构建系统进化树;使用SWISS-MODEL在线软件(https://swissmodel.expasy.org/),以2ZSH.1.B蛋白模型为模板,采用同源建模方式进行蛋白三级结构预测分析。
1.5 基因表达分析对克隆的油茶DELLA基因进行核苷酸序列分析,利用Primer Premier 5软件设计荧光定量PCR引物(表 1),引物参数为:PCR产物长度100~200 bp,引物退火温度约为60 ℃。以CoCESA和CoTUBα3为内参基因[17],使用SYBR Green Ⅰ嵌合荧光法(2×RealStar Fast SYBR qPCR Mix, GenSTAR)进行荧光定量PCR检测,分析DELLA基因在油茶不同组织中的相对表达量。实验设置3个生物学重复和3个技术重复,采用2–△△CT计算相对表达量。
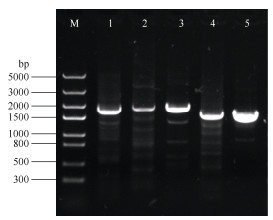
2 结果和分析 2.1 DELLA基因的克隆将提取的油茶总RNA进行反转录成cDNA, PCR扩增产物经凝胶电泳检测,获得5个克隆片段,长度均为1 500~2 000 bp,与其他物种中已知DELLA基因相似(图 1)。PCR产物经纯化回收连接至克隆载体中,转化DH5α大肠杆菌感受态细胞,菌落PCR筛选获得阳性菌落,经测序获得油茶DELLA基因序列。所克隆的5个油茶DELLA基因分别命名为CoDELLA1、CoDELLA2、CoDELLA3、CoDELLA4和CoDELLA5,其编码区长度为1 581~1 875 bp。
|
图 1 油茶DELLA基因的PCR扩增。M: Marker; 1~5: 5个CoDELLA基因。 Fig. 1 PCR amplification of DELLA genes in Camellia oleifera. M: Marker; 1-5: Five CoDELLAs. |
利用在线软件ProtParam对油茶CoDELLAs基因编码的蛋白质进行理化性质预测,CoDELLAs基因分别编码527~625个氨基酸,蛋白质分子量为5.78~6.90 kD,等电点为5.24~5.52,脂溶系数为–0.314~–0.091,疏水指数为75.83~91.10,均为亲水性蛋白(表 2)。
| 表 2 油茶5个DELLA的基本信息 Table 2 Basic informations of 5 DELLAs in Camellia oleifera |
经在线软件NetPhos 3.1 Server预测,油茶CoDELLAs蛋白的磷酸化位点中以丝氨酸残基最多,其次为苏氨酸和酪氨酸残基(图 2)。同时,磷酸化位点多分布于CoDELLAs蛋白的N端DELLA结构域,CoDELLA1和CoDELLA2蛋白的磷酸化位点最为典型,在DELLA结构域和GRAS结构域之间(polyS/T/V)存在大量磷酸化位点(图 2),表明CoDELLAs蛋白具有典型的DELLA蛋白磷酸化位点。

|
图 2 油茶CoDELLA1~CoDELLA5 (A~E)的磷酸化位点预测 Fig. 2 Prediction of phosphorylation sites in CoDELLA1-CoDELLA5 (A-E) of Camellia oleifera |
利用DNAMAN软件对油茶的DELLAs、拟南芥的RGA、GAI、RGL1、RGL2和RGL3、番茄的GAI及水稻的SLR1进行氨基酸序列比对,结果表明(图 3),这些物种的DELLA蛋白均有典型的DELLA结构域(DELLA基序、TVHYNP结构域)、GRAS结构域(VHVID基序、RVER基序、SAW基序)和核定位信号(NLS)。进一步利用MEME软件进行蛋白质保守域分析,结果表明,这些物种DELLA氨基酸序列的保守基序存在差异,但均含有高度保守的特征性结构域,DELLA蛋白特有的结构域中, DELLA-LExLE基序(基序5)、TVHYNP基序(基序8)均存在(图 4: B~D)。此外,油茶DELLA蛋白的N端序列均具有典型DELLA结构域(图 4: E~I)。这说明,油茶的5个CoDELLA蛋白均具有保守的DELLA结构域和GRAS结构域,都属于典型的DELLA蛋白,这些蛋白可能在功能上也具有高度保守性。

|
图 3 不同物种中DELLA的氨基酸序列比对。Co: 油茶; At: 拟南芥; Le: 番茄; Os: 水稻; *: DELLA-LExLE和TVHYNP基序。 Fig. 3 Alignment of amino acid sequences of DELLA among different species. Co: Camellia oleifera; At: Arabidopsis thaliana; Le: Lycopersicon esculentum; Os: Oryza sativa; *: DELLA-LExLE and TVHYNP motifs. |

|
图 4 油茶DELLA蛋白结构预测。A: 不同物种的DELLA蛋白保守基序; B: 保守基序的氨基酸序列; C: DELLA基序的氨基酸分布特征; D: TVHYNP基序氨基酸分布特征; E~I: CoDELLA1~CoDELLA5蛋白三级结构预测。 Fig. 4 Structure prediction of DELLA proteins in Camellia oleifera. A: Conserved motifs of DELLA in different species; B: Amino acid sequence of conserved mitifs; C: Amino acid distribution of DELLA motif; D: Amino acid distribution of TVHYNP motif; E-I: Predicted 3D structure of CoDELLA1-CoDELLA5, respectively. |
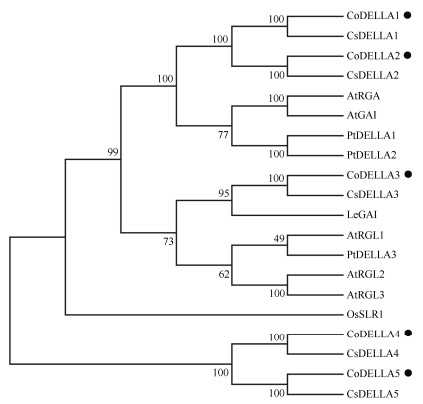
利用MEGA 7.0序列分析软件,对油茶和茶树的DELLA家族、拟南芥的DELLA家族(GAI、RGA、RGL1、RGL2和RGL3)、番茄的PRO、水稻的SLR1以及杨树的DELLA1、DELLA2和DELLA3进行系统发育树构建。从图 5可见,相较于拟南芥和水稻等物种,油茶的5个DELLA分别与茶树的CsDELLA1~CsDELLA5具有较高同源性,这与油茶和茶树同属山茶科植物的分类是相符的,说明油茶的5个DELLA蛋白在进化方面是相对较为保守的。油茶的CoDELLA1、CoDELLA2与拟南芥的AtGAI、AtRGA同源性较高,而CoDELLA3与番茄SlPRO的同源性较高,表明他们在功能上可能具有一定的相似性。
|
图 5 基于DELLA氨基酸序列的不同物种系统进化树。Co: 油茶; Cs: 茶树; At: 拟南芥; Le: 番茄; Os: 水稻; Pt: 杨树。 Fig. 5 Phylogenetic tree of different species based on DELLA amino acid sequences. Co: Camellia oleifera; Cs: C. sinensis; At: Arabidopsis thaliana; Le: Lycopersicon esculentum; OS: Oryza sativa; Pt: Populus trichocarpa. |
基因在植物不同组织中的表达特征与其功能发挥的组织特异性紧密相关。采用实时荧光定量PCR对5个CoDELLAs基因的表达模式进行分析。结果表明,油茶CoDELLAs基因在不同组织中均有表达,且均以花中的表达水平较高(图 6: A)。CoDELLA1和CoDELLA5的表达模式相似,均以花中的表达较高。CoDELLA2在茎和果实中的表达要低于叶片和花。CoDELLA3和CoDELLA4在果实中的表达水平均高于叶、茎和花。这表明CoDELLAs可能在油茶不同组织对赤霉素信号转导途径发挥调控作用。

|
图 6 油茶CoDELLAs基因的表达模式。A: 组织表达模式; B: 赤霉素; C: 独脚金内酯; D: 激素和非生物胁迫; *: P < 0.05 Fig. 6 Expression patterns of CoDELLAs in Camellia oleifera. A: Tissue expression pattern; B: Gibberellin; C: Strigolactone; D: Phytohormones and abiotic stresses; *: P < 0.05 |
用50 μmol/L赤霉素(GA3)处理油茶植株,取处理3、6、9、12、24 h后的幼芽进行荧光定量PCR检测,结果表明,与对照相比,GA3处理6 h的CoDELLA1表达量提高;而CoDELLA2和CoDELLA3在6~9 h的表达量略微下降,但在处理12 h后表达量提高;CoDELLA4与CoDELLA5的表达量差异不大(图 6: B)。这说明不同CoDELLAs对GA3的响应时间存在差异。
CoDELLA1和CoDELLA2经50 μmol/L外源独脚金内酯(SL)处理3、12和24 h后的表达量比对照提高,而处理6、9 h的表达量略有下降(图 6: C), 而CoDELLA3、CoDELLA4和CoDELLA5的表达量变化不大,推测独脚金内酯可能对赤霉素信号转导活性有影响。
此外,植物在激素与胁迫响应中存在大量信号交联,为进一步了解CoDELLA基因的潜在功能, 分别对激素和胁迫处理6 h的油茶幼芽中CoDELLAs的表达特性进行了分析。结果表明, 与对照相比, 激素和胁迫处理对CoDELLA表达的影响不同(图 6: D), CoDELLA1和CoDELLA3的表达均受ABA、JA和干旱胁迫的诱导,且CoDELLA1的表达受2, 4-D的诱导;CoDELLA2、CoDELLA4、CoDELLA5的表达则受2, 4-D、盐胁迫等的抑制。这说明,油茶中不同CoDELLA的表达受多种激素和胁迫的调控, CoDELLA基因可能参与多种激素和胁迫调控的复杂生长发育过程。
3 结论和讨论在植物生长发育过程中,赤霉素通过抑制DELLA蛋白,激活大量下游响应基因的表达,从而影响种子休眠与萌发、植株大小及胁迫响应等生长发育过程[12, 15, 18–19]。赤霉素对油茶侧枝发育、种子萌发、花芽分化及茶籽油脂代谢等方面都具有重要调控作用,但其调控作用的分子机制尚需深入研究[5, 20–22]。本研究根据油茶全基因组序列,通过Primer Primer 5设计基因引物,采用PCR技术成功克隆了油茶的5个DELLA基因,通过对不同植物的DELLA氨基酸序列进行比对,结果表明DELLA蛋白具有典型的DELLA结构域和GRAS结构域,且保守基序的位置相近。DELLA蛋白N端的DELLA结构域,是其与赤霉素受体GID1蛋白结合互作的重要部位,具有调控细胞内赤霉素信号转导的作用[18, 23]。将油茶、茶树、拟南芥、番茄和杨树的DELLA家族构建系统发育树,可见油茶CoDELLAs与茶树的CsDELLAs具有较高的同源性,且CoDELLA1、CoDELLA2、CoDELLA3与拟南芥和番茄的DELLA具有较高同源性,表明其在功能上可能也具有一定相似性。磷酸化等多种翻译后修饰对蛋白稳定性具有重要影响,DELLA蛋白的丝氨酸、苏氨酸的磷酸化参与DELLA蛋白稳定性调控,该磷酸化作用主要出现在DELLA蛋白的DELLA/TVHYNP基序, 以及位于DELLA结构域与GRAS结构域之间的富含丝氨酸、苏氨酸、缬氨酸(polyS/T/V)的调控位点[11, 24–25]。CoDELLA的N端和polyS/T/V区域存在大量潜在的磷酸化位点, 表明这些CoDELLA均具有典型的蛋白稳定性调控机制。
实时荧光定量PCR分析表明,CoDELLAs在花、茎、叶、果实中均有表达。CoDELLA1、CoDELLA2和CoDELLA5均在花中的表达量最高,CoDELLA3和CoDELLA4在果中的表达均高于叶、茎和花。已有研究表明,拟南芥的GAI在所有组织中均有表达,RGL1的表达主要集中在花器官,而RGL1、RGL2和RGL3仅在萌发种子、花和果中表达量较高[19, 26]。同时, 花生(Arachis hypogaea)的AhDELLA3和AhDELLA4基因在花、种子中的表达量高于其他组织,暗示其在这些部位的功能特异性[27],故推测CoDELLAs可能在油茶赤霉素信号转导途径发挥调控作用。
本研究表明,外源赤霉素处理油茶,CoDELLA1的表达量在6 h、CoDELLA2和CoDELLA3在12 h提高,这与植物体内赤霉素的反馈调节相符。同样,独脚金内酯处理油茶,CoDELLA1和CoDELLA2在3、12和24 h的表达量提高, 而在6、9 h时略微下降。已有研究表明,DELLA蛋白介导的赤霉素信号转导途径通常与生长素、细胞分裂素等植物激素信号途径存在信号交联作用,其中独脚金内酯作为一种新发现的植物激素,与赤霉素信号途径之间也存在潜在相互作用[28],且其分子机制也比较相似[29], 故推测独脚金内酯可能对赤霉素信号转导的活性有一定影响。此外,油茶CoDELLAs的表达也受生长素、细胞分裂素及盐胁迫等不同途径的调控,表明其可能在植物赤霉素信号抑制作用和赤霉素信号途径与多种不同途径之间的信号交联作用发挥重要调控作用。
综上,油茶中的5个DELLA蛋白均与茶树等植物的DELLA同源性较高,且在花和果实中表达较高,说明该类蛋白的结构保守性和在不同组织中作用的差异性。GA3和SL等激素及干旱等非生物胁迫下CoDELLAs表达水平的差异说明CoDELLAs功能的多样性。
| [1] |
MA L, CHEN Y Z. Analyzed camellia oil of function characteristics[J]. Chin Agric Sci Bull, 2009, 25(8): 82-84. 马力, 陈永忠. 茶油的功能特性分析[J]. 中国农学通报, 2009, 25(8): 82-84. |
| [2] |
LIU Q T, NIE R B, HUANG Y L, et al. Studies on increasing fluecured tobacco seed vigor with gibberellins[J]. Seed, 2008, 27(3): 75-77. 刘启彤, 聂荣邦, 黄一兰, 等. 赤霉素提高烤烟种子活力的研究[J]. 种子, 2008, 27(3): 75-77. DOI:10.3969/j.issn.1001-4705.2008.03.024 |
| [3] |
LI N. The influence of spraying Gibberellin acid on rice seed quality[J]. Guangdong Agric Sci, 2011, 38(15): 13-15. 李楠. 喷施赤霉素对水稻种子品质的影响[J]. 广东农业科学, 2011, 38(15): 13-15. DOI:10.3969/j.issn.1004-874X.2011.15.004 |
| [4] |
LIU W, XIE B, NI G P, et al. Influence of gibberellin and amino acid on branch and leaf growth of Syzygium grijsii[J]. Bull Bot Res, 2011, 31(2): 218-226. 刘玮, 谢冰, 倪国平, 等. 赤霉素和氨基酸对三叶赤楠(Syzygium grijsii)枝叶生长影响研究[J]. 植物研究, 2011, 31(2): 218-226. DOI:10.7525/j.issn.1673-5102.2011.02.016 |
| [5] |
CHEN X, SUN Y, LI J A. Effects of GA and PP333 on growth of Camellia oleifera seedlings[J]. Nonwood For Res, 2013, 31(2): 86-90. 陈显, 孙颖, 李建安. 赤霉素和多效唑对油茶幼株生长的影响[J]. 经济林研究, 2013, 31(2): 86-90. DOI:10.14067/j.cnki.1003-8981.2013.02.028 |
| [6] |
ZHANG Q, SU Y Q, XU Y M. Effect of harvesting time on contents of secondary metabolites in the leaf of Eucommia ulmoides treated by gibberellin acid[J]. J NW For Univ, 2010, 25(6): 130-133. 张强, 苏印泉, 徐咏梅. 采样时间对赤霉素处理杜仲叶次生代谢物含量的影响[J]. 西北林学院学报, 2010, 25(6): 130-133. |
| [7] |
YUE C, ZENG J M, CAO H L, et al. Gibberellins metabolism and signaling pathway in higher plant[J]. Plant Physiol J, 2012, 48(2): 118-128. 岳川, 曾建明, 曹红利, 等. 高等植物赤霉素代谢及其信号转导通路[J]. 植物生理学报, 2012, 48(2): 118-128. DOI:10.13592/j.cnki.ppj.2012.02.014 |
| [8] |
DAVIÈRE J M, ACHARD P. Gibberellin signaling in plants[J]. Development, 2013, 140(6): 1147-1151. DOI:10.1242/dev.087650 |
| [9] |
ZHANG L, HUANG Z Y, SU S W, et al. Effects of exogenous hormones on the growth of lotus and expression of related genes[J]. J NW For Univ, 2019, 34(2): 35-41. 张琳, 黄志远, 苏少文, 等. 外源激素对荷花生长及相关基因表达的影响[J]. 西北林学院学报, 2019, 34(2): 35-41. DOI:10.3969/j.issn.1001-7461.2019.02.06 |
| [10] |
HUANG X Z, JIANG C F, LIAO L L, et al. Progress on molecular foundation of GA biosynthesis pathway and signaling[J]. Chin Bull Bot, 2006, 23(5): 499-510. 黄先忠, 蒋才富, 廖立力, 等. 赤霉素作用机理的分子基础与调控模式研究进展[J]. 植物学通报, 2006, 23(5): 499-510. DOI:10.3969/j.issn.1674-3466.2006.05.006 |
| [11] |
DAI C, XUE H W. Rice early flowering1, a CKI, phosphorylates DELLA protein SLR1 to negatively regulate gibberellin signalling[J]. EMBO J, 2010, 29(11): 1916-1927. DOI:10.1038/emboj.2010.75 |
| [12] |
TYLER L, THOMAS S G, HU J H, et al. DELLA proteins and gibberellin-regulated seed germination and floral development in Arabidopsis[J]. Plant Physiol, 2004, 135(2): 1008-1019. DOI:10.1104/pp.104.039578 |
| [13] |
HAN Y X, DAI H W, ZHENG S T, et al. Identification and expression analysis of the DELLA gene family in Camellia sinensis (L.) O. Ktze[J]. Plant Sci J, 2020, 38(5): 644-653. 韩雨欣, 代洪苇, 郑姝婷, 等. 茶树DELLA基因家族的鉴定及表达分析[J]. 植物科学学报, 2020, 38(5): 644-653. DOI:10.11913/PSJ.2095-0837.2020.50644 |
| [14] |
OLSZEWSKI N, SUN T P, GUBLER F. Gibberellin signaling: Biosynthesis, catabolism, and response pathways[J]. Plant Cell, 2002, 14(S1): S61-S80. DOI:10.1105/tpc.010476 |
| [15] |
FU X D, RICHARDS D E, AIT-ALI T, et al. Gibberellin-mediated proteasome-dependent degradation of the barley DELLA protein SLN1 repressor[J]. Plant Cell, 2002, 14(12): 3191-3200. DOI:10.1105/tpc.006197 |
| [16] |
ZHOU P, LI Q F, XIONG M, et al. Advances in DELLA proteinmediated phytohormonal crosstalk in regulation of plant growth and development[J]. Plant Physiol J, 2020, 56(4): 661-671. 周鹏, 李钱峰, 熊敏, 等. DELLA蛋白介导的激素互作调控植物生长发育研究进展[J]. 植物生理学报, 2020, 56(4): 661-671. DOI:10.13592/j.cnki.ppj.2019.0570 |
| [17] |
ZHOU C F, LIN P, YAO X H, et al. Selection of reference genes for quantitative real-time PCR in six oil-tea Camellia based on RNA-seq[J]. Mol Biol, 2013, 47(6): 836-851. DOI:10.1134/S0026893313060198 |
| [18] |
ZHU Z G, KANG X J, LOR V S, et al. Characterization of a semidominant dwarfing PROCERA allele identified in a screen for CRISPR/Cas9-induced suppressors of loss-of-function alleles[J]. Plant Biotechnol J, 2019, 17(2): 319-321. DOI:10.1111/pbi.13027 |
| [19] |
WEN C K, CHANG C. Arabidopsis RGL1 encodes a negative regulator of gibberellin responses[J]. Plant Cell, 2002, 14(1): 87-100. DOI:10.1105/tpc.010325 |
| [20] |
TIAN R Y, GUO Y, WANG Y G, et al. Effect of gibberellin treatment and wet sand stratification on seed germination of Camellia oleifera[J]. Seed, 2010, 29(8): 85-88. 田如英, 郭颖, 王元国, 等. 赤霉素浸种与湿沙层积对油茶种子发芽的影响[J]. 种子, 2010, 29(8): 85-88. DOI:10.3969/j.issn.1001-4705.2010.08.026 |
| [21] |
WEN Y, SU S C, MA L Y, et al. Effects of gibberellins on flower bud formation and fruit quality in Camellia oleifera[J]. J Zhejiang Agric For Univ, 2015, 32(6): 861-867. 温玥, 苏淑钗, 马履一, 等. 赤霉素处理对油茶花芽形成和果实品质的影响[J]. 浙江农林大学学报, 2015, 32(6): 861-867. DOI:10.11833/j.issn.2095-0756.2015.06.006 |
| [22] |
CAI Y, WANG D X, CHEN S C, et al. Effect of gibberellin on growth of spring shoots and flower bud differentiation in Camellia osmantha[J]. J SW For Univ, 2020, 40(4): 180-184. 蔡娅, 王东雪, 陈仕昌, 等. 赤霉素对香花油茶花芽分化和春梢生长的影响[J]. 西南林业大学学报, 2020, 40(4): 180-184. DOI:10.11929/j.swfu.201908001 |
| [23] |
UEGUCHI-TANAKA M, NAKAJIMA M, KATOH E, et al. Molecular interactions of a soluble gibberellin receptor, GID1, with a rice DELLA protein, SLR1, and gibberellin[J]. Plant Cell, 2007, 19(7): 2140-2155. DOI:10.1105/tpc.106.043729 |
| [24] |
BLANCO-TOURIÑÁN N, SERRANO-MISLATA A, ALABADÍ D. Regulation of DELLA proteins by post-translational modifications[J]. Plant Cell Physiol, 2020, 61(11): 1891-1901. DOI:10.1093/pcp/pcaa113 |
| [25] |
QIN Q Q, WANG W, GUO X L, et al. Arabidopsis DELLA protein degradation is controlled by a type-one protein phosphatase, TOPP4[J]. PLoS Genet, 2014, 10(7): e1004464. DOI:10.1371/journal.pgen.1004464 |
| [26] |
LEE K P, PISKUREWICZ U, TUREČKOVÁ V, et al. A seed coat bedding assay shows that RGL2-dependent release of abscisic acid by the endosperm controls embryo growth in Arabidopsis dormant seeds[J]. Proc Natl Acad Sci USA, 2010, 107(44): 19108-19113. DOI:10.1073/pnas.1012896107 |
| [27] |
AN J, HOU L, LI C, et al. Cloning and expression analysis of four DELLA genes in peanut[J]. Russ J Plant Physiol, 2015, 62(1): 116-126. DOI:10.1134/S1021443715010021 |
| [28] |
WALLNER E S, LÓPEZ-SALMERÓN V, GREB T. Strigolactone versus gibberellin signaling: Reemerging concepts?[J]. Planta, 2016, 243(6): 1339-1350. DOI:10.1007/s00425-016-2478-6 |
| [29] |
WATERS M T, GUTJAHR C, BENNETT T, et al. Strigolactone signaling and evolution[J]. Annu Rev Plant Biol, 2017, 68: 291-322. DOI:10.1146/annurev-arplant-042916-040925 |
 2023, Vol. 31
2023, Vol. 31



