2. 华南国家植物园, 广州 510650;
3. 中国科学院大学, 北京 100049;
4. 莱斯特大学遗传学和基因组生物学系, 莱斯特 LE1 7RH, 英国;
5. 中国科学院核心植物园, 广州 510650
2. South China National Botanical Garden, Guangzhou 510650, China;
3. University of Chinese Academy of Sciences, Beijing 100049, China;
4. Department of Genetics and Genome Biology, University of Leicester, Leicester, LE1 7RH, UK;
5. Center for Conservation Biology, Core Botanical Gardens, Chinese Academy of Sciences, Guangzhou 510650, China
Beta-葡聚糖是籽粒胚乳和糊粉层细胞壁的主要非纤维素多糖[1],在谷类作物及其他单子叶植物中均有分布[2]。Beta-葡聚糖可增加胆汁酸排泄,延迟葡萄糖吸收[3],降低胆固醇及血糖水平[4],在食品、药品等领域中具有较高的应用价值。最初在水稻(Oryza sativa)基因组中鉴定到β-葡聚糖合成酶基因家族成员CslF亚基因家族[5],并通过异源表达实验,证实CslF亚基因家族编码β-葡聚糖合成酶[6]。之后46种被子植物的研究表明,CslH和CslJ亚基因家族也具有合成β-葡聚糖的功能[7]。
由于对β-葡聚糖合成的调控网络,以及物种间β-葡聚糖含量差异的遗传因素缺乏精准认知, 作物β-葡聚糖育种还停留在传统育种阶段。本文拟通过整理β-葡聚糖合成酶基因家族研究进展, 提出未来研究的方向,为作物β-葡聚糖定向育种提供理论支撑。
1 Beta-葡聚糖的化学特性和功能应用 1.1 化学结构和溶解度Beta-葡聚糖是由d-吡喃葡萄糖苷单元通过β-(1, 3)和β-(1, 4)糖苷键连接组成的非纤维素多糖, 又名混合线性葡聚糖[Mixed-linkage (1, 3;1, 4)-β-D-glucan, MLG],无分支或取代基(图 1)。Beta-葡聚糖链中, β-(1, 4)糖苷键赋予葡聚糖链刚性,而随机分布的β-(1, 3)糖苷键增加了葡聚糖链空间扭转的灵活性[8]。邻近的糖苷键相同时,葡聚糖链空间扭曲度减弱,使葡聚糖链趋向聚合,即溶解度降低,水溶液凝胶特性增加[9]。

|
图 1 (1, 3;1, 4)-β-D-葡聚糖化学结构(根据McFarlane等[10]重绘) Fig. 1 Chemical structure of representative (1, 3;1, 4)-β-D-glucan (Redraw from McFarlane et al.[10]) |
Beta-葡聚糖的精细结构,表现为寡糖聚合度(degree of polymerization, DP)的比值。地衣酶能特异地断开β-(1, 4)糖苷键还原端,获得多种寡糖产物, 其中以β-纤维三糖(G4G4G3G)和β-纤维四糖(G4 G4G4G3G)为主,两者的比值DP3:DP4即为DP比值[11–12]。当DP比值为1~2.5时,β-葡聚糖的溶解度较高[13]。燕麦β-葡聚糖含量较高(6%~8%),具有较好的水溶性,是日常饮食中β-葡聚糖的理想来源[14] (表 1)。
| 表 1 作物中β-葡聚糖含量及溶解度 Table 1 Beta-glucan content and solubility in cereal crops |
Beta-葡聚糖在植物生长发育过程中发挥重要功能[15]。缺失β-葡聚糖的突变体,植株生长速率较低,细胞壁变薄,花药和花丝变形导致雄性不育[16]。Beta-葡聚糖还与葡萄糖的贮藏利用有关[17],植物通过水解细胞壁中的β-葡聚糖获得葡萄糖,由维管束运输到其他部位,提供代谢所需的能量[18–19]。
Beta-葡聚糖可有效降低血糖,改善血液中的胰岛素水平[20–21],参与人体胆汁酸的调节、胆固醇代谢、影响肠道微生物组成[22]。在小鼠试验中,β-葡聚糖能增强免疫系统应答能力[23],降低呼吸道的易感性[24]。Chang等[25]报道稳定摄入β-葡聚糖,可改善腹部脂肪堆积情况,降低人体肥胖度。在食品中添加β-葡聚糖,显著降低血糖生成指数,在烘焙类食物及肉奶制品中,常用β-葡聚糖替代脂肪[26]。Beta-葡聚糖有良好的皮肤渗透性,临床实验中表现出减少细纹生成[27],抑制微生物慢性感染,促进皮肤损伤后的愈合过程等功效[28],相关产品已投入生产。然而,β-葡聚糖对麦芽啤酒酿造会产生不良影响, 发酵过程中残余的β-葡聚糖增加过滤成本,并影响口感[29]。在单胃动物的饲料中,β-葡聚糖会降低动物胃肠道的消化吸收效率[30]。
2 Beta-葡聚糖合成酶基因家族 2.1 Beta-合成酶基因家族成员 2.1.1 CslF、CslH和CslJ亚基因家族Beta-葡聚糖合成酶基因家族包括3个主要成员: CslF、CslH和CslJ亚基因家族,都属于类纤维素合酶(cellulose synthase-like)基因家族(Csl)。该基因家族与多种非纤维素多糖的合成有关。早期拟南芥(Arabidopsis thaliana)的研究表明,Csl基因家族包括5个亚家族(CslA~CslE)[31]。2002年,Hazen等[5]在水稻基因组中鉴定到新的Csl家族成员:CslF和CslH亚基因家族。由于双子叶植物中未鉴定到这两个亚基因家族存在,有学者推测CslF和CslH亚基因家族为单子叶植物特有的Csl家族成员。
CslF是首个被证实与β-葡聚糖合成有关的亚基因家族。Burton等[6]将与β-葡聚糖含量相关的数量性状位点(quantitative trait locus, QTL)比对到水稻基因组中,在7号染色体上鉴定到6个基因组成的CslF基因簇(OsCslF1、OsCslF2、OsCslF3、OsCslF4、OsCslF8和OsCslF9),在8号和10号染色体上鉴定到OsCslF6和OsCslF7基因。通过农杆菌介导法,将3个基因OsCslF2、OsCslF4和OsCslF9转入拟南芥基因组中异源表达,在叶片表皮层细胞壁中检测到微量的β-葡聚糖,证实OsCslF基因参与β-葡聚糖合成[6]。
物种间CslF亚基因家族成员的数目存在差异, 且部分基因只存在于特定物种中(表 2)。Burton等[32]在大麦(Hordeum vulgare) 2号染色体上鉴定到与水稻相同的CslF基因簇,除HvCslF3、HvCslF4、HvCslF8外,还包括新的HvCslF10基因, 而HvCslF6、HvCslF7和HvCslF9等基因分布在其他染色体上。Schreiber等[33]在重新组装的大麦基因组中,鉴定出3个在水稻中未发现的CslF基因:HvCslF11、HvCslF12和HvCslF13。
| 表 2 作物中β-葡聚糖合成酶基因家族成员 Table 2 Number of the β-glucan synthase gene family members in cereal crops |
CslF基因的异源表达实验中,β-葡聚糖的合成量较低,据此Doblin等[34]认为可能存在其他基因共同调控合成β-葡聚糖,如大麦QTL位点附近的基因HvCslH1。通过将HvCslH1转入拟南芥中进行异源表达,Doblin等[34]报道叶片细胞壁中存在β-葡聚糖沉积,证实CslH家族参与β-葡聚糖合成。CslH亚基因家族在早熟禾亚科(Pooideae)的大麦和燕麦(Avena sativa)中仅有单个成员,而在禾本科其他亚科的物种中则有多个成员[35]。
Little等[7]的研究指出,CslF、CslH和CslJ亚家族均可介导β-葡聚糖的合成[7]。Farrokhi等[36]推测与CslF和CslH亚家族类似,单子叶植物中的CslJ亚家族可能参与β-葡聚糖合成。其后在双子叶植物中,也发现CslJ基因存在,而该基因的功能一直未得到确认[37]。Little等[7]将双子叶植物中的CslJ亚家族命名为CslM,目前CslJ亚家族仅分布在单子叶植物中。通过异源表达实验,Little等[7]证实CslJ亚基因家族成员HvCslJ基因参与β-葡聚糖合成,表达活性类似CslH亚家族。
燕麦β-葡聚糖合成酶基因家族的研究不多。Newell等[38]比较了燕麦β-葡聚糖含量相关的QTL位点,有1个QTL标记与水稻7号染色体上的CslF基因簇具有序列同源性。Zhang等[39]采用同源序列搜索,在燕麦基因组中鉴定到5个CslF基因家族成员(AsCslF3、AsCslF4、AsCslF6、AsCslF8和AsCslF9), CslH与CslJ亚家族都仅具有单个成员,而CslF基因簇在燕麦基因组中是否存在有待检测。
2.1.2 主效基因CslF6CslF6基因是β-葡聚糖合成中的主效基因。化学诱变实验表明,在大麦β-葡聚糖缺失突变体中, 存在HvCslF6基因突变[40];敲除实验表明,HvCslF3、HvCslF9和HvCslH1基因敲除后,并不影响大麦籽粒β-葡聚糖含量,而敲除HvCslF6基因后,大麦籽粒中检测不到β-葡聚糖含量[41];HvCslF6基因过表达后,大麦籽粒β-葡聚糖含量平均增长45%,而过表达HvCslH1基因后,大麦籽粒β-葡聚糖含量无显著变化[18, 42];在转录水平上,大麦籽粒CslF6基因的转录峰值时期与胚芽鞘中β-葡聚糖含量达到峰值的时期相对应[32, 43]。水稻和小麦(Triticum aestivum)中的CslF6基因研究也得到了相似结果[44–46]。CslF亚家族其他成员在β-葡聚糖合成过程中的功能尚不明确。
2.2 生物信息学分析 2.2.1 系统发育关系目前的观点认为,β-葡聚糖基因家族出现在约130~140百万年前,早于单双子叶植物的分化时间[7]。Yin等[37]认为β-葡聚糖合成酶基因家族的祖先存在于陆地植物的共同祖先中,该家族的分化时间要早于陆地植物的分化。在陆地植物演化过程中,CslH和CslJ亚家族在苔藓中丢失,仅残余序列片段。CslF、CslH和CslJ亚家族的远缘关系说明,β-葡聚糖合成酶基因家族来源于多次独立的亚家族分化事件。
Beta-葡聚糖合成酶基因家族成员位于系统发育树的不同分支:CslF亚家族仅分布于部分鸭跖草类(Commelid)植物的CesA/CslD/CslF分支,可能是由CslD亚家族演化而来。而CslH和CslJ亚家族在单子叶植物中广泛存在,其中CslH亚家族是CslB亚家族(真双子叶植物特有)的姊妹分支,CslJ亚家族是CslM亚家族(广义双子叶植物特有)的姊妹分支,分别位于CslB/CslH和CslE/CslG/CslJ/CslM分支[7]。
CslF亚家族包括CslF1~CslF13共13个成员, 成员间的演化关系较为清晰。CslF6与CslF7基因较早分化出来,其后通过复制事件产生CslF基因簇,该基因簇包含除CslF6与CslF7外的大部分CslF亚家族基因,并在禾本科物种中高度保守[47]。共线性分析的结果表明,CslF基因簇来源于串联重复事件[48]。复制事件后的基因后代容易发生功能分化,如CslF3和CslF10基因,其在系统发育树上组成单独的分支,它们可能与木葡聚糖的合成有关[49]。CslH和CslJ亚家族成员较少,缺少系统发育关系的专门研究。
Beta-葡聚糖合成酶基因家族成员普遍受到负选择压力,表明作为细胞壁组成成分,β-葡聚糖在演化过程中具有保守性[47]。CslF亚家族部分基因受到显著的正选择压力,如CslF6、CslF7、CslF3和CslF10基因,受到正选择压力的基因可能与植物的竞争优势有关。CslH和CslJ亚家族受到的负选择压力稍弱,存在极高的核苷酸替换速率[47]。目前的研究多数是在大跨度进化框架下,探究β-葡聚糖合成酶基因家族的系统发育关系,而着眼于作物及野生近缘种之间精细差别的研究较少。
2.2.2 表达模式CslF亚家族基因主要在生长旺盛的幼嫩组织中表达。CslF基因簇内基因的表达模式并不相同, 例如CslF3基因在胚芽鞘组织中表达水平较高,而CslF4基因在叶基部组织中表达水平较高[32–33]。CslF6基因在作物的根尖、叶基部、籽粒等幼嫩组织中都具有较高的表达水平,而CslF7基因的表达部位尚不清楚[48]。
CslH亚家族在叶尖等成熟组织中具有较高的表达水平,可能与细胞停止生长后,化合物的沉积过程有关[50–51]。二穗短柄草(Brachypodium distachyon)籽粒萌发过程中,BdCslH基因的表达水平与BdCslF6基因相似,而大麦和小麦萌发籽粒中并未检测到CslH基因的表达[52]。CslJ亚家族表达模式的研究不多,仅在燕麦籽粒中检测到较高的表达水平[39]。对于β-葡聚糖合成酶基因家族成员之间的差异表达模式,一种观点认为与组织间β-葡聚糖溶解性差异有关[51],其他观点则认为可能是基因家族扩张后功能分化的结果[53]。
Beta-葡聚糖合成酶基因的表达水平受环境因素影响,尤其是光照。当光照增加时,玉米(Zea mays)胚轴中ZmCslF5的表达水平增加,而ZmCslF6的表达水平降低,推测与光照对生长素的调节有关[54–55]。Zhang等[39]在燕麦β-葡聚糖合成酶基因家族的启动子区域(约2 000 bp)中检测到大量的光响应元件,并且叶片中AsCslF6基因的表达水平随光照强度上升而增加, β-葡聚糖含量也随之增加[39]。除光照强度之外, 其他环境因素对作物β-葡聚糖含量的影响尚未得到评估。
3 Beta-葡聚糖合成酶Beta-葡聚糖合成酶氨基酸序列主要包含有: (1) 亲水催化功能域(PF00535);(2) 催化功能基序(D、D×D、D和Q××RW),其中Q是谷胺酰胺,R是精氨酸,W是色氨酸,×表示任意氨基酸;(3) 存在多个跨膜结构域(transmembrane helices, TMH)[56] (图 2)。
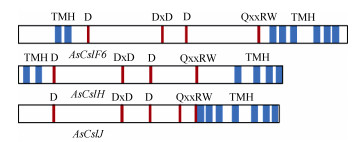
|
图 2 Beta-葡聚糖合成酶蛋白二级结构的比较(根据Zhang等[39]重绘) Fig. 2 Comparison of secondary structure of β-glucan synthase protein (Redrawn from Zhang et al.[39]) |
Beta-葡聚糖合成酶存在两种催化活性,分别合成β-(1, 4)葡萄糖苷键和β-(1, 3)葡萄糖苷键。Fincher[57]认为,β-葡聚糖合成酶仅具有添加β-(1, 3)葡萄糖苷键的活性,β-(1, 4)葡萄糖苷键来源于异源表达实验中,植物细胞自身存在的β-(1, 4)葡聚糖合酶。Kim等[56]选择无β-(1, 4)葡聚糖合酶的毕赤酵母作为材料,异源表达BdCslF6基因后成功检测到β-葡聚糖的合成。据此认为,β-葡聚糖合成酶具有催化两种糖苷键合成的活性。
Beta-葡聚糖合成酶催化β-葡聚糖合成的具体过程已得到初步研究。Schwerdt等[47]提出大麦HvCSLF6酶蛋白的同源模型,认为在合成过程中存在明显的跨膜运输和催化中心。Kim等[56]进一步证明BdCSLF6酶蛋白的催化功能域、N-末端和C-末端都暴露在胞质中。并推测β-葡聚糖的合成过程为:胞质内的合成底物尿苷二磷酸葡萄糖(UDP-glucose, UDP-Glc),通过暴露在胞质中的催化结构域,不断被添加到葡聚糖链上, 再穿过跨膜螺旋形成的膜孔,被运输到膜的另一侧。而β-葡聚糖合成酶如何调控β-(1, 3)和β-(1, 4)糖苷键合成比例,以及克服合成过程中空间扭转的合成机制问题,还需更多的研究来解答。
3.2 亚细胞定位Beta-葡聚糖合成的亚细胞定位争议未决。纤维素[β-(1, 4)糖苷键链接]和胼胝质[β-(1, 3)糖苷键链接]在质膜合成[10, 58],而果胶和其余非纤维素多糖在高尔基体合成[59]。早期学者根据体外合成实验,认为高尔基体是细胞内的β-葡聚糖合成部位[60–61]。随着免疫细胞学方法应用[62],新的研究结果并不支持β-葡聚糖在高尔基体上合成,小麦糊粉层细胞中的高尔基体仅有少量的免疫细胞学标记信号[63],大麦胚芽鞘细胞的高尔基体上也检测不到明显信号[64]。2010年,Carpita等[65]提出,β-葡聚糖的合成运输是一个动态的过程,β-葡聚糖在高尔基体中合成,经由囊泡运输到质膜,最终在细胞壁上沉积。
Beta-葡聚糖合成酶的亚细胞定位也存在不同的观点。Doblin等[35]将CSLH蛋白定位于高尔基体囊泡和内质网上。Kim等[56]将CSLF6蛋白定位于高尔基体,并观察到通过分泌途径的管腔转运。Wilson等[66]在内质网、高尔基体、分泌囊泡和质膜中都发现了CSLF6和CSLH蛋白信号,分布丰度不同。有学者认为,在β-葡聚糖分泌和运输过程中,伴随着β-葡聚糖合成酶蛋白从高尔基体到内质网的膜系统转移,这也是导致β-葡聚糖合成酶蛋白亚细胞定位争议未决的根本原因[67]。
4 作物β-葡聚糖定向育种Beta-葡聚糖合成酶蛋白上的特定氨基酸位点突变后,可显著影响β-葡聚糖含量和精细结构,或可成为作物β-葡聚糖定向育种的一种手段。通过构建CSLF6片段嵌合体蛋白,并分析其在本氏烟草(Nicotiana benthamiana)叶片瞬时表达的结果, Jobling等[68]报道HVCSLF6中的I757L氨基酸位点突变,可导致β-葡聚糖DP比值明显下降,而在玉米ZMCSLF6中,同一位点突变则会导致DP比值明显上升,实验证明,氨基酸位点突变可能是造成不同作物β-葡聚糖水溶性差异的主要原因。Dimitroff等[69]采用相似的方法,观察到SBCSLF6蛋白上G638D氨基酸位点突变后,会引起β-葡聚糖产物DP比值变小, 且β-葡聚糖含量随之下降。根据β-葡聚糖酶蛋白的三维结构,解析特定氨基酸位点突变对β-葡聚糖含量和精细结构的影响,是育种关注的研究方向。
染色体工程是一种常见的育种手段,通过附加、代换、消减和易位等染色体操作,改变研究对象的染色体组成,定向调整作物遗传特性[70]。通过染色体工程导入异源β-葡聚糖合成酶基因后,如将大麦或山羊草属(Aegilops)物种中包含CslF6基因的染色体转移到小麦中,可观察到籽粒中β-葡聚糖含量的增高[71–72]。
采用荧光原位杂交技术,实现基因在染色体上的精准定位,可应用于作物染色体工程育种。小麦D亚基因组上的TaCslF6_D基因,被认为在籽粒β-葡聚糖合成中的贡献更大,通过导入异源CslF6基因,替换AB亚基因组上的TaCslF6基因后,小麦籽粒中的β-葡聚糖含量明显增加[73]。Fogarty等[74]报道在β-葡聚糖产量高的燕麦品种中,AsCslF6_C基因表达较低,而在β-葡聚糖含量低的燕麦品种中, 其表达水平较高,推测C基因组的AsCslF6_C基因在β-葡聚糖合成中起负向调节作用,但对于燕麦中CslF6基因的分布,还缺乏染色体定位证据。
物种间的β-葡聚糖含量的差异,是作物β-葡聚糖定向育种研究的基础。燕麦属A、C基因组二倍体物种间存在明显的β-葡聚糖含量差异,大西洋燕麦(Avena atlantica, AA)的β-葡聚糖含量,约为偏肥燕麦(A. ventricosa, CC)的4倍[75]。小麦属近缘种中, 山羊草(Aegilops tauschii, DD)籽粒的β-葡聚糖含量, 约为乌拉图小麦(Triticum urartu, AA)的4倍[76]。Garcia-Gimenez等[77]报道,β-葡聚糖在籽粒发育过程中,表现为明显的基因型依赖性积累。并且无论在何种生长条件下,高β-葡聚糖品种籽粒的β-葡聚糖含量, 均高于在同等条件下的低β-葡聚糖品种[78]。因此,研究β-葡聚糖含量形成的遗传机制,具有十分重要的育种价值。
5 展望Beta-葡聚糖可以显著降低食品的血糖生成指数,改善人体脂肪堆积情况,对威胁人类健康的高血脂症和高血糖具有防治作用。燕麦中β-葡聚糖含量较高,且具有良好的溶解度,是食物中理想的β-葡聚糖来源。近年来在功能食品及化妆品领域,燕麦β-葡聚糖相关的产品已形成一定的市场规模,而燕麦β-葡聚糖育种仍然停留在杂交育种阶段,无法满足市场需求。通过染色体工程导入异源β-葡聚糖合成酶基因,可显著改变作物籽粒中β-葡聚糖含量。其中β-葡聚糖合成酶基因在染色体上的定位, 是检测异源基因是否成功导入的关键。设计特异性探针,完成主效基因CslF6的染色体定位,是提高燕麦籽粒中β-葡聚糖含量的突破口,可推动染色体工程在燕麦β-葡聚糖定向育种中的应用。
随着燕麦属物种基因组数据积累,通过比较基因组学方法,探讨β-葡聚糖合成酶基因家族的基因拷贝数目、染色体分布模式等特征与β-葡聚糖含量的相关性,为在基因水平解释物种间β-葡聚糖含量差异形成的原因提供可能性,也为燕麦属野生种质资源的开发利用提供科技支撑。结合作物不同发育时期的转录组数据,可筛选出与β-葡聚糖合成酶基因家族成员共表达的基因,确定关键的调控基因或转录因子,来构建β-葡聚糖合成的调控网络。
本文综述了β-葡聚糖的化学特性、功能应用, β-葡聚糖合成酶基因家族成员的系统发育关系、表达模式、β-葡聚糖合成酶的亚细胞定位,及作物β-葡聚糖定向育种中的研究进展。并提出开发燕麦属野生种质资源,促进染色体工程在作物β-葡聚糖定向育种中的应用,是厘清β-葡聚糖合成酶基因家族的调控网络的研究方向。
| [1] |
SHEWRY P R, WAN Y F, HAWKESFORD M J, et al. Spatial distribution of functional components in the starchy endosperm of wheat grains[J]. J Cereal Sci, 2020, 91: 102869. DOI:10.1016/j.jcs.2019.102869 |
| [2] |
STINARD P S, NEVINS D J. Distribution of noncellulosic β-D-glucans in grasses and other monocots[J]. Phytochemistry, 1980, 19(7): 1467-1468. DOI:10.1016/0031-9422(80)80196-8 |
| [3] |
NIRMALA PRASADI P V, JOYE I J. Dietary fibre from whole grains and their benefits on metabolic health[J]. Nutrients, 2020, 12(10): 3045. DOI:10.3390/nu12103045 |
| [4] |
SHVACHKO N A, LOSKUTOV I G, SEMILET T V, et al. Bioactive components in oat and barley grain as a promising breeding trend for functional food production[J]. Molecules, 2021, 26(8): 2260. DOI:10.3390/molecules26082260 |
| [5] |
HAZEN S P, SCOTT-CRAIG J S, WALTON J D. Cellulose synthaselike genes of rice[J]. Plant Physiol, 2002, 128(2): 336-340. DOI:10.1104/pp.010875 |
| [6] |
BURTON R A, WILSON S M, HRMOVA M, et al. Cellulose synthase-like CslF genes mediate the synthesis of cell wall (1, 3;1, 4)-β-D-glucans[J]. Science, 2006, 311(5769): 1940-1942. DOI:10.1126/science.1122975 |
| [7] |
LITTLE A, SCHWERDT J G, SHIRLEY N J, et al. Revised phylogeny of the Cellulose synthase gene superfamily: Insights into cell wall evolution[J]. Plant Physiol, 2018, 177(3): 1124-1141. DOI:10.1104/pp.17.01718 |
| [8] |
STAUDTE R G, WOODWARD J R, FINCHER G B, et al. Watersoluble (1→3), (1→4)-β-D-glucans from barley (Hordeum vulgare) endosperm: Ⅲ. Distribution of cellotriosyl and cellotetraosyl residues[J]. Carbohydr Polym, 1983, 3(4): 299-312. DOI:10.1016/0144-8617(83)90027-9 |
| [9] |
MIKKELSEN M S, JESPERSEN B M, LARSEN F H, et al. Molecular structure of large-scale extracted β-glucan from barley and oat: Identification of a significantly changed block structure in a high β-glucan barley mutant[J]. Food Chem, 2013, 136(1): 130-138. DOI:10.1016/j.foodchem.2012.07.097 |
| [10] |
MCFARLANE H E, DÖRING A, PERSSON S. The cell biology of cellulose synthesis[J]. Annu Rev Plant Biol, 2014, 65: 69-94. DOI:10.1146/annurev-arplant-050213-040240 |
| [11] |
BULONE V, SCHWERDT J G, FINCHER G B. Co-evolution of enzymes involved in plant cell wall metabolism in the grasses[J]. Front Plant Sci, 2019, 10: 1009. DOI:10.3389/fpls.2019.01009 |
| [12] |
CHANG S C, SALDIVAR R K, LIANG P H, et al. Structures, biosynthesis, and physiological functions of (1, 3;1, 4)-β-D-glucans[J]. Cells, 2021, 10(3): 510. DOI:10.3390/cells10030510 |
| [13] |
LAZARIDOU A, BILIADERIS C G. Molecular aspects of cereal β-glucan functionality: Physical properties, technological applications and physiological effects[J]. J Cereal Sci, 2007, 46(2): 101-118. DOI:10.1016/j.jcs.2007.05.003 |
| [14] |
KIM H J, WHITE P J. Impact of the molecular weight, viscosity, and solubility of β-glucan on in vitro oat starch digestibility[J]. J Agric Food Chem, 2013, 61(13): 3270-3277. DOI:10.1021/jf305348j |
| [15] |
KIEMLE S N, ZHANG X, ESKER A R, et al. Role of (1, 3)(1, 4)-β-glucan in cell walls: Interaction with cellulose[J]. Biomacromolecules, 2014, 15(5): 1727-1736. DOI:10.1021/bm5001247 |
| [16] |
BAIN M, VAN DE MEENE A, COSTA R, et al. Characterisation of cellulose synthase like F6 (CslF6) mutants shows altered carbon metabolism in β-D-(1, 3;1, 4)-glucan deficient grain in Brachypodium distachyon[J]. Front Plant Sci, 2021, 11: 602850. DOI:10.3389/fpls.2020.602850 |
| [17] |
MORRALL P, BRIGGS D E. Changes in cell wall polysaccharides of germinating barley grains[J]. Phytochemistry, 1978, 17(9): 1495-1502. DOI:10.1016/S0031-9422(00)94628-4 |
| [18] |
BURTON R A, COLLINS H M, KIBBLE N A J, et al. Over-expression of specific HvCslF cellulose synthase-like genes in transgenic barley increases the levels of cell wall (1, 3;1, 4)-β-D-glucans and alters their fine structure[J]. Plant Biotechnol J, 2011, 9(2): 117-135. DOI:10.1111/j.1467-7652.2010.00532.x |
| [19] |
BURTON R A, FINCHER G B. Current challenges in cell wall biology in the cereals and grasses[J]. Front Plant Sci, 2012, 3: 130. DOI:10.3389/fpls.2012.00130 |
| [20] |
SCHUSTER J, BENINCÁ G, VITORAZZI R, et al. Effects of oats on lipid profile, insulin resistance and weight loss[J]. Nutr Hosp, 2015, 32(5): 2111-2116. DOI:10.3305/nh.2015.32.5.9590 |
| [21] |
YANG C J, CHEN M S, DAI T T, et al. Research advances in functional properties and application of oat β-glucan[J]. J Chin Inst Food Sci Technol, 2021, 21(6): 301-311. 杨成峻, 陈明舜, 戴涛涛, 等. 燕麦β-葡聚糖功能与应用研究进展[J]. 中国食品学报, 2021, 21(6): 301-311. DOI:10.16429/j.1009-7848.2021.06.036 |
| [22] |
JOYCE S A, KAMIL A, FLEIGE L, et al. The cholesterol-lowering effect of oats and oat beta glucan: Modes of action and potential role of bile acids and the microbiome[J]. Front Nutr, 2019, 6: 171. DOI:10.3389/fnut.2019.00171 |
| [23] |
PAN W, HAO S S, ZHENG M X, et al. Oat-derived β-glucans induced trained immunity through metabolic reprogramming[J]. Inflammation, 2020, 43(4): 1323-1336. DOI:10.1007/s10753-020-01211-2 |
| [24] |
CHEN O, MAH E, DIOUM E, et al. The role of oat nutrients in the immune system: A narrative review[J]. Nutrients, 2021, 13(4): 1048. DOI:10.3390/nu13041048 |
| [25] |
CHANG H C, HUANG C N, YEH D M, et al. Oat prevents obesity and abdominal fat distribution, and improves liver function in humans[J]. Plant Foods Hum Nutr, 2013, 68(1): 18-23. DOI:10.1007/s11130-013-0336-2 |
| [26] |
ZHU F M, DU B, XU B J. A critical review on production and industrial applications of beta-glucans[J]. Food Hydrocoll, 2016, 52: 275-288. DOI:10.1016/j.foodhyd.2015.07.003 |
| [27] |
FU H, WU D, BING X, et al. Study on the skin care effect of oat β-glucan extracted by bidirectional fermentation[J]. China Surfactant Deterg Cosmet, 2021, 51(4): 324-330. 付豪, 吴迪, 邴雪, 等. 双向发酵提取燕麦β-葡聚糖的护肤功效研究[J]. 日用化学工业, 2021, 51(4): 324-330. DOI:10.3969/j.issn.1001-1803.2021.04.010 |
| [28] |
RIBEIRO D M L, CARVALHO JÚNIOR A R, VALE DE MACEDO G H R, et al. Polysaccharide-based formulations for healing of skinrelated wound infections: Lessons from animal models and clinical trials[J]. Biomolecules, 2019, 10(1): 63. DOI:10.3390/biom10010063 |
| [29] |
GUPTA M, ABU-GHANNAM N, GALLAGHAR E. Barley for brewing: Characteristic changes during malting, brewing and applications of its by-products[J]. Compr Rev Food Sci Food Saf, 2010, 9(3): 318-328. DOI:10.1111/j.1541-4337.2010.00112.x |
| [30] |
BRENNAN C S, CLEARY L J. The potential use of cereal (1→3;1→4)-β-D-glucans as functional food ingredients[J]. J Cereal Sci, 2005, 42(1): 1-13. DOI:10.1016/j.jcs.2005.01.002 |
| [31] |
RICHMOND T A, SOMERVILLE C R. The cellulose synthase superfamily[J]. Plant Physiol, 2000, 124(2): 495-498. DOI:10.1104/pp.124.2.495 |
| [32] |
BURTON R A, JOBLING S A, HARVEY A J, et al. The genetics and transcriptional profiles of the cellulose synthase-like HvCslF gene family in barley[J]. Plant Physiol, 2008, 146(4): 1821-1833. DOI:10.1104/pp.107.114694 |
| [33] |
SCHREIBER M, WRIGHT F, MACKENZIE K, et al. The barley genome sequence assembly reveals three additional members of the CslF (1, 3;1, 4)-β-glucan synthase gene family[J]. PLoS One, 2014, 9(3): e90888. DOI:10.1371/journal.pone.0090888 |
| [34] |
DOBLIN M S, PETTOLINO F A, WILSON S M, et al. A barley cellulose synthase-like CslH gene mediates (1, 3;1, 4)-beta-D-glucan synthesis in transgenic Arabidopsis[J]. Proc Natl Acad Sci USA, 2009, 106(14): 5996-6001. DOI:10.1073/pnas.0902019106 |
| [35] |
WU B, ZHANG Z W. Cloning and analysis of β-glucan synthase gene AsCSLH in Avena sativa L.[J]. Acta Agron Sin, 2011, 37(4): 723-728. 吴斌, 张宗文. 燕麦葡聚糖合酶基因AsCSLH的克隆及特征分析[J]. 作物学报, 2011, 37(4): 723-728. DOI:10.3724/SP.J.1006.2011.00723 |
| [36] |
FARROKHI N, BURTON R A, BROWNFIELD L, et al. Plant cell wall biosynthesis: Genetic, biochemical and functional genomics approaches to the identification of key genes[J]. Plant Biotechnol J, 2006, 4(2): 145-167. DOI:10.1111/j.1467-7652.2005.00169.x |
| [37] |
YIN Y B, HUANG J L, XU Y. The cellulose synthase superfamily in fully sequenced plants and algae[J]. BMC Plant Biol, 2009, 9(1): 99. DOI:10.1186/1471-2229-9-99 |
| [38] |
NEWELL M A, ASORO F G, SCOTT M P, et al. Genome-wide association study for oat (Avena sativa L.) beta-glucan concentration using germplasm of worldwide origin[J]. Theor Appl Genet, 2012, 125(8): 1687-1696. DOI:10.1007/s00122-012-1945-0 |
| [39] |
ZHANG J, YAN L, LIU M X, et al. Analysis of β-D-glucan biosynthetic genes in oat reveals glucan synthesis regulation by light[J]. Ann Bot, 2021, 127(3): 371-380. DOI:10.1093/aob/mcaa185 |
| [40] |
TAKETA S, YUO T, TONOOKA T, et al. Functional characterization of barley betaglucanless mutants demonstrates a unique role for CslF6 in (1, 3;1, 4)-β-D-glucan biosynthesis[J]. J Exp Bot, 2012, 63(1): 381-392. DOI:10.1093/jxb/err285 |
| [41] |
GARCIA-GIMENEZ G, BARAKATE A, SMITH P, et al. Targeted mutation of barley (1, 3;1, 4)-β-glucan synthases reveals complex relationships between the storage and cell wall polysaccharide content[J]. Plant J, 2020, 104(4): 1009-1022. DOI:10.1111/tpj.14977 |
| [42] |
LIM W L, COLLINS H M, SINGH R R, et al. Method for hull-less barley transformation and manipulation of grain mixed-linkage betaglucan[J]. J Integr Plant Biol, 2018, 60(5): 382-396. DOI:10.1111/jipb.12625 |
| [43] |
GIBEAUT D M, PAULY M, BACIC A, et al. Changes in cell wall polysaccharides in developing barley (Hordeum vulgare) coleoptiles[J]. Planta, 2005, 221(5): 729-738. DOI:10.1007/s00425-005-1481-0 |
| [44] |
NEMETH C, FREEMAN J, JONES H D, et al. Down-regulation of the CSLF6 gene results in decreased (1, 3;1, 4)-β-D-glucan in endosperm of wheat[J]. Plant Physiol, 2010, 152(3): 1209-1218. DOI:10.1104/pp.109.151712 |
| [45] |
VEGA-SÁNCHEZ M E, VERHERTBRUGGEN Y, CHRISTENSEN U, et al. Loss of cellulose synthase-like F6 function affects mixed-linkage glucan deposition, cell wall mechanical properties, and defense responses in vegetative tissues of rice[J]. Plant Physiol, 2012, 159(1): 56-69. DOI:10.1104/pp.112.195495 |
| [46] |
MARCOTULI I, COLASUONNO P, BLANCO A, et al. Expression analysis of cellulose synthase-like genes in durum wheat[J]. Sci Rep, 2018, 8(1): 15675. DOI:10.1038/s41598-018-34013-6 |
| [47] |
SCHWERDT J G, MACKENZIE K, WRIGHT F, et al. Evolutionary dynamics of the cellulose synthase gene superfamily in grasses[J]. Plant Physiol, 2015, 168(3): 968-983. DOI:10.1104/pp.15.00140 |
| [48] |
WANG L Q, GUO K, LI Y, et al. Expression profiling and integrative analysis of the CESA/CSL superfamily in rice[J]. BMC Plant Biol, 2010, 10(1): 282. DOI:10.1186/1471-2229-10-282 |
| [49] |
LITTLE A, LAHNSTEIN J, JEFFERY D W, et al. A novel (1, 4)-β-linked glucoxylan is synthesized by members of the cellulose synthaselike F gene family in land plants[J]. ACS Cent Sci, 2019, 5(1): 73-84. DOI:10.1021/acscentsci.8b00568 |
| [50] |
ERMAWAR R A, COLLINS H M, BYRT C S, et al. Distribution, structure and biosynthetic gene families of (1, 3;1, 4)-β-glucan in Sorghum bicolor[J]. J Integr Plant Biol, 2015, 57(4): 429-445. DOI:10.1111/jipb.12338 |
| [51] |
ERMAWAR R A, COLLINS H M, BYRT C S, et al. Genetics and physiology of cell wall polysaccharides in the model C4 grass, Setaria viridis spp[J]. BMC Plant Biol, 2015, 15(1): 236. DOI:10.1186/s12870-015-0624-0 |
| [52] |
CHRISTENSEN U, ALONSO-SIMON A, SCHELLER H V, et al. Characterization of the primary cell walls of seedlings of Brachypodium distachyon: A potential model plant for temperate grasses[J]. Phytochemistry, 2010, 71(1): 62-69. DOI:10.1016/j.phytochem.2009.09.019 |
| [53] |
ZHANG G B, GE C X, XU P P, et al. The reference genome of Miscanthus floridulus illuminates the evolution of Saccharinae[J]. Nat Plants, 2021, 7(5): 608-618. DOI:10.1038/s41477-021-00908-y |
| [54] |
VAN ERP H, WALTON J D. Regulation of the cellulose synthase-like gene family by light in the maize mesocotyl[J]. Planta, 2009, 229(4): 885-897. DOI:10.1007/s00425-008-0881-3 |
| [55] |
DARAS G, TEMPLALEXIS D, AVGERI F, et al. Updating insights into the catalytic domain properties of plant cellulose synthase (CesA) and cellulose synthase-like (Csl) proteins[J]. Molecules, 2021, 26(14): 4335. DOI:10.3390/molecules26144335 |
| [56] |
KIM S J, ZEMELIS S, KEEGSTRA K, et al. The cytoplasmic localization of the catalytic site of CSLF6 supports a channeling model for the biosynthesis of mixed-linkage glucan[J]. Plant J, 2015, 81(4): 537-547. DOI:10.1111/tpj.12748 |
| [57] |
FINCHER G B. Revolutionary times in our understanding of cell wall biosynthesis and remodeling in the grasses[J]. Plant Physiol, 2009, 149(1): 27-37. DOI:10.1104/pp.108.130096 |
| [58] |
SCHELLER H V, ULVSKOV P. Hemicelluloses[J]. Annu Rev Plant Biol, 2010, 61: 263-289. DOI:10.1146/annurev-arplant-042809-112315 |
| [59] |
ATMODJO M A, HAO Z Y, MOHNEN D. Evolving views of pectin biosynthesis[J]. Annu Rev Plant Biol, 2013, 64: 747-779. DOI:10.1146/annurev-arplant-042811-105534 |
| [60] |
GIBEAUT D M, CARPITA N C. Synthesis of (1→3), (1→4)-beta-D-glucan in the golgi apparatus of maize coleoptiles[J]. Proc Natl Acad Sci USA, 1993, 90(9): 3850-3854. DOI:10.1073/pnas.90.9.3850 |
| [61] |
URBANOWICZ B R, RAYON C, CARPITA N C. Topology of the maize mixed linkage (1→3), (1→4)-β-D-glucan synthase at the golgi membrane[J]. Plant Physiol, 2004, 134(2): 758-768. DOI:10.1104/pp.103.032011 |
| [62] |
MEIKLE P J, HOOGENRAAD N J, BONIG I, et al. A (1→3, 1→4)-β-glucan-specific monoclonal antibody and its use in the quantitation and immunocytochemical location of (1→3, 1→4)-β-glucans[J]. Plant J, 1994, 5(1): 1-9. DOI:10.1046/j.1365-313x.1994.5010001.x |
| [63] |
PHILIPPE S, SAULNIER L, GUILLON F. Arabinoxylan and (1→3), (1→4)-β-glucan deposition in cell walls during wheat endosperm development[J]. Planta, 2006, 224(2): 449-461. DOI:10.1007/s00425-005-0209-5 |
| [64] |
WILSON S M, BURTON R A, DOBLIN M S, et al. Temporal and spatial appearance of wall polysaccharides during cellularization of barley (Hordeum vulgare) endosperm[J]. Planta, 2006, 224(3): 655-667. DOI:10.1007/s00425-006-0244-x |
| [65] |
CARPITA N C, MCCANN M C. The maize mixed-linkage (1→3), (1→4)-β-D-glucan polysaccharide is synthesized at the golgi membrane[J]. Plant Physiol, 2010, 153(3): 1362-1371. DOI:10.1104/pp.110.156158 |
| [66] |
WILSON S M, HO Y Y, LAMPUGNANI E R, et al. Determining the subcellular location of synthesis and assembly of the cell wall polysaccharide (1, 3;1, 4)-β-D-glucan in grasses[J]. Plant Cell, 2015, 27(3): 754-771. DOI:10.1105/tpc.114.135970 |
| [67] |
KIM S J, ZEMELIS-DURFEE S, JENSEN J K, et al. In the grass species Brachypodium distachyon, the production of mixed-linkage (1, 3;1, 4)-β-glucan (MLG) occurs in the golgi apparatus[J]. Plant J, 2018, 93(6): 1062-1075. DOI:10.1111/tpj.13830 |
| [68] |
JOBLING S A. Membrane pore architecture of the CslF6 protein controls (1-3, 1-4)-β-glucan structure[J]. Sci Adv, 2015, 1(5): e1500069. DOI:10.1126/sciadv.1500069 |
| [69] |
DIMITROFF G, LITTLE A, LAHNSTEIN J, et al. (1, 3;1, 4)-β-glucan biosynthesis by the CSLF6 enzyme: Position and flexibility of catalytic residues influence product fine structure[J]. Biochemistry, 2016, 55(13): 2054-2061. DOI:10.1021/acs.biochem.5b01384 |
| [70] |
YANG X Y, SU H D, ZHANG M Z, et al. Polyploidization and domestication[J]. Sci Sin Vitae, 2021, 51(10): 1457-1466. 杨学勇, 苏汉东, 张梦卓, 等. 多倍化和驯化研究进展与展望[J]. 中国科学: 生命科学, 2021, 51(10): 1457-1466. DOI:10.1360/SSV-2021-0220 |
| [71] |
RAKSZEGI M, MOLNÁR I, LOVEGROVE A, et al. Addition of Aegilops U and M chromosomes affects protein and dietary fiber content of wholemeal wheat flour[J]. Front Plant Sci, 2017, 8: 1529. DOI:10.3389/fpls.2017.01529 |
| [72] |
DANILOVA T V, FRIEBE B, GILL B S, et al. Development of a complete set of wheat-barley group-7 robertsonian translocation chromosomes conferring an increased content of β-glucan[J]. Theor Appl Genet, 2018, 131(2): 377-388. DOI:10.1007/s00122-017-3008-z |
| [73] |
DANILOVA T V, POLAND J, FRIEBE B. Production of a complete set of wheat-barley group-7 chromosome recombinants with increased grain β-glucan content[J]. Theor Appl Genet, 2019, 132(11): 3129-3141. DOI:10.1007/s00122-019-03411-3 |
| [74] |
FOGARTY M C, SMITH S M, SHERIDAN J L, et al. Identification of mixed linkage β-glucan quantitative trait loci and evaluation of AsCslF6 homoeologs in hexaploid oat[J]. Crop Sci, 2020, 60(2): 914-933. DOI:10.1002/csc2.20015 |
| [75] |
TANG X Q. Determination of β-glucan content and CSLH gene cloning among Avena species[D]. Ya'an: Sichuan Agricultural University, 2014. 唐雪琴. 燕麦属物种β-葡聚糖含量测定及CSLH基因克隆[D]. 雅安: 四川农业大学, 2014. |
| [76] |
MARCOTULI I, COLASUONNO P, CUTILLO S, et al. β-glucan content in a panel of Triticum and Aegilops genotypes[J]. Genet Resour Crop Evol, 2019, 66(4): 897-907. DOI:10.1007/s10722-019-00753-1 |
| [77] |
GARCIA-GIMENEZ G, RUSSELL J, AUBERT M K, et al. Barley grain (1, 3;1, 4)-β-glucan content: Effects of transcript and sequence variation in genes encoding the corresponding synthase and endohydrolase enzymes[J]. Sci Rep, 2019, 9(1): 17250. DOI:10.1038/s41598-019-53798-8 |
| [78] |
DOEHLERT D C, MCMULLEN M S, HAMMOND J J. Genotypic and environmental effects on grain yield and quality of oat grown in North Dakota[J]. Crop Sci, 2001, 41(4): 1066-1072. DOI:10.2135/cropsci2001.4141066x |
 2023, Vol. 31
2023, Vol. 31


