光合作用是绿色植物利用光能,把CO2和H2O转化为储存能量的有机物,同时释放O2的过程。光合作用能为整个生命系统提供物质和能量。光呼吸伴随着光合作用而进行,绿色植物、藻类和蓝细菌利用光能并在O2的参与下将有机物分解为CO2和H2O,该过程即为光呼吸[1–3]。每年大约有29 Gt的光合产物以CO2形式被重新释放到大气中[4–5]。光呼吸的产生是因为1,5-二磷酸核酮糖羧化酶/加氧酶Rubisco具有双功能酶活性,1,5-二磷酸核酮糖在Rubisco的催化下既可进行羧化反应又可进行加氧反应,而加氧或者羧化反应的程度取决于CO2和O2浓度的比值,高比值有利于羧化反应,低比值有利于加氧反应[6]。RuBP的羧化结果是产生2分子3-磷酸甘油酸,然后进入卡尔文循环,而RuBP氧化结果则是产生1分子3-磷酸甘油酸(3-PGA)和1分子2-磷酸乙醇酸(2-PG)。2-PG经过光呼吸代谢途径再部分生成3-PGA回流进入卡尔文循环[7],光呼吸代谢途径能够回收75%的2-PG中的碳,其余25%的碳以CO2的形式在线粒体中释放[8–9]。叶绿体、过氧化物体、线粒体及细胞质中均有光呼吸代谢过程,此过程还涉及多种酶的共同参与[10]。光呼吸的代谢过程见图1。
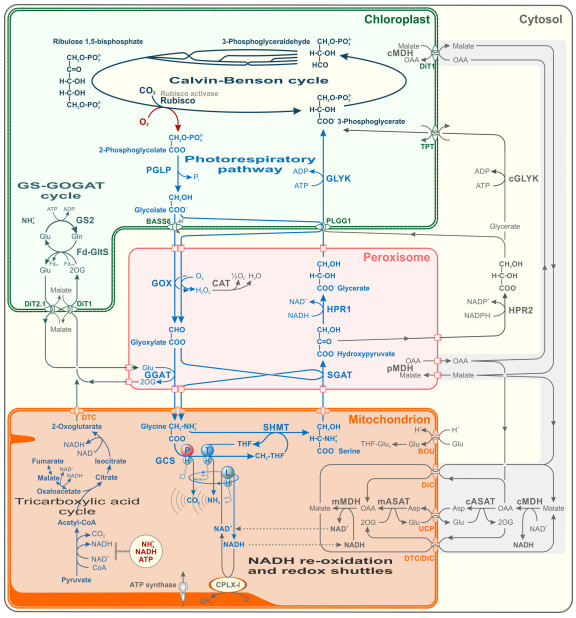
|
图 1 高等植物光呼吸途径及其与卡尔文循环和氨同化的相互联系[11]。2-OG: 2-酮戊二酸; 3-PGA: 3-磷酸甘油酸; 2-PG: 2-磷酸乙醇酸; Ala: 丙氨酸; Asn: 天冬酰胺; ASNS: 天冬酰胺合成酶; Asp: 天冬氨酸; CAT: 过氧化氢酶; Gln: 谷氨酰胺; FD-GOGAT: 铁氧还蛋白依赖性谷氨酸合酶; Glu: 谷氨酸; GDC: 甘氨酸脱羧酶复合物; GLO: 乙醇酸氧化酶; GLYK: 甘油酸3-激酶; GGAT: 谷氨酸:乙醛酸转氨酶; GS2: 谷氨酰胺合成酶; SGAT: 丝氨酸:乙醛酸转氨酶; SHMT: 丝氨酸羟甲基转移酶; Rubisco: 核酮糖1,5-二磷酸羧化/加氧酶; HPR1: 羟基丙酮酸还原酶1。 Fig. 1 Photorespiration pathway in higher plants and their relationship with Calvin cycle and ammonia assimilation[11]. 2-OG: 2-Oxoglutaric acid; 3-PGA: 3-Phosphoglycerate; 2-PG: Phosphoglycolic acid; Ala: Alanine; Asn: Asparagine; ASNS: Asparagine synthetase; Asp: Aspartic acid; CAT: Catalase; Gln: Glutamine; FD-GOGAT: Ferridoxin-dependent glutamate synthase; Glu: Glutamate; GDC: Glycine decarboxylase complex; GLO: Glycolate oxidase; GLYK: Glycerate 3-kinase; GGAT: Glutamate:glyoxylate aminotransferase; GS2: Plastid glutamine synthetase 2; SGAT: Serine:glyoxylate aminotransferase; SHMT: Serine hydroxymethyltransferase; Rubisco: Ribulose 1,5-bisphosphate carboxylase/oxygenase; HPR1: Hydroxypyruvate reductase 1. |
Rubisco在叶绿体中催化RuBP产生2-PG,乙醇酸磷酸酶(PGLP)催化2-PG去磷酸化生成乙醇酸,乙醇酸通过乙醇酸/甘油酸逆向转运蛋白从叶绿体运输至细胞质中,然后进入过氧化物酶体中[12]。在乙醇酸氧化酶(GLO)的催化下,乙醇酸氧化生成乙醛酸并产生H2O2,H2O2被过氧化氢酶(CAT)分解。乙醛酸在谷氨酸:乙醛酸转氨酶(GGAT)和丝氨酸:乙醛酸转氨酶(SGAT)的作用下转化为甘氨酸。甘氨酸从过氧化物酶体转移到线粒体后,由甘氨酸脱羧酶(GDC)和丝氨酸羟甲基转移酶(SHMT)将2个甘氨酸分子催化为1个丝氨酸并释放1分子CO2和NH3。氧化脱羧步骤GDC复合体将NAD+还原成NADH。在SGAT的作用下,丝氨酸从线粒体返回到过氧化物酶体产生羟基丙酮酸(HP)。在羟基丙酮酸还原酶(HPR1)的催化下,HP还原为甘油酸。最后甘油酸返回叶绿体,在甘油酸激酶(GLYK)的作用下,甘油酸磷酸化生成3-磷酸甘油酸(3-PGA),重新返回到卡尔文循环中[13]。
2 光呼吸的生理功能光呼吸的主要生理功能是清除Rubisco加氧反应生成的毒性中间代谢物2-PG并回收部分碳素, 但该过程释放的CO2降低了植物的净光合效率[14–16]。然而值得关注的是,在逆境条件下光呼吸对C3植物的光合作用及产量维持可能具有保护作用[17–18]。此外已知光呼吸还与植物其它多种生物学过程密切相关, C3植物光呼吸减弱后会导致氮素吸收与同化受到抑制[19–20]。Gao等[21]报道拟南芥(Arabidopsis thaliana) cat2突变体中光呼吸生成的H2O2可诱导叶片生长素浓度降低并出现叶片边缘卷曲表型,而Kerchev等[22]报道外源施加生长素类似物可消除cat2突变体因光呼吸H2O2积累而诱发的细胞程序性死亡表型。在拟南芥、烟草(Nicotiana tabacum)和水稻(Oryza sativa)等植物中光呼吸还参与了其非寄主抗性防卫信号以及病原菌诱发的水杨酸信号的转导过程[23–25]。光呼吸不仅在C3植物中具有重要功能, 而且在光呼吸速率相对低的C4植物或蓝藻中也起着不可或缺的作用,因为发现这些低光呼吸生物的突变体同样表现出典型的光呼吸表型[26–27]。总之,关于光呼吸的功能至今虽然仍存在颇多争议, 但似乎已一致认可光呼吸与植物碳代谢、氮同化、能量代谢以及H2O2信号发生等多个代谢过程密切相关[28–29],并且还可能在植物抗逆性和抗病性中发挥重要作用[30–32]。
3 优化光呼吸的3种策略光呼吸是一个耗能的代谢过程,在正常生长条件下,C3植物中可消耗掉净光合产物的25%~30%[7,33], 当遭遇干旱、高温、高光等逆境胁迫时,其损耗可高达50%[34]。鉴于光呼吸是制约光合作用效率的一个关键因子,为提高作物的生产力,目前主要采取了3种改造策略以试图降低光呼吸速率而提高光合效率:第1种是通过酶工程来提高Rubisco的羧化效率,即直接提高Rubisco催化的羧化反应速率或减少其加氧反应速率[35];第2种是通过基因工程与合成生物学技术将某些植物所具备的CO2浓缩机制(CCM)导入到C3植物中,增加Rubisco周围的CO2浓度而抑制光呼吸;第3种是在C3植物中创建光呼吸代谢支路,以降低光呼吸代谢的能量损耗并限制光呼吸。
3.1 分子改造Rubisco由于光呼吸发生的根本原因是CO2和O2竞争性地与Rubisco活性中心结合,同时Rubisco催化RuBP发生羧化/加氧反应,影响着植物光合作用和光呼吸速率[36]。因此Rubisco一直被视为固碳系统改造的首要选择目标,目的是提高其底物选择性和动力学性能。
Rubisco复合体的组装过程复杂,对其改造极具挑战性,尝试用蓝藻和红藻的酶替代植物Rubisco但并没有成功[37]。将编码红藻Rubisco小亚基的基因通过质体转化在烟草叶绿体高效表达后,烟草本身的大亚基却不能与红藻Rubisco小亚基组装成全酶[4]。
最近有报道称将蓝藻的CO2浓缩机制和Rubisco引入高等植物的叶绿体中可以提高植物CO2的固定速率,同时降低光呼吸速率[38]。虽然来源于红藻的Rubisco基因转化至烟草叶绿体并进行了转录表达,但转基因植株缺乏功能性的Rubisco,认为共表达相容性分子伴侣对于红藻Rubisco在植物中的成功组装是必要的[39]。在水稻Rubisco中将RbcS完全替换为高粱RbcS,获得的杂交Rubisco表现出几乎与C4植物类似的催化特性,但与野生型水稻相比,转基因株系中积累的Rubisco水平降低[40]。来自类球红细菌(Rhodobacter sphaeroides)的red-type Rubisco (RsRubisco)可在烟草叶绿体中完成组装, 但其活性只有野生型烟草Rubisco活性的40%, 并且转基因烟草生长缓慢。转入RsRubisco活化酶RsRca后, RsRubisco活性显著提高,转基因烟草的光合效率和生长速率明显高于野生型[41]。因此, 通过改善Rubisco的动力学特性来提高植物的光合效率是一个长期的目标[42–43]。
3.2 提高Rubisco周围CO2浓度另一种策略旨在优化并利用几种自然存在的CO2浓缩机制(CCM)。C4植物、藻类和蓝细菌依赖于自身的CCM可以增加Rubisco附近的CO2浓度,从而增加羧化作用进而抑制加氧作用,该机制可在低CO2条件下提高光合作用的运行效率。蓝细菌及藻类中已进化出的CCM组分包括Rubisco、无机碳转运蛋白周围的羧酶体或蛋白核结构和碳酸酐酶[44–46]。在蓝细菌和一些变形菌中,Rubisco聚集在一组称为羧酶体的微室中[46],这些微室是CCM的一部分,与CO2和HCO3–转运蛋白一起参与反应, 从而提高细胞质中HCO3–含量。其外层由蛋白质壳包围,内含碳酸酐酶、Rubisco蛋白等[46–48]。
目前已经成功将藻类的CCM部分结构导入植物体,这为在植物体内构建功能性CCM奠定了基础[38,49]。将莱茵衣藻CO2浓缩机制(CCM)的成分整合到烟草叶绿体中,转基因株系表现出光合速率可提高约15%,生物量明显增加,并积累了更多的碳水化合物[50]。研究还表明,将蓝细菌β-羧酶体组分CcmM35与Rubisco大、小亚基整合至烟草中,可形成早期羧酶体结构,并在叶绿体基质中形成大分子复合物。当在烟草中瞬时表达多种β-羧酶体蛋白(CcmK2、CcmM、CcmL、CcmO和CcmN等),可形成高度有序的空室类似结构[37],其中,CcmM的SSUL模块可参与Rubisco蛋白聚集过程[51]。在高等植物叶绿体中组装蓝细菌α-羧酶体结构,可提升细胞内碳酸氢盐含量,从而将CO2大量聚集在Rubisco周围[52],促进羧化反应的进行。但目前研究工作也仅限于在烟草叶绿体中合成简化版的羧酶体结构, 在植物叶绿体中完全组装羧酶体和蛋白核结构仍存在一些限制因素。同时只有将Rubisco正确组装到CCM微室,才能确保CCM结构完整性。只有在进一步了解CCM结构、组装机理和功能的基础上完善所导入CCM结构的完整性,才可能最终产生改善光合作用的实效[53]。
在C3作物中引入C4植物CCM系统也可增加Rubisco附近CO2浓度,将玉米磷酸烯醇/丙酮酸羧化酶(PEPC)和丙酮酸磷酸双激酶(PPDK)转入水稻中,虽然这2种酶能够高表达,但是转基因水稻的光合特性没有改变,在C3植物中引入C4植物光合作用酶还需进一步探索[54]。将C4植物光合系统相关酶引入C3植物时,需要考虑其光合过程中生化反应的变化,由于C4植物Kranz结构特殊性及其光合作用相关的关键酶的分布和作用具有显著的时空特异性,所以仅将C4植物光合基因引入C3植物形成单一细胞型C4植物无法真正达到C4植物的高光效功能[5,55–56]。C4植物中Rubisco附近的CO2浓度由叶肉细胞和束鞘细胞之间的代谢活动来提供的, 在叶肉细胞中,CO2首先被磷酸烯醇式丙酮酸(PEP)羧化酶转化为四碳酸,随后被主动运输到维管束鞘细胞中[57]进行脱羧,增加Rubisco附近的CO2浓度。这种C4植物“CO2泵”每固定1 mol CO2需要消耗2个ATP。在水稻中过表达玉米(Zea mays) GOLDEN2-LIKE基因,水稻维管束鞘细胞中光合作用酶得到积累并起作用,表明C4水稻工程的关键一步已完成[58]。将玉米C4途径关键酶ZmPEPC、ZmPPDK、ZmNADP-MDH和ZmNADP-ME的原始基因分别转入水稻中, 转基因植株的相应酶活性增强,但光合速率几乎没有变化。虽然能完成C4光合途径的羧化反应,但不能完成后续的PEP再生构建完整的C4循环[59],水稻等作物的人工C3到C4的转化将会是一项长期的任务[57]。
3.3 创建光呼吸支路通过降低光呼吸的能量需求和将光呼吸释放的CO2重新定位到叶绿体中,可以增强作物光合作用提高产量。构建新型光呼吸支路被认为是提高植物Rubisco固碳能力的有效途径之一。由于光呼吸中间代谢产物2-磷酸乙醇酸与乙醇酸等对细胞具有毒性,因此大部分光呼吸支路的构建都以乙醇酸为底物。作为降低Rubisco氧化作用和自身光呼吸途径效率的替代方法,在C3植物中创建光合CO2浓缩机制一直是光呼吸研究领域关注的热点。
根据时间线对已创建的光呼吸支路予以梳理, 2007年,Kebeish等[60]在拟南芥叶绿体中过表达大肠杆菌乙醇酸脱氢酶(GDH)、乙醛酸聚醛酶(GCL)和羟基丙二酸半醛还原酶(TSR),简称GGT支路, 此支路在叶绿体中将2个乙醇酸转化为甘油酸和CO2, 这样降低了光呼吸代谢ATP需求量,同时避免了NH3的产生,GGT转基因植株在特定培养条件下[光照8 h,光强为100 μmol/(m2·s)]光合速率提高,同时光呼吸降低,生物量显著提高。2012年,Maier等[61]在拟南芥的叶绿体中表达乙醇酸氧化酶、苹果酸合酶和过氧化氢酶(简称GMC支路),此支路能完成乙醇酸代谢循环,不仅在叶绿体中释放CO2, 同时部分避免了NH3的释放。特定培养条件下, 过氧化氢酶活性显著提高的过表达株系表现出光合速率和生物量的增加。然而,GMC途径会比植物自身的光呼吸途径更加耗能[62]。2014年Nolke等[63]在马铃薯(Solanum tuberosum)叶绿体中仅导入大肠杆菌乙醇酸脱氢酶,转基因植株的光合效率、生物量和产量也大幅提升。2015年Dalal[64]在亚麻荠(Camelina sativa)中重复了GGT途径,将3个酶基因导入亚麻荠并定位到叶绿体中,与野生型相比, 转基因植株生长更迅速,提前约1周开花,光合速率和生物量得到提高,油籽产量提高了57%~73%,并降低了光呼吸速率。2016年在马铃薯叶绿体中重复了Kebeish等[60]的GGT支路,支路1导入乙醇酸脱氢酶,命名为synGDH;支路2导入乙醛酸聚醛酶和羟基丙二酸半醛还原酶,命名为synGT。synGDH转基因植株的生物量比野生型和synGT转基因植株增加了11%,虽然synGT转基因植株积累的生物量低于synGDH转基因植株,但还是明显高于野生型。同时synGDH转基因植株的产量分别比野生型和synGT转基因植株增加38%和16%,这说明在植物叶绿体中表达乙醇酸代谢途径是有效的[3]。2019年South等[65]设计了3条光呼吸支路,支路1是重复Kebeish等[60]的GGT支路;支路2是重复Maier等[61]的GMC支路;支路3是将苹果酸合酶(MS)和乙醇酸脱氢酶(CrGDH)基因导入叶绿体中, 利用RNAi技术下调光呼吸途径中乙醇酸转运蛋白(PLGG1)的表达,进而限制乙醇酸的转运量。温室种植条件下,3条支路的植株生物量与野生型相比,支路1提高13%,支路2的无明显提高,支路3增加18%,而同时下调PLGG1的支路3转基因植株的生物量显著增加24%。光合速率与生物量的提高表现一致,田间试验表明,支路3转基因植株的生物量增加25%,同时下调PLGG1的支路3转基因植株生物量增加了40%。
2019年Shen等[66]将水稻乙醇酸氧化酶(GLO)、草酸氧化酶(OXO)和过氧化氢酶(CAT)基因导入水稻叶绿体中,使乙醇酸在叶绿体中完成代谢并释放CO2,简称GOC支路(图2: A),GOC转基因植株比野生型的净光合速率与生物量显著提高。春季大田种植,产量显著提高,但秋季大田种植产量有所下降。GOC支路在分流光呼吸同时可减少碳、氮损耗,是第一次在主要粮食作物水稻中成功创建光呼吸代谢支路,具有重要意义。2020年Wang等[67]将水稻乙醇酸氧化酶(OsGLO1)、大肠杆菌过氧化氢酶(EcCAT)、大肠杆菌乙醛酸聚醛酶(EcGCL)和大肠杆菌羟基丙二酸半醛还原酶(EcTSR)基因(其中EcGCL和EcTSR以融合蛋白形式EcGCL*EcTSR)引入叶绿体中,简称GCGT支路(图2: B)。春季大田种植,转基因植株相比于野生型,净光合速率、生物量及单株产量显著提高。2021年Roell等[68]在拟南芥中导入β-羟基天冬氨酸循环4个核心酶,即天冬氨酸:乙醛酸转氨酶、β-羟基天冬氨酸醛缩酶、β-羟基天冬氨酸脱氢酶和亚氨基琥珀酸还原酶,通过融合过氧化物酶体靶信号(PTS)定位到过氧化物酶体(简称BHAC支路)。β-羟基天冬氨酸循环能不依赖于光呼吸中间产物3-磷酸甘油酸再生或光呼吸前体脱羧, 而是以高效碳、氮和节约能量的方式将光呼吸的代谢产物乙醛酸转化为草酰乙酸,这意味着在C3植物中构建C4循环具有可行性。
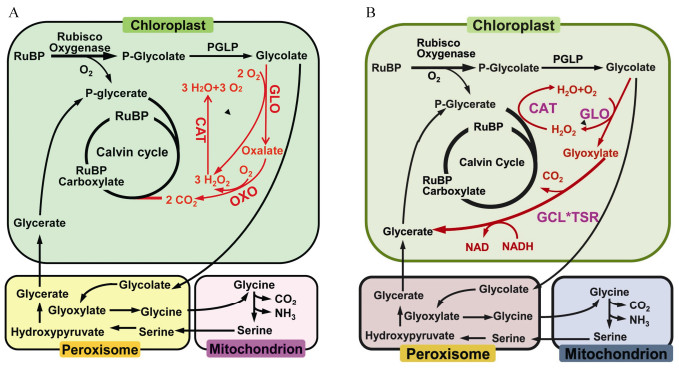
|
图 2 水稻叶绿体中GOC (A)与GCGT (B)光呼吸支路示意图[66–67] Fig. 2 Schematic diagram of photorespiration bypasss of GOC (A) and GCGT (B) in rice chloroplasts[66–67] |
Peterhansel等[62]对光呼吸改造支路代谢途径所消耗能量进行计算,自身光呼吸代谢过程需消耗12.25个ATP,GGT支路需消耗9.25个ATP,GMC支路需消耗15个ATP,GOC支路中没有产生额外还原力,整个支路需要消耗20个ATP,GCGT支路反应过程中也没有还原力的产生,整个支路要消耗11.75个ATP。GGT途径与自身光呼吸代谢相比减少了3个ATP的消耗,降低了光呼吸过程中的能量消耗。GMC支路与GOC支路在能量平衡方面没有优势,其能量消耗还高于自身光呼吸代谢,尤其是GOC高出7.75个ATP。但为何GMC支路与GOC支路转基因植株光合速率与产量仍有明显提高,可能是提高了叶绿体周围的CO2浓度,在叶绿体中形成了CO2浓缩机制所致[13]。
4 展望近年来对于如何进一步降低C3作物光呼吸速率,提高其光合效率以及产量进行了大量研究并取得了一定的进展,随着基因工程、酶工程及合成生物学的快速发展,降低Rubisco氧化反应,创建CO2浓缩机制及光呼吸支路的技术水平将会不断提升。相较于天然固碳途径,人工生物固碳似乎取得的效果更为明显,并逐渐成为当前关注的热点。相比于其它优化光呼吸的策略,未来通过光呼吸支路改造与优化以及Rubisco固碳效率的提升最具有潜力与前景,这对于解决环境、碳中和、粮食等重大问题,实现生态绿色可持续发展等战略要求具有重要的理论和实践意义。
| [1] |
HAGEMANN M, BAUWE H. Photorespiration and the potential to improve photosynthesis[J]. Curr Opin Chem Biol, 2016, 35: 109-116. DOI:10.1016/j.cbpa.2016.09.014 |
| [2] |
BAUWE H, HAGEMANN M, FERNIE A R. Photorespiration: Players, partners and origin[J]. Trends Plant Sci, 2010, 15(6): 330-336. DOI:10.1016/j.tplants.2010.03.006 |
| [3] |
AHMAD R, BILAL M, JEON J H, et al. Improvement of biomass accumulation of potato plants by transformation of cyanobacterial photorespiratory glycolate catabolism pathway genes[J]. Plant Biotechnol Rep, 2016, 10(5): 269-276. DOI:10.1007/s11816-016-0403-x |
| [4] |
WHITNEY S M, BIRCH R, KELSO C, et al. Improving recombinant Rubisco biogenesis, plant photosynthesis and growth by coexpressing its ancillary RAF1 chaperone[J]. Proc Natl Acad Sci USA, 2015, 112(11): 3564-3569. DOI:10.1073/pnas.1420536112 |
| [5] |
VON CAEMMERER S, QUICK W P, FURBANK R T. The development of C4 rice: Current progress and future challenges[J]. Science, 2012, 336(6089): 1671-1672. DOI:10.1126/science.1220177 |
| [6] |
SHI X X, BLOOM A. Photorespiration: The futile cycle?[J]. Plants, 2021, 10(5): 908. DOI:10.3390/plants10050908 |
| [7] |
SOMERVILLE C R. An early Arabidopsis demonstration: Resolving a few issues concerning photorespiration[J]. Plant Physiol, 2001, 125(1): 20-24. DOI:10.1104/pp.125.1.20 |
| [8] |
PETERHANSEL C, HORST I, NIESSEN M, et al. Photorespiration[J]. Arabidopsis Book, 2010, 2010(8): e130. DOI:10.1199/tab.0130 |
| [9] |
BAUWE H, KOLUKISAOGLU Ü. Genetic manipulation of glycine decarboxylation[J]. J Exp Bot, 2003, 54(387): 1523-1535. DOI:10.1093/jxb/erg171 |
| [10] |
DELLERO Y, JOSSIER M, SCHMITZ J, et al. Photorespiratory glycollate-glyoxylate metabolism[J]. J Exp Bot, 2016, 67(10): 3041-3052. DOI:10.1093/jxb/erw090 |
| [11] |
FERNIE A R, BAUWE H. Wasteful, essential, evolutionary stepping stone? The multiple personalities of the photorespiratory pathway[J]. Plant J, 2020, 102(4): 666-677. DOI:10.1111/tpj.14669 |
| [12] |
PICK T R, BRÄUTIGAM A, SCHULZ M A, et al. PLGG1, a plastidic glycolate glycerate transporter, is required for photorespiration and defines a unique class of metabolite transporters[J]. Proc Natl Acad Sci USA, 2013, 110(8): 3185-3190. DOI:10.1073/pnas.1215142110 |
| [13] |
WANG L M. Construction of the GCGT photorespiratory bypass and its effects on photosynthesis and yield in rice [D]. Guangzhou: South China Agricultural University, 2019. 王丽敏. GCGT光呼吸支路的构建及其对水稻光合效率和产量的影响研究 [D]. 广州: 华南农业大学, 2019. |
| [14] |
FOYER C H, BLOOM A J, QUEVAL G, et al. Photorespiratory metabolism: Genes, mutants, energetics, and redox signaling[J]. Annu Rev Plant Biol, 2009, 60: 455-484. DOI:10.1146/annurev.arplant.043008.091948 |
| [15] |
OBATA T, FLORIAN A, TIMM S, et al. On the metabolic interactions of (photo) respiration[J]. J Exp Bot, 2016, 67(10): 3003-3014. DOI:10.1093/jxb/erw128 |
| [16] |
HEYNEKE E, FERNIE A R. Metabolic regulation of photosynthesis[J]. Biochem Soc Trans, 2018, 46(2): 321-328. DOI:10.1042/BST20170296 |
| [17] |
SIMKIN A J, LÓPEZ-CALCAGNO P E, RAINES C A. Feeding the world: Improving photosynthetic efficiency for sustainable crop production[J]. J Exp Bot, 2019, 70(4): 1119-1140. DOI:10.1093/jxb/ery445 |
| [18] |
UMNAJKITIKORN K, SADE N, DEL MAR RUBIO M, et al. Silencing of OsCV (chloroplast vesiculation) maintained photo respiration and N assimilation in rice plants grown under elevated CO2[J]. Plant Cell Environ, 2020, 43(4): 920-933. DOI:10.1111/pce.13723 |
| [19] |
BLOOM A J, ASENSIO J S R, RANDALL L, et al. CO2 enrichment inhibits shoot nitrate assimilation in C3 but not C4 plants and slows growth under nitrate in C3 plants[J]. Ecology, 2012, 93(2): 355-367. DOI:10.1890/11-0485.1 |
| [20] |
WUJESKA-KLAUSE A, CROUS K Y, GHANNOUM O, et al. Lower photorespiration in elevated CO2 reduces leaf N concentrations in mature Eucalyptus trees in the field[J]. Glob Chang Biol, 2019, 25(4): 1282-1295. DOI:10.1111/gcb.14555 |
| [21] |
GAO X, YUAN H M, HU Y Q, et al. Mutation of Arabidopsis CATALASE2 results in hyponastic leaves by changes of auxin levels[J]. Plant Cell Environ, 2014, 37(1): 175-188. DOI:10.1111/pce.12144 |
| [22] |
KERCHEV P, DE SMET B, WASZCZAK C, et al. Redox strategies for crop improvement[J]. Antioxid Redox Sign, 2015, 23(14): 1186-1205. DOI:10.1089/ars.2014.6033 |
| [23] |
ROJAS C M, MYSORE K S. Glycolate oxidase is an alternative source for H2O2 production during plant defense responses and functions independently from NADPH oxidase[J]. Plant Signal Behav, 2012, 7(7): 752-755. DOI:10.4161/psb.20429 |
| [24] |
ZHANG Z S, XU Y Y, XIE Z W, et al. Association-dissociation of glycolate oxidase with catalase in Rice: A potential switch to modulate intracellular H2O2 levels[J]. Mol Plant, 2016, 9(5): 737-748. DOI:10.1016/j.molp.2016.02.002 |
| [25] |
YUAN H M, LIU W C, LU Y T. CATALASE2 coordinates SA-mediated repression of both auxin accumulation and JA biosynthesis in plant defenses[J]. Cell Host Microbe, 2017, 21(2): 143-155. DOI:10.1016/j.chom.2017.01.007 |
| [26] |
EISENHUT M, RUTH W, HAIMOVICH M, et al. The photorespiratory glycolate metabolism is essential for cyanobacteria and might have been conveyed endosymbiontically to plants[J]. Proc Natl Acad Sci USA, 2008, 105(44): 17199-17204. DOI:10.1073/pnas.0807043105 |
| [27] |
ZELITCH I, SCHULTES N P, PETERSON R B, et al. High glycolate oxidase activity is required for survival of maize in normal air[J]. Plant Physiol, 2009, 149(1): 195-204. DOI:10.1104/pp.108.128439 |
| [28] |
GUO Y P. A study on advances in plant photorespiration[J]. Acta Pratacult Sin, 2014, 23(4): 322-329. 郭玉朋. 植物光呼吸途径研究进展[J]. 草业学报, 2014, 23(4): 322-329. DOI:10.11686/cyxb20140439 |
| [29] |
ZHANG Z S, PENG X X. Multifunctional roles of photorespiration and its regulation for the balance[J]. Plant Physiol J, 2016, 52(11): 1692-1702. 张智胜, 彭新湘. 光呼吸的功能及其平衡调控[J]. 植物生理学报, 2016, 52(11): 1692-1702. DOI:10.13592/j.cnki.ppj.2016.1017 |
| [30] |
KANGASJÄRVI S, NEUKERMANS J, LI S C, et al. Photosynthesis, photorespiration, and light signalling in defence responses[J]. J Exp Bot, 2012, 63(4): 1619-1636. DOI:10.1093/jxb/err402 |
| [31] |
LI J Y, HU J P. Using co-expression analysis and stress-based screens to uncover Arabidopsis peroxisomal proteins involved in drought response[J]. PLoS One, 2015, 10(9): e0137762. DOI:10.1371/journal.pone.0137762 |
| [32] |
HODGES M, DELLERO Y, KEECH O, et al. Perspectives for a better understanding of the metabolic integration of photorespiration within a complex plant primary metabolism network[J]. J Exp Bot, 2016, 67(10): 3015-3026. DOI:10.1093/jxb/erw145 |
| [33] |
WINGLER A, LEA P J, QUICK W P, et al. Photorespiration: Metabolic pathways and their role in stress protection[J]. Philos Trans R Soc Lond B Biol Sci, 2000, 355(1402): 1517-1529. DOI:10.1098/rstb.2000.0712 |
| [34] |
TIMM S, HAGEMANN M. Photorespiration: How is it regulated and how does it regulate overall plant metabolism?[J]. J Exp Bot, 2020, 71(14): 3955-3965. DOI:10.1093/jxb/eraa183 |
| [35] |
ORT D R, MERCHANT S S, ALRIC J, et al. Redesigning photosynthesis to sustainably meet global food and bioenergy demand[J]. Proc Natl Acad Sci USA, 2015, 112(28): 8529-8536. DOI:10.1073/pnas.1424031112 |
| [36] |
SOUTH P F, CAVANAGH A P, LOPEZ-CALCAGNO P E, et al. Optimizing photorespiration for improved crop productivity[J]. J Integr Plant Biol, 2018, 60(12): 1217-1230. DOI:10.1111/jipb.12709 |
| [37] |
LIN M T, OCCHIALINI A, ANDRALOJC P J, et al. A faster Rubisco with potential to increase photosynthesis in crops[J]. Nature, 2014, 513(7519): 547-550. DOI:10.1038/nature13776 |
| [38] |
OCCHIALINI A, LIN M T, ANDRALOJC P J, et al. Transgenic tobacco plants with improved cyanobacterial Rubisco expression but no extra assembly factors grow at near wild-type rates if provided with elevated CO2[J]. Plant J, 2016, 85(1): 148-160. DOI:10.1111/tpj.13098 |
| [39] |
LIN M T, HANSON M R. Red algal Rubisco fails to accumulate in transplastomic tobacco expressing Griffithsia monilis RbcL and RbcS genes[J]. Plant Direct, 2018, 2(2): e00045. DOI:10.1002/pld3.45 |
| [40] |
MATSUMURA H, SHIOMI K, YAMAMOTO A, et al. Hybrid Rubisco with complete replacement of rice Rubisco small subunits by sorghum counterparts confers C4 plant-like high catalytic activity[J]. Mol Plant, 2020, 13(11): 1570-1581. DOI:10.1016/j.molp.2020.08.012 |
| [41] |
GUNN L H, AVILA E M, BIRCH R, et al. The dependency of red Rubisco on its cognate activase for enhancing plant photosynthesis and growth[J]. Proc Natl Acad Sci USA, 2020, 117(41): 25890-25896. DOI:10.1073/pnas.2011641117 |
| [42] |
MUELLER-CAJAR O, WHITNEY S M. Directing the evolution of Rubisco and Rubisco activase: First impressions of a new tool for photosynthesis research[J]. Photosynth Res, 2008, 98(1/2/3): 667-675. DOI:10.1007/s11120-008-9324-z |
| [43] |
WHITNEY S M, SHARWOOD R E. Construction of a tobacco master line to improve Rubisco engineering in chloroplasts[J]. J Exp Bot, 2008, 59(7): 1909-1921. DOI:10.1093/jxb/erm311 |
| [44] |
SINETOVA M A, KUPRIYANOVA E V, MARKELOVA A G, et al. Identification and functional role of the carbonic anhydrase Cah3 in thylakoid membranes of pyrenoid of Chlamydomonas reinhardtii[J]. Biochim Biophys Acta, 2012, 1817(8): 1248-1255. DOI:10.1016/j.bbabio.2012.02.014 |
| [45] |
NIEDERHUBER M J, LAMBERT T J, YAPP C, et al. Superresolution microscopy of the β-carboxysome reveals a homogeneous matrix[J]. Mol Biol Cell, 2017, 28(20): 2734-2745. DOI:10.1091/mbc.E17-01-0069 |
| [46] |
SOMMER M, CAI F, MELNICKI M, et al. β-carboxysome bioinformatics: Identification and evolution of new bacterial microcompartment protein gene classes and core locus constraints[J]. J Exp Bot, 2017, 68(14): 3841-3855. DOI:10.1093/jxb/erx115 |
| [47] |
RAE B D, LONG B M, WHITEHEAD L F, et al. Cyanobacterial carboxysomes: Microcompartments that facilitate CO2 fixation[J]. J Mol Microbiol Biotechnol, 2013, 23(4/5): 300-307. DOI:10.1159/000351342 |
| [48] |
SHI M L, ZHOU L, WANG Q, et al. Advances in the study on the modification of carbon dioxide metabolic pathways in plants [J/OL]. Synth Biol J, 2022. (2022-06-01). http://kns.cnki.net/kcms/detail/10.1687.Q.20220531.1513.002.html. 史梦琳, 周琳, 王庆, 等. 植物二氧化碳代谢途径改造研究进展 [J/OL]. 合成生物学, 2022. (2022-06-01). http://kns.cnki.net/kcms/detail/10.1687.Q.20220531.1513.002.html. |
| [49] |
HANSON M R, LIN M T, CARMO-SILVA A E, et al. Towards engineering carboxysomes into C3 plants[J]. Plant J, 2016, 87(1): 38-50. DOI:10.1111/tpj.13139 |
| [50] |
STEIN M, GABDOULLINE R R, WADE R C. Cross-species analysis of the glycolytic pathway by comparison of molecular interaction fields[J]. Mol Biosyst, 2010, 6(1): 152-164. DOI:10.1039/b912398a |
| [51] |
WANG H, YAN X, AIGNER H, et al. Rubisco condensate formation by CcmM in β-carboxysome biogenesis[J]. Nature, 2019, 566(7742): 131-135. DOI:10.1038/s41586-019-0880-5 |
| [52] |
LONG B M, HEE W Y, SHARWOOD R E, et al. Carboxysome encapsulation of the CO2-fixing enzyme Rubisco in tobacco chloroplasts[J]. Nat Commun, 2018, 9(1): 3570. DOI:10.1038/s41467-018-06044-0 |
| [53] |
SHARWOOD R E. A step forward to building an algal pyrenoid in higher plants[J]. New Phytol, 2017, 214(2): 496-499. DOI:10.1111/nph.14514 |
| [54] |
KU M S B, CHO D, LI X, et al. Introduction of genes encoding C4 photosynthesis enzymes into rice plants: Physiological consequences[J]. Novartis Found Symp, 2001, 236: 100-111. DOI:10.1002/9780470515778.ch8 |
| [55] |
ZHU X G, LONG S P, ORT D R. Improving photosynthetic efficiency for greater yield[J]. Annu Rev Plant Biol, 2010, 61: 235-261. DOI:10.1146/annurev-arplant-042809-112206 |
| [56] |
LING X L. Construction of GOC photorespiratory bypass in Solanum tuberosum and its impact on photosynthetic efficiency and yield [D]. Guangzhou: South China Agricultural University, 2019. 林秀灵. 马铃薯中导入GOC光呼吸支路对其光合效率和产量的影响研究 [D]. 广州: 华南农业大学, 2019. |
| [57] |
FURBANK R T. Walking the C4 pathway: Past, present, and future[J]. J Exp Bot, 2017, 68(2): e1-e10. DOI:10.1093/jxb/erx006 |
| [58] |
WANG P, KHOSHRAVESH R, KARKI S, et al. Re-creation of a key step in the evolutionary switch from C3 to C4 leaf anatomy[J]. Curr Biol, 2017, 27(21): 3278-3287. DOI:10.1016/j.cub.2017.09.040 |
| [59] |
LIN H C, ARRIVAULT S, COE R A, et al. A partial C4 photosynthetic biochemical pathway in rice[J]. Front Plant Sci, 2020, 11: 564463. DOI:10.3389/fpls.2020.564463 |
| [60] |
KEBEISH R, NIESSEN M, THIRUVEEDHI K, et al. Chloroplastic photorespiratory bypass increases photosynthesis and biomass production in Arabidopsis thaliana[J]. Nat Biotechnol, 2007, 25(5): 593-599. DOI:10.1038/nbt1299 |
| [61] |
MAIER A, FAHNENSTICH H, VON CAEMMERER S, et al. Transgenic introduction of a glycolate oxidative cycle into A. thaliana chloroplasts leads to growth improvement[J]. Front Plant Sci, 2012, 3: 38. DOI:10.3389/fpls.2012.00038 |
| [62] |
PETERHANSEL C, BLUME C, OFFERMANN S. Photorespiratory bypasses: How can they work?[J]. J Exp Bot, 2013, 64(3): 709-715. DOI:10.1093/jxb/ers247 |
| [63] |
NÖLKE G, HOUDELET M, KREUZALER F, et al. The expression of a recombinant glycolate dehydrogenase polyprotein in potato (Solanum tuberosum) plastids strongly enhances photosynthesis and tuber yield[J]. Plant Biotechnol J, 2014, 12(6): 734-742. DOI:10.1111/pbi.12178 |
| [64] |
DALAL J, LOPEZ H, VASANI N B, et al. A photorespiratory bypass increases plant growth and seed yield in biofuel crop Camelina sativa[J]. Biotechnol Biofuels, 2015, 8(1): 175. DOI:10.1186/s13068-015-0357-1 |
| [65] |
SOUTH P F, CAVANAGH A P, LIU H W, et al. Synthetic glycolate metabolism pathways stimulate crop growth and productivity in the field[J]. Science, 2019, 363(6422): eaat9077. DOI:10.1126/science.aat9077 |
| [66] |
SHEN B R, WANG L M, LIN X L, et al. Engineering a new chloroplastic photorespiratory bypass to increase photosynthetic efficiency and productivity in rice[J]. Mol Plant, 2019, 12(2): 199-214. DOI:10.1016/j.molp.2018.11.013 |
| [67] |
WANG L M, SHEN B R, LI B D, et al. A synthetic photorespiratory shortcut enhances photosynthesis to boost biomass and grain yield in rice[J]. Mol Plant, 2020, 13(12): 1802-1815. DOI:10.1016/j.molp.2020.10.007 |
| [68] |
ROELL M S, VON BORZYSKOWSKI L S, WESTHOFF P, et al. A synthetic C4 shuttle via the β-hydroxyaspartate cycle in C3 plants[J]. Proc Natl Acad Sci USA, 2021, 118(21): e2022307118. DOI:10.1073/pnas.2022307118 |
 2022, Vol. 30
2022, Vol. 30



