2. 中国科学院大学, 北京 100049;
3. 顺德职业技术学院, 广东 佛山 528300
2. University of Chinese Academy of Sciences, Beijing 100049, China;
3. Shunde Polytechnic, Foshan 528300, Guangdong, China
转录因子(transcription factors, TFs)是指能结合基因启动子从而调控基因表达的一类蛋白质分子。转录因子的调节是一个十分复杂的过程,在调控真核生物生长发育中起着重要作用。1996年,Laux等[1]首次从拟南芥(Arabidopsis thaliana)中鉴定到WUSCHEL (WUS)转录因子,该转录因子在顶端分生组织维持起到关键作用[2]。随后,越来越多的植物WUSCHEL-related转录因子被克隆和分析,均含有同源异形盒(homeobox, HB)的保守结构域, 属于homeobox超家族的成员,被命名为WOX转录因子[1, 3]。
WOX转录因子在植物发育过程中扮演的角色非常多样化,且其调控作用非常重要。以模式植物拟南芥为例,目前发现拟南芥共有15个WOX成员,功能涉及从干细胞的维持到胚胎模式的形成,包括枝条、花分生组织形成、根形成等生长发育过程[1, 4–5],均被证明发挥着广泛的作用;水稻(Oryza sativa)中共有13个WOX成员[6–9],如WOX13影响水稻花发育和抗旱性[6],WOX11调控不定根生长发育[7],WOX3影响茎尖分生组织和叶片发育[8]等。在铁皮石斛(Dendrobium officinale)中,不同WOX基因在原球茎发育过程中表达具有差异性,有研究表明DoWOX8基因可能与顶端分生组织发育相关,其在顶端分生组织形成期表达量高于其他DoWOXs[10], 这为兰科草本植物的WOX家族成员研究添上了重要一笔。在茉莉花(Jasminum samba)中,有研究证明WOX4和WOX1分别与愈伤组织增殖和根再生有关[11],为WOX基因在木本植物器官发生的功能研究提供了一条引领性的线索。在矮牵牛(Petunia hybrida)和金鱼草(Antirrhinum majus)中,有研究表明WUS直系同源物突变体导致了相关功能的缺失[12–13],这证实了WOX基因在维持茎尖分生组织(shoot apical meristem, SAM)起着不可或缺的作用。在多种植物中, WOX基因参与调控植物的花发育和花结构的形成[14]。不同物种中WOX功能的相关研究取得了重要进展,为解释WOX转录因子在植物发育相关过程和调控机理奠定基础。
在植物发育过程中,胚胎发育是保证植物正常生长发育的起源和核心[15],WOX转录因子在植物胚胎发育的启动区域特异性转录程序过程中有着重要的作用[4],WUSCHEL是胚胎发育早期表达的分生组织的基因[16]。尽管胚胎发育这一过程至关重要,但是其相关机制仍不清晰,因此,对调控胚胎发育的重要转录因子的研究进而解释其相关机制充满机遇和挑战。本文对WOX家族不同成员、不同物种中WOXs成员的表达调控进行综述,将植物表观调控对植物发育的潜在影响和WOX家族成员对植物发育的影响联系起来,旨在为揭示其相关机理提供依据,并为相关研究提供思路。
1 WOX家族成员概述HOMEOBOX最早发现于黑腹果蝇中[17],其结构特征是有一小段氨基酸(60~66个残基),这些氨基酸折叠成一个称为同源异形结构域的DNA结合域,该结构域由HB DNA序列编码[18]。动物HOX蛋白是典型的HB调节因子,HOX在黑腹果蝇中最初是通过同源异型突变导致身体片段的转变被鉴定发现的[19]。更多的研究表明,HB转录因子也在植物中发挥广泛作用。1996年,Laux等[1]从拟南芥中首次鉴定到一个在顶端分生组织维持起到关键作用的WUSCHEL (WUS)。WOX转录因子家族在植物发育过程的地位至关重要,参与植物的多种发育过程,如顶端分生组织的维持、花序发育、根发育和胚胎发育等。
关于WOX家族成员分类的研究主要集中在进化起源上。WOX家族成员的进化情况可以根据HB同源异形结构域蛋白序列的系统发育重建进行推测,有研究表明DNA-binding motif可能起源于真核生物分化之前[20]。以HB蛋白质的进化发育情况为基础,植物WOX的系统发育树可以分为3个分支,分别是WUS clade、Intermediate clade和Ancient clade[21–22]。在拟南芥中,WUS和WOX1~WOX7及其在种子植物中的直系同源蛋白属于WUS clade, WOX8、WOX9、WOX11和WOX12及其在维管植物中的直系同源蛋白属于Intermediate clade, WOX10、WOX13和WOX14及其在维管和非维管植物中的直系同源蛋白属于Ancient clade[4, 23]。研究表明只有Ancient clade包含来自绿藻和非维管苔藓小立碗藓(Physcomitrella patens)的WOX序列[24]; Intermediateclade除开花植物外还包括维管植物卷柏(Selaginella moellendorffii)[24];WUS clade仅包含开花植物的蛋白质序列[24]。这说明引起Intermediate clade的旁系同源基因必须已经存在于维管植物的最后共同祖先中。
WOX编码的蛋白结构的典型特征是存在同源异形结构域(homeodomain, HD)(图 1),该结构域通过形成螺旋-转角-螺旋(helix-turn-helix, HTH)结构与DNA结合,调控下游基因的转录。植物WOX同源异形结构域通常在高度保守的结构中有2个延伸的环, 该环在生物发育中具有重要意义[7],且在HB其他家族如TALE家族也被发现[25]。此外,研究表明WUS box是WUS clade成员特有的结构域[4], 在WUS和其他3个成员(即WOX1、WOX4和WOX5)的WUSbox上游大约10个残基处有1个酸性结构域,可能行使转录激活作用。而ERF-associated amphiphilic repression (EAR)结构域可能具有转录抑制功能,在拟南芥中只有WUS、WOX5和WOX7包含严格意义上的羧基末端EAR motif[24]。有趣的是,在WOX2的同源异形结构域的下游还有1个假定的锌指结构域[4](图 1),这是WOX2成员特有的。
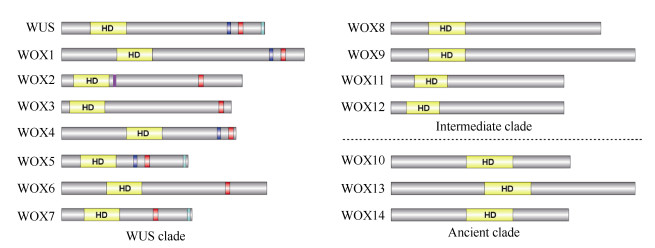
|
图 1 拟南芥AtWOX家族蛋白结构示意图。同源域(HD)(黄色)是该家族最突出和最明确的特征;WUS box motif (红色)在严格意义上被定义为T-L-[DEQP]-L-F-P-[GITVL]-[GSKNTCV];酸性结构域(蓝色)位于WUS-box上游大约10个残基处;EAR结构域(绿色)位于羧基末端,其结构特征是[LVI]-X-[LVI]-X-[LVI];1个假定的锌指结构域(紫色)位于HD下游,是WOX2特有的。 Fig. 1 Schematic domain structures of WOX family proteins in Arabidopsis thaliana. The homeodomain (HD) (yellow) is the most prominent and defining feature of the family. The WUS box motif (red) was defined in a strict sense, as T-L-[DEQP]-L-F-P-[GITVL]-[GSKNTCV]. An acidic domain (blue) was identified with approximately ten residues upstream of the WUS box. The EAR domain (green) is located at the carboxyl terminus and is characterized by [LVI]-X-[LVI]-X-[LVI]. A putative zinc finger domain (purple) is downstream of HD and is specific to WOX2. |
WOX转录因子的表达模式具有组织特异性。具体表现为有些WOXs成员可能在某个特定器官中高表达,但在其他器官基本检测不到,如WOX5在拟南芥的根尖分生组织的静止中心细胞和侧根原基中表达[5, 26–27],但在其他部位的表达量低或者检测不到[27]。除了特异表达外,WOX家族成员也存在一定的相关性,也可以同时在多个器官表达进而调控植物的生长发育,如WUS不仅在拟南芥的茎中表达[5],而且在拟南芥和禾本科植物的胚珠和花药中表达[28]。尽管WOX家族成员间在结构上存在很大的差异,如同源异形结构域、保守肽等,但是从进化角度看,这些WOX之间似乎会存在从祖先而来的共同功能[29],这为寻找WOX家族成员的共性提供线索。早前研究表明,WOX基因PRESSED FLOWER (PRS)和WOX1的表达模式是重叠或相似的[29],且通过wox1prs双突变体的表型证明了他们在功能上是冗余的;PRS/WOX3在玉米(Zea mays)中的同源基因是NARROW SHEATH 1和NARROW SHEATH 2,对叶缘发育具有重要调控作用[30],进而说明单子叶和双子叶植物的花发育过程中PRS/WOX3基因具有功能保守性[14]。
WOX除了在重叠的表达模式下具有功能冗余外, 也存在表达模式不同但功能具有冗余性的成员。WUS clade具有特殊的WUS box,有研究表明WUS可以弥补在不同启动子表达下prs和wox5突变表型[31],且拟南芥中的大部分WUS clade成员均可回补烟草(Nicotiana tabacum) wox1同源突变体lam1的缺陷[32],但非WUS clade则不能回补am1突变体的表型[14]。这证明了不同WUS clade成员在不同表达模式下功能仍具有重叠性,且说明了WUS clade特有的WUS box具有一定特殊性和重要性。此外,有研究表明,与WUS box或SRDX阻遏物域融合的嵌合WOX7、WOX9和WOX13蛋白(非WUS clade)可以回补lam1的表型,这进一步证实了WUS box的功能重要性[32]。这暗示了WUS clade成员功能上的重叠可能是存在某种共性,如均含有功能特殊的WUS box,相较于更古老的Ancient clade和Intermediate clade,WUS clade存在的1个或多个转录抑制因子结构域[14],或许在植物的某个生长发育阶段扮演着核心的角色。
综上所述,WOX转录因子表达模式的不同或相似导致了它们在功能上的多样性或冗余性。同时,功能的多样性在不同植物不同发育过程所起的作用也得到了充分体现,进一步为WOX在植物发育扮演多样化角色奠定了基础;此外,功能的冗余性增加了WOX的稳定性,例如某个WOX转录因子突变导致的缺陷或许可以由其他具有相同或相似功能的WOX弥补,保障了植物的正常生长发育。
3 WOX参与植物发育许多同源框基因在植物发育中发挥了重要作用。WOX家族作为植物特定的转录因子家族,在植物的生长发育扮演了许多重要的角色,如植物胚胎发育和体胚发生、花发育、根发育、愈伤组织的形成和维持以及干细胞维持等。
3.1 参与植物胚胎发育和体胚发生植物胚胎发育是一个十分复杂的过程。目前有关早期胚胎发生的分子机理的研究进展缓慢,早期胚胎小且被母体细胞包围导致难以跟踪观察[33]。体细胞胚胎发生(简称体胚发生)是指在没有受精的情况下从体细胞发育出胚,进而发育成为独立个体的过程[34–35],在很大程度上克服了研究胚胎发育的局限性。此外,体胚类似于合子胚,两者经历几乎相同的发育阶段[36–37],很好诠释了植物细胞的全能性表达。近年来该方面的研究发展迅速。有研究表明,体胚发生过程涉及的发育转换(诱导体细胞形成全能胚胎进而再生为完整植株)与一系列识别内部信号和外部刺激的分子密切相关[38]。其中转录因子在胚胎发育和体胚发生等复杂的生物过程发挥着至关重要的调节功能。
大量研究表明,WOX转录因子在植物的胚胎发育中起着关键的调控作用[4, 14, 39–40],WOX的差异表达影响胚胎早期细胞的分化命运。在拟南芥中, 不同WOX家族成员的表达动态影响了胚胎发育中的区域特异性转录,为揭示拟南芥胚胎发育模式的建成机理起到了很好的助推作用,如WOX2和WOX8在卵细胞和受精卵以共表达和不对称分裂模式分别局限于受精卵的顶子细胞和基部子细胞, WOX9在胚胎发育的中心胚区启动发挥重要作用, WOX5在大多数早期球形胚胎的垂体中特异表达等[4], WUS在胚胎发生被激活重新表达[41];在番茄(Lycopersicon esculentum)中,WOX9是胚胎和胚后发育期间维持细胞分裂活性的必需基因[42];在水稻中, WOX在调控胚胎发育的区域特异性转录扮演着重要角色[43]。此外,研究表明,WUS突变后,胚胎的顶端分生组织无法正常形成,且突变植株中茎顶端分生组织形成受到影响[1]。植物的胚胎发育是复杂的,胚胎模式不仅受到生长素反应的影响[44],而且细胞之间的分子差异在胚胎发育中相对较早地建立[45–46]。
WOX转录因子在植物体胚发生扮演着重要的角色。有研究表明,WUS同源异形结构域蛋白的异位表达可以促进幼苗的体细胞胚发育[47–48]。WUS不直接决定胚胎命运,而是主要维持未分化细胞状态,以响应不同的刺激,从而改变植物体内组织的发育命运[48–49],说明其可能在体胚发生等未分化状态具有重要调控作用。在拟南芥中,体胚发生可通过单个转录因子如WUSCHEL (WUS)的异位过表达[48]来实现。有研究表明,WOX2和WOX3是拟南芥体胚发生必不可少的关键转录因子[50];在云杉(Picea asperata)中,PaWOX2与体胚发生过程存在相关性,例如PaWOX2在早期体细胞胚胎发育过程中高度表达,但随着胚胎的成熟而下降[22];在火炬松(Pinus taeda)中,STIMPY/WOX9通过调节WUS的活性来维持干细胞群[51–52],暗示其在胚胎发生过程中扮演重要角色。且遗传学研究表明, WUS/ CLAVATA (CLV)自我调节回路对于维持干细胞的特性非常重要[53–55]。因此,WUS-CLV是一个极其复杂的调控网络,对于体胚发生机理的进一步阐明是一个挑战。
此外,关于胚胎发育的机理,有研究表明通过调控运输和局部生长素反应可以影响胚胎发育模式[4]。例如,胚胎中特定的生长素反应作为信号分子的调控可以保证根发育正常[56],但是相关胚胎模式的形成机制目前尚不太清楚,其中作为胚胎发育的关键调控基因——WOX家族成员是否与生长素调控胚胎发育存在一定联系,或许可以为揭示胚胎发育调控的网络机制提供突破口。庆幸的是,目前胚胎发育的研究跳出了以往传统的思维,全基因组的表观遗传分析揭示了siRNA在表观遗传学层面上维持胚胎基因组稳定的作用[57],这是一个巨大突破。然而,目前尚未直接有从表观遗传角度研究以揭示WOX调控胚胎发育机理的相关报道,因此WOX对胚胎发育的研究仍需进一步探讨。
3.2 参与植物花发育花作为植物的重要功能器官,在植物繁殖和植物进化过程中发挥着关键的作用。研究表明WOX在植物的器官发育尤其是花器官发育具有重要作用[14, 58]。在拟南芥中,WUS是花中的干细胞维持所必需的, WOX13和WOX14影响花的发育[59],PRS/WOX3可以调控拟南芥花的侧轴发育和影响花的外观[60];在矮牵牛中,WOX1可以维持花发育中的正常表型, WOX8是花序发育和体系结构所必需的[61];在紫花苜蓿(Medicago sativa)和烟草中,花的表型受到了WOX1的影响[32];在玉米中,NARROW SHEATH1 (NS1)和NS2 (WOX3的同源物)保证叶片和花器官的正常表型[62]。这说明WOX转录因子有助于维持花卉结构和形态,是植物花发育不可或缺的角色。
尽管不同的WOX家族成员在结构上差异很大, 但部分成员在功能上存在冗余。研究表明, WOX1在调控不同植物如矮牵牛、紫花苜宿、烟草和豌豆等的花发育中是功能冗余的[29];EVERG REEN (EVG, WOX9亚家族代表之一)是一个复制型基因,它在花序发育过程中获得了新的功能[63];在玉米叶片的侧结构域发育中,窄鞘复制型基因可以执行祖先保守的冗余功能[62]。此外,花其实是经过修饰的叶片,WOX在影响花发育的同时会影响营养生长,如拟南芥中WOX基因PRS1的无效突变不仅会导致叶片上的侧托叶缺失,还会使花朵上的侧萼片和雄蕊缺失[31],因此,关注植物的叶片生长或许可以填补植物花发育研究的部分空白领域。
3.3 参与根的发育根尖分生组织是根生长发育的细胞源泉[64]。植物根系发育是复杂的,不仅包括胚胎期间的初生根,而且包括萌发后产生的侧根和不定根,因此了解其相关的分子机理非常必要。目前有关WOX转录因子调控植物根发育的研究较多。在水稻中, WOX11是调控不定根生长发育的关键基因,很大程度上可以影响冠状根发育[7, 65];在拟南芥中,根的发育不仅受到WOX13和WOX14的影响[59]以及WOX11和WOX12的共同调控[66],且WOX5可以在根分生组织中发挥调控作用[5],WOX4在形成层(根和茎中的一种分生组织)中也发挥了重要作用[26, 67]; 在茉莉花中,WOX1对根的影响主要体现在根原基起始和分化[11];在小麦(Triticum aestivum)中,WOX5在根中大量表达[68]。这说明WOX家族成员在不同植物根发育中扮演的角色重要且多样。
高等植物的根发育过程由多种信号因子调控, 如光、植物激素、环境信号等多种信号调控。植物激素对根系的调节起着重要的作用,其中WOX家族成员从植物激素角度调控植物根系发育的相关研究[69–71]较多。正常的根发育需要在胚胎模式中有特定的生长素反应,起着发出信号的作用。研究表明,WOX11可能是生长素和细胞分裂素的整合子,它可以通过对生长素和细胞分裂素信号协调来影响水稻根的细胞分裂[65],这为WOX11参与植物根的转录程序机制揭开了面纱,进而补充了WOX转录因子调控植物根发育的相关研究。
3.4 参与植物愈伤组织的形成和维持植物具有强大的发育可塑性,愈伤组织可以使植物获得再生。WOX家族成员对植物愈伤组织的形成和维持起着关键的调控作用。研究表明,B型ARR-HD-ZIPⅢ复合体可以诱导WUS在植物愈伤组织表达[41]。愈伤组织转变为绿色或黄绿色后,需要重新编程为芽祖细胞以形成不定芽。研究表明, 芽祖细胞的形成需要细胞分裂素诱导WUS的表达[41, 72]。在拟南芥中,拟南芥转录因子LBD16被WOX11激活后在CIM (愈伤组织培养基)中特异表达, WOX11-LBD16途径是愈伤组织获得多能性的保证[73]。愈伤组织命运转变(再生能力细胞转变为基源细胞)的首步需要生长素激活WOX11和WOX12,第二步是WOX11/WOX12激活WOX5/WOX7和LBD16的表达[66];水稻愈伤组织中含有WOX11转录本[7], 暗示WOX11可能影响水稻愈伤组织的生长;在茉莉花中,WOX4、WOX13和一个特异的同源异形盒基因对愈伤组织的诱导和增殖起调控作用,进一步表明了WOX家族成员在愈伤组织诱导中存在多方面的遗传相互作用[11]。因此,了解WOX家族成员在愈伤组织的激活,有助于揭示植物发育中调控植物愈伤组织的机制。
尽管对植物细胞全能性的研究历史很悠久,但其分子机制在很大程度上仍然是模糊的。目前关于WOX家族影响植物愈伤组织的研究不多,其中比较热门的是结合组学技术的研究。选择WOX转录因子作为切入点进行分析进而实现精准定位的组学技术,或许可以解析WOX在愈伤组织表达情况,进而揭示调控愈伤组织的相关机制。
3.5 WOX参与植物干细胞发育干细胞是一类未分化和持续分裂的细胞群,植物干细胞实为茎尖分生组织(SAM)、根尖分生组织(RAM)等各类分生组织,可以高效率地利用生物反应器生产活性物质,在药品(下游制药)、食品(功能性食品)以及化妆品行业具有广阔的应用前景[74]。
大量研究发现WOX家族成员在植物未分化细胞的发生和维持具有重要的作用。WUS是WOX家族的创始成员,可以促进干细胞的识别和维持[1]。在拟南芥中,WUS在茎尖分生组织(SAM)中的干细胞维持扮演着关键调控因子的重要角色[1, 75],同时它也对茎尖顶端分生组织的维持和形成具有调控作用[48],因此可以认为WUS是干细胞维持的中央调节剂,是植物发育调控网络的一个密不可分的角色。此外,有研究表明细胞分裂素反应调节因子被WUS直接调节,从而控制分生组织功能[76]。有趣的是,WUS特有结构WUS box motif调控着植物发育中芽再生过程的干细胞生态位的建立[2],暗示WUS box motif或许是植物干细胞发育调控网络的关键因子,但仍需要进一步的实验验证。
植物中干细胞群体的自我更新是动态的,涉及了复杂的调控网络。WUS蛋白是调控植物茎端干细胞的核心因子,以WOX转录因子功能为中心的干细胞生态位的遗传机制的本质一直难以捉摸,目前有关干细胞生态位中保守的WOX功能的分子联系以及WOX如何与其他因子相关作用形成调控网络只有少量研究。因此,深入研究WOX蛋白及其互作因子,对于解析植物干细胞发育过程很关键。有研究表明,拟南芥HAIRY MERISTEM (HAM)家族转录调控因子与WOX蛋白相互作用,以促进干细胞的增殖,同时,形成不同干细胞生态位的基础之一是WOX和HAM家族成员重叠表达模式的差异,为植物发育过程中干细胞生成的调控机理建立了一个新的框架[77]。在拟南芥中,WUS和CLV相互作用共同参与芽和花分生组织的发育过程,是一个CLV抑制WUS转录水平、WUS表达诱导CLV3的动态反馈回路[54]。WUS、STM和CLV这3大因子在植物干细胞的调控关系是其解析调控网络的重要突破。研究表明,WUS与STM直接相互作用形成异源二聚体并结合到CLV3的启动子上,共同调控植物的茎端干细胞[78]。此外,WUS和其他WOXs成员功能常常可以替代,如WUS和WOX5基因在调节茎和根的干细胞维持的调控功能是可以互换的[5],说明WOX基因间具有冗余性。生长素可以促进形成层活动,离不开WOX4的参与[26–67]。研究表明,WOX4在形成层中表达丰富,对于形成层干细胞具有非常重要的调节作用。此外,WOX5在根分生组织中的静止中心特异表达,通过抑制干细胞的分化进而发挥调控作用[5]。其中,负责茎和根干细胞维持的作用机制与CLV3-WUS途径的负反馈环机制类似,通过CLE40 (一种与CLV3密切相关的肽)与ACR4受体结合从而抑制WOX5的转录活性,形成了一条CLE40-WOX5信号转导途径[79],表明分生组织的维持机制在进化上可能具有保守性。
综上所述,WOX转录因子可以调控植物的干细胞命运。WOX具有保守性功能[80],有研究表明,相关机制可能是WOXs的非细胞自主性赋予了其在干细胞的功能特性[31],可移动的WOX蛋白与其他关键蛋白互作或结合,共同发挥作用,并构成完整的胞间信号网络。
3.6 WOX与表观遗传共同调控植物发育WOX基因是植物发育的关键调控因子(图 2)。在植物发育中,WOX重编程基因表达的染色质机制在很大程度上是未知的。截至目前,已有多个WOX与DNA甲基化、组蛋白修饰等表观遗传相关的报道。初步研究表明WUS的调节会受到表观遗传机制的影响[81]。其中,DNA甲基化和基因表达之间存在潜在的相关性[82],有研究表明,在苹果(Malus domestica)中WOX转录因子在花芽中高表达,这与基因体区域的低甲基化水平有关[83];海棠(Malus hupehensis)成叶中2个WOXs基因的甲基化水平高于幼叶[84]; 有研究表明,WUS基因的表达受DNA甲基化的调控[85],且水稻和拟南芥的WOX启动子具有相同的DNA甲基化的特征性表观遗传标记[86], 暗示了WOXs可能在进化过程中具有表观遗传甲基化修饰以调节其表达。此外有研究表明,拟南芥组蛋白乙酰转移酶对WUSCHEL/AGAMOUS基因有负作用,他们共同调控花分生组织[87];WOX11通过与组蛋白H3K27me3去甲基化酶JMJ705相互作用,使得水稻茎顶端发育基因发生表观重编程以刺激水稻茎端生长[9];HaWUS通过观测其组蛋白乙酰化修饰水平与其他基因的相关性进一步解析了向日葵(Helianthus annuus)胚胎发育的调控机理[88]; WOX5通过组蛋白去乙酰化抑制分化因子CDF4以抑制拟南芥根中干细胞的分化[89]。综上所述,本文从表观遗传角度对WOX不同基因、不同植物中WOX基因的表达调控等方面进行了综述,将植物表观基因组对植物发育的影响和WOX家族对植物发育的调控联系起来,旨在为进一步揭示WOX调控植物发育的相关机理提供理论依据。
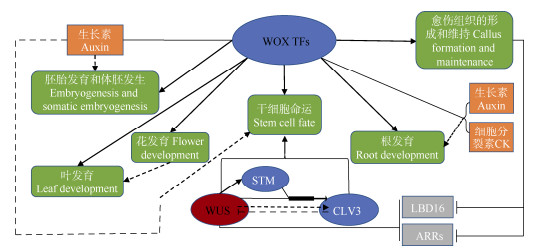
|
图 2 WOX转录因子基因在植物生长发育过程中的调控功能。Auxin: 生长素;CK: 细胞分裂素; ARRs: 拟南芥反应调节因子;LBD16: 侧器官边界-结构域16。实线箭头表示直接调节,虚线箭头表示间接调节,其机制尚不清楚。 Fig. 2 Regulation functions of WOX TFs during plant growth and development. Auxin: Auxin; CK: Cytokinin; ARRS: ARABIDOPSIS RESPONSE REGULATORS; LBD16: LATERAL ORGAN BOUNDARIES-DOMAIN 16 (LBD16). Arrows with a solid line indicate direct regulation and arrows with dotted lines show indirect regulation, and its mechanism is not clear. |
WOX转录因子参与植物的发育如胚胎发育、体胚发生,以及植物花、叶、根的发育,同时在愈伤组织形成中发挥重要作用。尽管大多数WOX亚家族的结构明显不同(如外显子数差异、每个亚家族特有的保守肽基序等),但这些亚家族中许多蛋白质确实存在共享一些祖先的共同功能现象,如功能重叠、功能互补等,这或许是研究WOX家族成员进化历程的突破口。其中,有些WOX家族成员尽管表达模式不同,但它们的蛋白质序列保持着相似的能力即功能重叠,说明了WOX家族成员功能的进化可能主要通过表达模式的改变。然而,WOX家族不同成员的功能互补主要通过功能多样性的多种成分调控元件实现,暗示WOX功能的进化可能是氨基酸序列变化起作用。WUS1可以补充拟南芥根分生组织中WOX5功能的缺失[5],它们主要通过不同启动子的特异性实现WUS1和PRS1/WOX3等价的蛋白-蛋白相互作用。
植物和动物最大的区别之一是植物不能移动, 因此,为了应对复杂多变的生长环境,植物必须进化出独特的信号机制来完成器官形成和发育,主要方式之一是通过胞间连丝进行细胞间运输,相关的移动分子包括蛋白、植物激素等。其中,蛋白大多数是非细胞自主性转录因子,植物中非细胞自主性转录因子参与了植物发育调控网络。在植物生长发育的过程中,WOX家族成员作为植物特有的转录因子家族,可以调控植物生长发育的多个环节,在调控过程中也涉及到了细胞分裂素、生长素等生长素的信号传导以及反应调节因子等多种因素,是一个极其复杂的调控网络。虽然前人已经进行了大量的相关研究,但是很多机理如这些因素在植物发育过程中“何时”、“何地”进行相互作用至今没有定论。
此外,从表观遗传角度研究WOX基因对植物发育的影响具有巨大的潜在研究价值。前人研究表明,植物为了防止异位表达,具有重要发育作用的转录因子在编码过程中往往会受到表观遗传抑制, 如组蛋白修饰和DNA甲基化[90]。植物WOX转录因子基因家族是重要的且特异的,有研究表明,转录因子WUS在组织培养中起关键作用,表观遗传调控缺陷的突变体在愈伤组织的表型差异与重编程调节子的差异表达有关,这主要通过调节WUS表达和生长素信号传导来实现[85]。综上所述,未来的研究或许可以从表观遗传层面对WOX家族成员参与植物发育的调控展开研究,并结合最新的组学技术和CRIPSR技术等工具,研究在不同物种的WOX家族成员在进化过程中出现的亚功能化和新功能化等相关机制,相信未来我们可以更加清晰地解析WOX家族成员调控不同植物不同发育过程涉及的作用机制。
| [1] |
LAUX T, MAYER K F, BERGER J, et al. The WUSCHEL gene is required for shoot and floral meristem integrity in Arabidopsis[J]. Development, 1996, 122(1): 87-96. DOI:10.1242/dev.122.1.87 |
| [2] |
IKEDA M, MITSUDA N, OHME-TAKAGI M. Arabidopsis WUSCHEL is a bifunctional transcription factor that acts as a repressor in stem cell regulation and as an activator in floral patterning[J]. Plant Cell, 2009, 21(11): 3493-3505. DOI:10.1105/tpc.109.069997 |
| [3] |
LIU X G, KIM Y J, MÜLLER R, et al. AGAMOUS terminates floral stem cell maintenance in Arabidopsis by directly repressing WUSCHEL through recruitment of polycomb group proteins[J]. Plant Cell, 2011, 23(10): 3654-3670. DOI:10.1105/tpc.111.091538 |
| [4] |
HAECKER A, GROß-HARDT R, GEIGES B, et al. Expression dynamics of WOX genes mark cell fate decisions during early embryonic patterning in Arabidopsis thaliana[J]. Development, 2004, 131(3): 657-668. DOI:10.1242/dev.00963 |
| [5] |
SARKAR A K, LUIJTEN M, MIYASHIMA S, et al. Conserved factors regulate signalling in Arabidopsis thaliana shoot and root stem cell organizers[J]. Nature, 2007, 446(7137): 811-814. DOI:10.1038/nature05703 |
| [6] |
MINH-THU P T, KIM J S, CHAE S, et al. A WUSCHEL homeobox transcription factor, OsWOX13, enhances drought tolerance and triggers early flowering in rice[J]. Mol Cells, 2018, 41(8): 781-798. DOI:10.14348/molcells.2018.0203 |
| [7] |
ZHAO Y, HU Y, DAI M, et al. The WUSCHEL-related homeobox gene WOX11 is required to activate shoot-borne crown root development in rice[J]. Plant Cell, 2009, 21(3): 736-748. DOI:10.1105/tpc.108.061655 |
| [8] |
DAI M, HU Y, ZHAO Y, et al. A WUSCHEL-LIKE HOMEOBOX gene represses a YABBY gene expression required for rice leaf development[J]. Plant Physiol, 2007, 144(1): 380-390. DOI:10.1104/pp.107.095737 |
| [9] |
CHENG S, TAN F, LU Y, et al. WOX11 recruits a histone H3K27me3 demethylase to promote gene expression during shoot development in rice[J]. Nucl Acid Res, 2018, 46(5): 2356-2369. DOI:10.1093/nar/gky017 |
| [10] |
LIANG Y. Expression analysis of DoWOX and DoSERK in Dendrobium officinale during protocorm development [D]. Chengdu: Southwest Jiaotong University, 2018. 梁易. 铁皮石斛原球茎发育过程中DoWOX与DoSERK的表达分析[D]. 成都: 西南交通大学, 2018. |
| [11] |
LU Y, LIU Z Y, LYU M, et al. Characterization of JsWOX1 and JsWOX4 during callus and root induction in the shrub species Jasminum sambac[J]. Plants (Basel), 2019, 8(4): 79. DOI:10.3390/plants8040079 |
| [12] |
STUURMAN J, JAGGI F, KUHLEMEIER C. Shoot meristem maintenance is controlled by a GRAS-gene mediated signal from differentiating cells[J]. Genes Dev, 2002, 16(17): 2213-2218. DOI:10.1101/gad.230702 |
| [13] |
KIEFFER M, STERN Y, COOK H, et al. Analysis of the transcription factor WUSCHEL and its functional homologue in Antirrhinum reveals a potential mechanism for their roles in meristem maintenance[J]. Plant Cell, 2006, 18(3): 560-573. DOI:10.1105/tpc.105.039107 |
| [14] |
COSTANZO E, TREHIN C, VAN DEN BUSSCHE M. The role of WOX genes in flower development[J]. Ann Bot, 2014, 114(7): 1545-1553. DOI:10.1093/aob/mcu123 |
| [15] |
WEST M, HARADA J J. Embryogenesis in higher plants: An overview[J]. Plant Cell, 1993, 5(10): 1361-1369. DOI:10.1105/tpc.5.10.1361 |
| [16] |
LENHARD M, JURGENS G, LAUX T. The WUSCHEL and SHOOTMERISTEMLESS genes fulfil complementary roles in Arabidopsis shoot meristem regulation[J]. Development, 2002, 129(13): 3195-3206. DOI:10.1242/dev.129.13.3195 |
| [17] |
CARROLL S B. Homeotic genes and the evolution of arthropods and chordates[J]. Nature, 1995, 376(6540): 479-485. DOI:10.1038/376479a0 |
| [18] |
GEHRING W J, MÜLLER M, AFFOLTER M, et al. The structure of the homeodomain and its functional implications[J]. Trends Genet, 1990, 6: 323-329. DOI:10.1016/0168-9525(90)90253-3 |
| [19] |
GEHRING W J. Exploring the homeobox[J]. Gene, 1993, 135(1/2): 215-221. DOI:10.1016/0378-1119(93)90068-e |
| [20] |
ARIEL F D, MANAVELLA P A, DEZAR C A, et al. The true story of the HD-Zip family[J]. Trends Plant Sci, 2007, 12(9): 419-426. DOI:10.1016/j.tplants.2007.08.003 |
| [21] |
RICHARDT S, LANG D, RESKI R, et al. PlanTAPDB, a phylogenybased resource of plant transcription-associated proteins[J]. Plant Physiol, 2007, 143(4): 1452-1466. DOI:10.1104/pp.107.095760 |
| [22] |
PALOVAARA J, HAKMAN I. Conifer WOX-related homeodomain transcription factors, developmental consideration and expression dynamic of WOX2 during Picea abies somatic embryogenesis[J]. Plant Mol Biol, 2008, 66(5): 533-549. DOI:10.1007/s11103-008-9289-5 |
| [23] |
NARDMANN J, WERR W. The invention of WUS-like stem cellpromoting functions in plants predates leptosporangiate ferns[J]. Plant Mol Biol, 2012, 78(1/2): 123-134. DOI:10.1007/s11103-011-9851-4 |
| [24] |
VAN DER GRAAFF E, LAUX T, RENSING S A. The WUS homeobox-containing (WOX) protein family[J]. Genome Biol, 2009, 10(12): 248. DOI:10.1186/gb-2009-10-12-248 |
| [25] |
MUKHERJEE K, BÜRGLIN T R. Comprehensive analysis of animal TALE homeobox genes: New conserved motifs and cases of accelerated evolution[J]. J Mol Evol, 2007, 65(2): 137-153. DOI:10.1007/s00239-006-0023-0 |
| [26] |
JI J B, SHIMIZU R, SINHA N, et al. Analyses of WOX4 transgenics provide further evidence for the evolution of the WOX gene family during the regulation of diverse stem cell functions[J]. Plant Signal Behav, 2010, 5(7): 916-920. DOI:10.4161/psb.5.7.12104 |
| [27] |
DITENGOU F A, TEALE W D, KOCHERSPERGER P, et al. Mechanical induction of lateral root initiation in Arabidopsis thaliana[J]. Proc Natl Acad Sci USA, 2008, 105(48): 18818-18823. DOI:10.1073/pnas.0807814105 |
| [28] |
DEYHLE F, SARKAR A K, TUCKER E J, et al. WUSCHEL regulates cell differentiation during anther development[J]. Dev Biol, 2007, 302(1): 154-159. DOI:10.1016/j.ydbio.2006.09.013 |
| [29] |
VAN DENBUSSCHE M, HORSTMAN A, ZETHOF J, et al. Differential recruitment of WOX transcription factors for lateral development and organ fusion in petunia and Arabidopsis[J]. Plant Cell, 2009, 21(8): 2269-2283. DOI:10.1105/tpc.109.065862 |
| [30] |
NARDMANN J, JI J B, WERR W, et al. The maize duplicate genes narrow sheath1 and narrow sheath2 encode a conserved homeobox gene function in a lateral domain of shoot apical meristems[J]. Development, 2004, 131(12): 2827-2839. DOI:10.1242/dev.01164 |
| [31] |
SHIMIZU R, JI J B, KELSEY E, et al. Tissue specificity and evolution of meristematic WOX3 function[J]. Plant Physiol, 2009, 149(2): 841-850. DOI:10.1104/pp.108.130765 |
| [32] |
LIN H, NIU L F, MCHALE N A, et al. Evolutionarily conserved repressive activity of WOX proteins mediates leaf blade outgrowth and floral organ development in plants[J]. Proc Natl Acad Sci USA, 2013, 110(1): 366-371. DOI:10.1073/pnas.1215376110 |
| [33] |
ZHOU J, SUN J B, PENG M. The research progresses of somatic embryogenesis in higher plant[J]. Chin Agric Sci Bull, 2008, 24(2): 129-133. 高等植物体细胞胚胎发生的研究进展[J]. 中国农学通报, 2008, 24(2): 129-133. |
| [34] |
SCHMIDT E D, GUZZO F, TOONEN M A, et al. A leucine-rich repeat containing receptor-like kinase marks somatic plant cells competent to form embryos[J]. Development, 1997, 124(10): 2049-2062. DOI:10.1242/dev.124.10.2049 |
| [35] |
BIRNBAUM K D, ALVARADO A S. Slicing across kingdoms: Regeneration in plants and animals[J]. Cell, 2008, 132(4): 697-710. DOI:10.1016/j.cell.2008.01.040 |
| [36] |
DODEMAN V L, DUCREUX G, KREIS M. Zygotic embryogenesis versus somatic embryogenesis[J]. J Exp Bot, 1997, 48(313): 1493-1509. DOI:10.1093/jexbot/48.313.1493 |
| [37] |
MORDHORST A P, TOONEN M A J, DE VRIES S C. Plant embryogenesis[J]. Crit Rev Plant Sci, 1997, 16(6): 535-576. DOI:10.1080/07352689709701959 |
| [38] |
CHUGH A, KHURANA P. Gene expression during somatic embryogenesis: Recent advances[J]. Curr Sci, 2002, 83(6): 715-730. |
| [39] |
BREUNINGER H, RIKIRSCH E, HERMANN M, et al. Differential expression of WOX genes mediates apical-basal axis formation in the Arabidopsis embryo[J]. Dev Cell, 2008, 14(6): 867-876. DOI:10.1016/j.devcel.2008.03.008 |
| [40] |
WU X L, CHORY J, WEIGEL D. Combinations of WOX activities regulate tissue proliferation during Arabidopsis embryonic development[J]. Dev Biol, 2007, 309(2): 306-316. DOI:10.1016/j.ydbio.2007.07.019 |
| [41] |
ZHANG T Q, LIAN H, ZHOU C M, et al. A two-step model for de novo activation of WUSCHEL during plant shoot regeneration[J]. Plant Cell, 2017, 29(5): 1073-1087. DOI:10.1105/tpc.16.00863 |
| [42] |
LIPPMAN Z B, COHEN O, ALVAREZ J P, et al. The making of a compound inflorescence in tomato and related nightshades[J]. PLoS Biol, 2008, 6(11): e288. DOI:10.1371/journal.pbio.0060288 |
| [43] |
CHENG S F, HUANG Y L, ZHU N, et al. The rice WUSCHEL-related homeobox genes are involved in reproductive organ development, hormone signaling and abiotic stress response[J]. Gene, 2014, 549(2): 266-274. DOI:10.1016/j.gene.2014.08.003 |
| [44] |
JÜRGENS G. Apical-basal pattern formation in Arabidopsis embryogenesis[J]. EMBO J, 2001, 20(14): 3609-3616. DOI:10.1093/emboj/20.14.3609 |
| [45] |
LU P, PORAT R, NADEAU J A, et al. Identification of a meristem L1 layer-specific gene in Arabidopsis that is expressed during embryonic pattern formation and defines a new class of homeobox genes[J]. Plant Cell, 1996, 8(12): 2155-2168. DOI:10.1105/tpc.8.12.2155 |
| [46] |
WETERINGS K, APUYA N R, BI Y P, et al. Regional localization of suspensor mRNAs during early embryo development[J]. Plant Cell, 2001, 13(11): 2409-2425. DOI:10.1105/tpc.010326 |
| [47] |
ZUO J R, NIU Q W, FRUGIS G, et al. The WUSCHEL gene promotes vegetative-to-embryonic transition in Arabidopsis[J]. Plant J, 2002, 30(3): 349-359. DOI:10.1046/j.1365-313X.2002.01289.x |
| [48] |
GALLOIS J L, NORA F R, MIZUKAMI Y, et al. WUSCHEL induces shoot stem cell activity and developmental plasticity in the root meristem[J]. Genes Dev, 2004, 18(4): 375-380. DOI:10.1101/gad.291204 |
| [49] |
GALLOIS J L, WOODWARD C, REDDY G V, et al. Combined SHOOT MERISTEMLESS and WUSCHEL trigger ectopic organogenesis in Arabidopsis[J]. Development, 2002, 129(13): 3207-3217. DOI:10.1242/dev.129.13.3207 |
| [50] |
WANG F X, SHANG G D, WU L Y, et al. Chromatin accessibility dynamics and a hierarchical transcriptional regulatory network structure for plant somatic embryogenesis[J]. Dev Cell, 2020, 54(6): 742-757.E8. DOI:10.1016/j.devcel.2020.07.003 |
| [51] |
CAIRNEY J, ZHENG L, COWELS A, et al. Expressed sequence tags from loblolly pine embryos reveal similarities with angiosperm embryogenesis[J]. Plant Mol Biol, 2006, 62(4–5): 485-501. |
| [52] |
WU X L, DABI T, WEIGEL D. Requirement of homeobox gene STIMPY/WOX9 for Arabidopsis meristem growth and maintenance[J]. Curr Biol, 2005, 15(5): 436-440. DOI:10.1016/j.cub.2004.12.079 |
| [53] |
BRAND U, FLETCHER J C, HOBE M, et al. Dependence of stem cell fate in Arabidopsis on a feedback loop regulated by CLV3 activity[J]. Science, 2000, 289(5479): 617-619. DOI:10.1126/science.289.5479.617 |
| [54] |
SCHOOF H, LENHARD M, HAECKER A, et al. The stem cell population of Arabidopsis shoot meristems is maintained by a regulatory loop between the CLAVATA and WUSCHEL genes[J]. Cell, 2000, 100(6): 635-644. DOI:10.1016/s0092-8674(00)80700-x |
| [55] |
WAITES R, SIMON R. Signaling cell fate in plant meristems: Three clubs on one tousle[J]. Cell, 2000, 103(6): 835-838. DOI:10.1016/s0092-8674(00)00186-0 |
| [56] |
HADFI K, SPETH V, NEUHAUS G. Auxin-induced developmental patterns in Brassica juncea embryos[J]. Development, 1998, 125(5): 879-887. DOI:10.1242/dev.125.5.879 |
| [57] |
HE G M, ELLING A A, DENG X W. The epigenome and plant development[J]. Annu Rev Plant Biol, 2011, 62: 411-435. DOI:10.1146/annurev-arplant-042110-103806 |
| [58] |
ZHANG C L, WANG J F, WANG X, et al. UF, a WOX gene, regulates a novel phenotype of un-fused flower in tomato[J]. Plant Sci, 2020, 297: 110523. DOI:10.1016/j.plantsci.2020.110523 |
| [59] |
DEVEAUX Y, TOFFANO-NIOCHE C, CLAISSE G, et al. Genes of the most conserved WOX clade in plants affect root and flower development in Arabidopsis[J]. BMC Evol Biol, 2008, 8: 291. DOI:10.1186/1471-2148-8-291 |
| [60] |
MATSUMOTO N, OKADA K. A homeobox gene, PRESSED FLOWER, regulates lateral axis-dependent development of Arabidopsis flowers[J]. Genes Dev, 2001, 15(24): 3355-3364. DOI:10.1101/gad.931001 |
| [61] |
SOUER E, VAN DER KROL A, KLOOS D, et al. Genetic control of branching pattern and floral identity during Petunia inflorescence development[J]. Development, 1998, 125(4): 733-742. DOI:10.1242/dev.125.4.733 |
| [62] |
SCANLON M J, FREELING M. The narrow sheath leaf domain deletion: A genetic tool used to reveal developmental homologies among modified maize organs[J]. Plant J, 1998, 13(4): 547-561. |
| [63] |
REBOCHO A B, BLIEK M, KUSTERS E, et al. Role of EVERGREEN in the development of the cymose petunia inflorescence[J]. Dev Cell, 2008, 15(3): 437-447. DOI:10.1016/j.devcel.2008.08.007 |
| [64] |
GALUN E. Patterning of flowers and their members [M]// GALUN E. Plant Patterning. Singapore: World Scientific, 2007: 383–440. doi: 10.1142/9789812778840_0012.
|
| [65] |
ZHANG T, LI R N, XING J L, et al. The YUCCA-Auxin-WOX11 module controls crown root development in rice[J]. Front Plant Sci, 2018, 9: 523. DOI:10.3389/fpls.2018.00523 |
| [66] |
LIU J C, SHENG L H, XU Y Q, et al. WOX11 and 12 are involved in the first-step cell fate transition during de novo root organogenesis in Arabidopsis[J]. Plant Cell, 2014, 26(3): 1081-1093. DOI:10.1105/tpc.114.122887 |
| [67] |
SUER S, AGUSTI J, SANCHEZ P, et al. WOX4 imparts auxin responsiveness to cambium cells in Arabidopsis[J]. Plant Cell, 2011, 23(9): 3247-3259. DOI:10.1105/tpc.111.087874 |
| [68] |
ZHAO S, JIANG Q T, MA J, et al. Characterization and expression analysis of WOX5 genes from wheat and its relatives[J]. Gene, 2014, 537(1): 63-69. DOI:10.1016/j.gene.2013.12.022 |
| [69] |
IOIO R D, LINHARES F S, SABATINI S. Emerging role of cytokinin as a regulator of cellular differentiation[J]. Curr Opin Plant Biol, 2008, 11(1): 23-27. DOI:10.1016/j.pbi.2007.10.006 |
| [70] |
DE SMET I, JÜRGENS G. Patterning the axis in plants–auxin in control[J]. Curr Opin Genet Dev, 2007, 17(4): 337-343. DOI:10.1016/j.gde.2007.04.012 |
| [71] |
INUKAI Y, SAKAMOTO T, UEGUCHI-TANAKA M, et al. Crown rootless1, which is essential for crown root formation in rice, is a target of an AUXIN RESPONSE FACTOR in auxin signaling[J]. Plant Cell, 2005, 17(5): 1387-1396. DOI:10.1105/tpc.105.030981 |
| [72] |
MENG W J, CHENG Z J, SANG Y L, et al. Type-B ARABIDOPSIS RESPONSE REGULATORs specify the shoot stem cell niche by dual regulation of WUSCHEL[J]. Plant Cell, 2017, 29(6): 1357-1372. DOI:10.1105/tpc.16.00640 |
| [73] |
LIU J, HU X M, QIN P, et al. The WOX11-LBD16 pathway promotes pluripotency acquisition in callus cells during de novo shoot regeneration in tissue culture[J]. Plant Cell Physiol, 2018, 59(4): 739-748. DOI:10.1093/pcp/pcy010 |
| [74] |
LIU L, WANG Y, SHI Z Y, et al. Advances in plant stem cell culture[J]. Chin J Biotechnol, 2018, 34(11): 1734-1741. 植物干细胞培养研究进展[J]. 生物工程学报, 2018, 34(11): 1734-1741. DOI:10.13345/j.cjb.180047 |
| [75] |
MAYER K F X, SCHOOF H, HAECKER A, et al. Role of WUSCHEL in regulating stem cell fate in the Arabidopsis shoot meristem[J]. Cell, 1998, 95(6): 805-815. DOI:10.1016/s0092-8674(00)81703-1 |
| [76] |
LEIBFRIED A, TO J P C, BUSCH W, et al. WUSCHEL controls meristem function by direct regulation of cytokinin-inducible response regulators[J]. Nature, 2005, 438(7071): 1172-1175. DOI:10.1038/nature04270 |
| [77] |
ZHOU Y, LIU X, ENGSTROM E M, et al. Control of plant stem cell function by conserved interacting transcriptional regulators[J]. Nature, 2015, 517(7534): 377-380. DOI:10.1038/nature13853 |
| [78] |
SU Y H, ZHOU C, LI Y J, et al. Integration of pluripotency pathways regulates stem cell maintenance in the Arabidopsis shoot meristem[J]. Proc Natl Acad Sci USA, 2020, 117(36): 22561-22571. DOI:10.1073/pnas.2015248117 |
| [79] |
STAHL Y, WINK R H, INGRAM G C, et al. A signaling module controlling the stem cell niche in Arabidopsis root meristems[J]. Curr Biol, 2009, 19(11): 909-914. DOI:10.1016/j.cub.2009.03.060 |
| [80] |
CHANDLER J, NARDMANN J, WERR W. Plant development revolves around axes[J]. Trends Plant Sci, 2008, 13(2): 78-84. DOI:10.1016/j.tplants.2007.11.010 |
| [81] |
IWASAKI M, PASZKOWSKI J. Epigenetic memory in plants[J]. EMBO J, 2014, 33(18): 1987-1098. DOI:10.15252/embj.201488883 |
| [82] |
SERMAN A, VLAHOVIĆ M, SERMAN L, et al. DNA methylation as a regulatory mechanism for gene expression in mammals[J]. Coll Antropol, 2006, 30(3): 665-671. |
| [83] |
XING L B, LI Y M, QI S Y, et al. Comparative RNA-sequencing and DNA methylation analyses of apple (Malus domestica Borkh.) buds with diverse flowering capabilities reveal novel insights into the regulatory mechanisms of flower bud formation[J]. Plant Cell Physiol, 2019, 60(8): 1702-1721. DOI:10.1093/pcp/pcz080 |
| [84] |
XING L B, QI S Y, ZHOU H, et al. Epigenomic regulatory mechanism in vegetative phase transition of Malus hupehensis[J]. J Agric Food Chem, 2020, 68(17): 4812-4829. DOI:10.1021/acs.jafc.0c00478 |
| [85] |
LI W, LIU H, CHENG Z J, et al. DNA methylation and histone modifications regulate De novo shoot regeneration in Arabidopsis by modulating WUSCHEL expression and auxin signaling[J]. PLoS Genet, 2011, 7(8): e1002243. DOI:10.1371/journal.pgen.1002243 |
| [86] |
LIAN G B, DING Z W, WANG Q, et al. Origins and evolution of WUSCHEL-related homeobox protein family in plant kingdom[J]. Sci World J, 2014(2014): 534140. DOI:10.1155/2014/534140 |
| [87] |
BERTRAND C, BERGOUNIOUX C, DOMENICHINI S, et al. Arabidopsis histone acetyltransferase AtGCN5 regulates the floral meristem activity through the WUSCHEL/AGAMOUS pathway[J]. J Biol Chem, 2003, 278(30): 28246-28251. DOI:10.1074/jbc.M302787200 |
| [88] |
SALVINI M, FAMBRINI M, GIORGETTI L, et al. Molecular aspects of zygotic embryogenesis in sunflower (Helianthus annuus L.): Correlation of positive histone marks with HaWUS expression and putative link HaWUS/HaL1L[J]. Planta, 2016, 243(1): 199-215. DOI:10.1007/s00425-015-2405-2 |
| [89] |
PI L M, AICHINGER E, VAN DER GRAAFF E, et al. Organizerderived WOX5 signal maintains root columella stem cells through chromatin-mediated repression of CDF4 expression[J]. Dev Cell, 2015, 33(5): 576-588. DOI:10.1016/j.devcel.2015.04.024 |
| [90] |
IKEUCHI M, IWASE A, SUGIMOTO K. Control of plant cell differentiation by histone modification and DNA methylation[J]. Curr Opin Plant Biol, 2015, 28: 60-67. DOI:10.1016/j.pbi.2015.09.004 |
 2022, Vol. 30
2022, Vol. 30



