水稻(Oryza sativa)是最重要的粮食作物,其产量约占世界粮食总产量的1/3[1]。然而,随着全球气候变暖,极端天气发生日趋频繁,低温冷害已成为限制水稻安全生产的主要因子[2]。据统计,全世界每年约有1.50×107 hm2的水稻在不同生长时期遭受寒害或冷害,轻者导致秧苗黄化,生长迟钝;重者出现烂秧、烂芽或死苗,甚至颗粒无收[3]。我国每年因低温冷害导致水稻减产5.0×109~1.00× 1010 kg[2]。因此,研究水稻耐冷机制,挖掘水稻遗传潜能,培育水稻耐冷新品种具有重要的理论和实践意义。
东乡野生稻(O. rufipogon)是全球分布最北的普通野生稻,蕴藏着强耐冷、抗旱和高产等优异基因。强耐冷性是其最显著的特点,苗期和抽穗期的耐冷性均强于粳稻,地下茎能耐受–12.8℃而安全越冬[4–5]。因此研究和利用东乡野生稻的强耐冷性受到广泛关注。前人在东乡野生稻的耐冷性鉴定评价、数量性状基因位点(quantitative trait locus, QTL)分析和差异表达谱等方面已做了大量工作[5–7],然而,东乡野生稻的耐冷性遗传机理非常复杂,不同研究群体和不同鉴定方法所检测的耐冷QTL数目、染色体区域和效应大小有较大差异,可比性较低,东乡野生稻耐冷的分子调控机制仍然难以琢磨[8–9]。因此,进一步全面深入探讨东乡野生稻耐冷基因的调控网路及分子机制,具有重要的理论价值和现实意义。
转录水平的调控是生物体很重要的一种调控方式,与基因组学相比具有针对性强、研究范围小的优点且被认为是一种在转录水平上更精准的分析方法,鉴于此,本研究在前期研究的基础上[10], 利用转录组学测序技术(RNA-Seq)分析其东乡野生稻低温胁迫下和常温培养条件下的基因表达谱特征,获取差异表达基因信息数据。采用qRT-PCR方法分析、验证差异表达基因的表达特性。将转录组的基因表达谱数据与报道的水稻耐冷相关的QTL结果进行联合分析,为挖掘东乡野生稻耐冷相关基因、阐明东乡野生稻响应低温胁迫的应答机制提供科学依据。
1 材料和方法 1.1 材料供试材料为中国江西省东乡县的东乡野生稻(Oryza rufipogon)。将种子置于50℃烘箱中干燥3 d,挑选50粒成熟饱满的种子用3% NaClO消毒15 min, 清洗3遍,于28℃浸种3 d催芽,将露白种子均匀播种于垫有滤纸的90 mm×10 mm培养皿中,待胚芽长至8~12 mm后置于28℃、44.9 μmol/(m2·s)、光照14 h/d的光照培养箱中培养,加入MS营养液, 每隔5 d更换1次。待苗长至3叶1心,除去弱苗,记录每皿中成苗数。将幼苗分成两组,一组为对照(DY1、DY2和DY3)置于28℃下正常培养;另一组为处理组(DYL1、DYL2和DYL3)置于8℃冷胁迫处理72 h,每组均3次生物学重复。处理结束后, 取叶片于液氮中冻存,置于–80℃冰箱中保存备用。
1.2 方法RNA提取、文库构建 采用Thermo Fisher Scientific公司的Trizol试剂盒,参照Qiao等[9]的方法提取东乡野生稻幼苗的RNA,用琼脂糖凝胶电泳分析RNA的完整性和纯净度,委托北京诺禾致源生物信息科技有限公司完成RNA的质控,建库合格后上机(Illumina HiSeq)进行测序工作。
测序数据分析 测序片段经CASAVA碱基识别转化为Fastq格式序列数据(Reads),其中包含测序片段的序列信息和其对应的测序质量信息。对原始数据进行过滤,去除接头、含N、低质量Reads后得到Clean reads,然后计算Q20、Q30和GC含量。从基因组网站下载基因模型注释文件和参考基因组(https://www.ncbi.nlm.nih.gov/gene/),构建参考基因组的索引,将Clean reads与参照基因组比对。采用String Tie拼接出转录本后,利用Feature Counts计算映射到每个基因的读数,然后根据基因的长度计算每个基因的FPKM和映射到该基因的读数,利用DESeq2分析两组处理间的差异表达。本试验以P < 0.05以及|log2FoldChange| > 1作为差异表达基因的筛选阈值。通过clusterProfiler R软件实现差异表达基因的GO富集分析和KEGG通路差异表达基因的统计富集。
差异表达基因的qRT-PCR验证 参照Luo等[11]的方法,用Trizol试剂提取RNA后,按照反转录试剂盒First-Strand Synthesis of cDNA Kit (普洛麦格生物技术有限公司)说明书进行cDNA第一链的合成,根据CDS (编码序列)设计后续qRT-PCR的特异引物,所有引物由北京鼎国昌盛生物技术有限责任公司合成(附表),按照SYBR Premix Ex Taq Ⅱ (2×)(TaKaRa北京生物技术有限公司)说明书,在荧光定量PCR仪(美国Thermo Fisher Scien- tific公司)检测12个差异表达基因的表达情况, 以Cyclophilin作为内参基因。PCR反应体系总体积为20 μL,其中Taq聚合酶10 μL, 上、下游引物各0.4 μL,模板2 μL,灭菌水7.2 μL,每个样品均3次重复。反应程序为:95℃预变性5 min,然后95℃ 30 s,65℃ 30 s,共40个循环,相对表达量以2–ΔΔCT计算。
2 结果和分析 2.1 转录组测序结果和质控分析对东乡野生稻幼苗提取RNA后进行转录组测序, 获得样品原始数据序列Reads;舍弃低质量数据, 经序列拼接,最后获得Clean reads为47 041 126~ 51 358 394,占总数的93%~97%,错误率仅为0.03, Q20均高于95%,Q30均高于89%,GC含量约为52% (表 1),表明东乡野生稻幼苗叶片的转录测序质量良好。
| 表 1 转录组测序数据质量检测 Table 1 Quality test of transcriptome sequencing data |
低温胁迫下对低温敏感的基因会发生表达变化,表达差异程度可以反映其在低温胁迫过程中的活跃度。对差异表达基因(differentially expressed gene, DEG)数量进行统计学分析表明(图 1),与DY相比,DYL中P < 0.05、相对表达水平绝对值|log2FoldChange| > 1,即表达差异|log2FC|大于2倍的DEGs有10 200个,上调表达的有5 201个,占50.99%,下调表达的有4 999个,占49.01%。这些基因表达差异幅度较大, |log2FC|主要集中在10以内,占比90.18%; |log2FC| > 10的DEGs有24个, 其中21个上调表达, 3个下调表达, 分布在1号染色体上的有2个,3号染色体有6个,4号染色体有2个,5号染色体有4个,11号染色体有5个, 第2、6、8、10、12号染色体各1个。
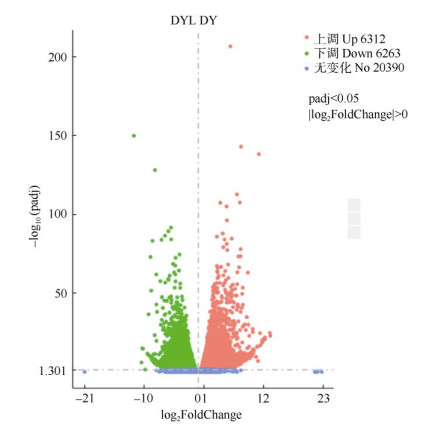
|
图 1 基因表达火山图 Fig. 1 Volcano map of gene expression |
从表 2可以看出,表达上调的基因主要与激素信号转导因子(脱落酸ABA、乙烯ETH、漆酶LAC)、膜蛋白等相关,下调的基因与乙醛脱氢、甲基转移、稻谷粒长相关。
| 表 2 |log2FC| > 10的显著差异表达基因 Table 2 Significant differentially expressed genes with |log2FC| > 10 |
为进一步分析与耐冷相关的差异基因,将|log2FC| > 1的DEGs与已知耐冷QTL进行联合匹配分析,结果表明,共有426个DEGs可以map到2个以上研究者报道的QTL区间,其中1号染色体上有61个,3号染色体有36个,4号染色体有50个,5号染色体有156个,6号染色体有21个, 7号染色体有69个,8号染色体有33个。
研究表明WRKY、AP2/ERF、MYB、bZIP、HSF等家族基因在调控水稻的生长发育和耐逆性中起着重要作用[12–15]。本研究结果表明,位于耐冷QTL区间内的426个DEGs中,有37个为耐冷调控相关的家族基因(表 3)。在经过低温胁迫后, WRKY、AP2/ERF、PP2C、bZIP、HSP70、HSF家族基因和低温调节蛋白基因的DEGs都上调表达, 而ABC转运蛋白家族基因则下调表达,其余家族基因均有上调和下调表达的基因(表 3);还有89个包括AOX、petidase-S8、p450等与植物耐逆性相关的其他家族基因。此外,还检测到1个|log2FC| > 10且功能尚未报道的DEG (Os04g0121800)。
| 表 3 耐冷QTL区间内的家族基因 Table 3 Family genes located in the QTL loci of cold tolerance |
GO分析表明(表 4),|log2FC| > 1的DEGs富集到896条GO注释中,其中生物过程(biological process, BP)占48.19%,细胞组分(cellular component, CC)占12.72%, 分子功能(molecular function, MF)占39.07%, 说明大多数差异表达基因与一些生物学功能显著相关。BP中主要富集在有机氮化合物生物合成过程(GO: 1901566,251个)、碳水化合物代谢过程(GO: 0005975, 217个)、小分子代谢过程(GO: 0044281, 194个)和细胞酰胺代谢过程(GO: 0043603, 188个)等4个方面;在CC中主要富集在细胞质(GO: 0005737, 272个)、胞质部分(GO: 0044444, 228个)和无膜细胞器(GO: 0043228, 168个)等3个方面; 在MF中主要富集在跨膜转运体活动(GO: 0022857, 197个)、核酸结合转录因子活性(GO: 0001071, 190个)等2个方面。这表明在低温胁迫下,东乡野生稻进行着十分复杂的新陈代谢,涉及有机氮化合物生物合成、细胞质、胞质部分和碳水化合物代谢等生物学功能方面的基因较为活跃。
| 表 4 差异表达基因的GO注释 Table 4 Differentially expressed genes by GO annotation |
为探索低温胁迫下东乡野生稻代谢途径的变化,揭示DEGs的生物学功能,对DEGs进行KEGG代谢途径富集分析表明(图 3),DEGs共富集到113条信号通路上,主要富集在核糖体(ribosome, dosa 03010)、真核生物的核糖体生物发生(ribosome bio- genesis in eukaryotes, dosa03008)、氨基酸的生物合成(biosynthesis of amino acids, dosa01230)、光合作用(photosynthesis, dosa00195)等通路上, 其中核糖体通路富集的DEGs最多。前3条通路都与蛋白质的合成相关,且大部分DEGs都上调。核糖体通路中167个DEGs上调;真核生物中核糖体生物发生通路中有58个DEGs上调;氨基酸的生物合成通路中有78个DEGs上调。这表明低温胁迫下, 东乡野生稻蛋白质的合成大幅提高,在响应低温时可能发挥着重要作用。
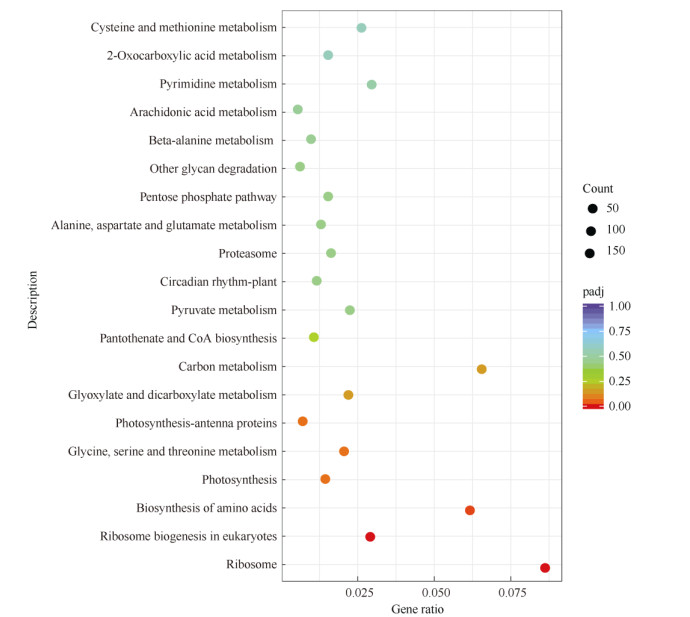
|
图 2 DEGs的KEGG通路富集散点图 Fig. 2 Scatter diagram of pathway enrichment analysis of DEGs |
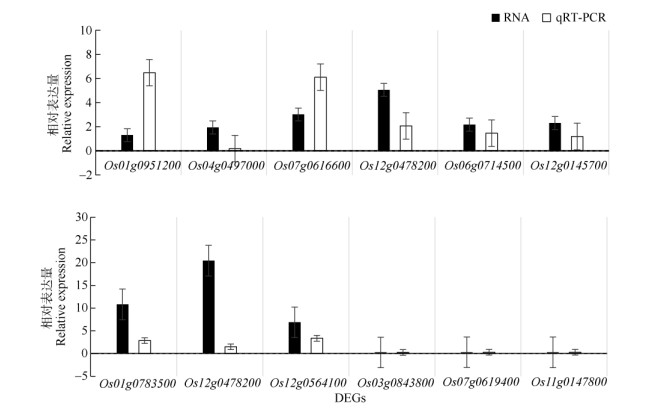
|
图 3 部分DEGs的RNA-Seq和qRT-PCR的表达模式比较 Fig. 3 Comparison of expression levels of some DEGs by RNA-Seq and qRT-PCR |
为了挖掘与验证东乡野生稻响应低温胁迫信号网络相关基因,本文随机选取了包括ABA响应蛋白基因、MYB转录因子和40S核糖体蛋白基因SA等可能参与响应低温胁迫的12个DEGs,对其转录水平的表达量进行qRT-PCR验证(图 4),绝大部分基因的qRT-PCR表达水平表现出与转录组RNA- Seq相同的变化趋势,其中9个DEGs的qRT-PCR和RNA-Seq表达水平为上调,分别是Os01g0951200 (尿苷-磷酸合成酶基因)、Os04g0497000 (NADPH氧化还原酶基因)、Os07g0616600 (40S核糖体蛋白基因SA)、Os12g0478200 (ABA响应蛋白基因)、Os06g 0714500 (假定spastin蛋白基因)、Os12g0145700 (丙酮酸激酶基因)、Os01g0783500 (结构域蛋白基因)、Os12g0147800 (植物磺肽激素基因)和Os12g0564100 (MYB转录因子)。有3个DEGs的qRT-PCR和RNA- Seq表达水平都下调,分别是Os03g0843800 (硫醇甲基转移酶基因)、Os07g0619400 (结构域蛋白基因)和Os11g0147800 (特异性茎蛋白基因TSJT1)。从图5可见,DEGs的qRT-PCR和RNA-Seq表达差异倍数不同但表达趋势相同,这表明qRT-PCR验证结果和转录组测序结果一致。这些基因在低温胁迫下发生表达变化, 预示其在东乡野生稻耐低温胁迫中可能发挥着重要作用。
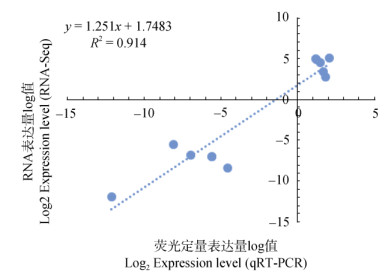
|
图 4 DEGs基因的RNA-Seq和qRT-PCR表达相关性 Fig. 4 Relationship of expression level of DEGs between RNA-Seq and qRT-PCR |
转录组测序技术能够全面且高效分析植物组织或细胞在物种、品种、发育阶段和环境下,全部转录产物的类别、结构及表达水平的变化,揭示特定生物学过程的分子调控机制[15]。目前植物的耐冷研究中,许多研究者利用该技术鉴定并成功揭示了包括拟南芥(Arabidopsis thaliana)、水稻、小麦(Triticum aestivum)等几十种重要作物的耐冷关键基因和复杂的低温胁迫响应机制[18],为作物的分子育种奠定了重要基础。
本研究采用RNA-seq技术对东乡野生稻苗期叶片在常温与低温诱导下的全转录组测序,比较了两者完整的差异基因表达谱。结果表明,与常温相比,东乡野生稻苗期叶片经低温诱导后,上调表达的DEGs有5 201个,比下调表达的多1.98%; GO功能分类和KEGG Pathway通路分析表明,DEGs主要富集在核糖体结合转录因子活性、氨基酸的生物合成和光合作用等代谢过程中,这表明东乡野生稻在面临低温时植物体内时刻发生着复杂的新陈代谢变化,通过改变次级代谢物合成、环境应激来响应低温胁迫,这与前人[19–21]对水稻苗期耐冷品种的转录组研究结果类似。
众多研究表明,植物激素信号转导途径(生长素IAA、脱落酸ABA、乙烯ETH等激素)对水稻苗期低温耐受性调控起重要作用[22–23]。很多研究表明, IAA反应因子(ARF)、ABA激活蛋白激酶、ETH信号转导因子在水稻中过度表达可以提高低温耐受力[24–25]。低温胁迫下,水稻内源ABA升高,其受体PYP/PYL/RCAR蛋白与ABA结合并与蛋白激酶PP2C互作,抑制蛋白激酶SnRK2与PP2C结合, SnRK2便能磷酸化转录因子,磷酸化后的转录因子激活ABA响应基因,进而提高对低温的耐受性[26]。本研究检测到了3个ETH信号转导因子(AP2/ERF家族基因)以及16个与ABA相关的基因(蛋白激酶Pkinase家族基因11个、蛋白磷酸酶PP2C家族基因4个上调表达、ABA响应蛋白1个)和1个IAA反应因子(ARF家族基因Os06g0702600),这些基因位于前人耐冷QTL区间内,并且在遭受低温胁迫后大量诱导表达,促使体内生理生化发生改变,帮助水稻抵抗寒冷,除此之外,我们还检测到2个ETH信号转导因子(ERF1和OsERF09), 前人也有研究报道。ABA响应蛋白(Os12g0478200)在qRT- PCR验证后表明,qRT-PCR和转录组测序RNA-seq的表达水平一致,暗示着这些植物激素信号传导的相关基因可能在东乡野生稻响应低温胁迫的过程中发挥着重要的作用。
有研究也表明,转录因子及其相关的调控基因对植物的耐寒性至关重要,它可以调控酶、蛋白和代谢物[27–30]。目前已有8类转录因子家族(锌指b- ZIP、MYB、AP2/ERF、BHLH、NAC、Bzip、WRKY和HSF)的转录因子基因被识别,低温诱导的最大转录因子家族组属于锌指家族[12–15]。锌指蛋白基因表达增强可提高作物对低温的耐受能力[31–32],水稻锌指蛋白基因ZFP177转入烟草均可增强转基因植株耐受低温胁迫的能力[33],许多WRKY家族转录因子影响着水稻的低温耐受性,过表达WRKY转录因子的转基因水稻株系对冷胁迫的抗性显著增强[34–35], MYB转录因子参与了植物的低温响应调控过程, 但是,不同的MYB成员所起的作用并不相同。本研究检测到1个b-ZIP、5个MYB_DNA-binding、3个WRKY和1个HSF家族基因及3个锌指蛋白基因,且这些基因都位于耐冷QTL区间内,此外,还检测到5个b-ZIP转录因子和16个WRKY家族基因, 也与前人研究结果一致。这些转录因子相关的调控基因不仅位于耐冷QTL区间内而且属于耐冷调控相关的家族,极有可能参与了应答低温胁迫的信号刺激。qRT-PCR验证表明MYB转录因子(Os01g0867300)在冷胁迫后上调表达,与转录组结果一致。这表明转录因子相关的调控基因极有可能参与了应答低温环境的信号刺激, 被诱导表达,进而提高水稻的抗逆能力。
水稻在遇到低温胁迫时会发生一系列生理生化和分子机制的变化,以响应、适应环境温度的变化,来增强自身的耐冷性,维持并稳定生长。本研究以水稻耐冷品种东乡野生稻为材料,对低温胁迫下叶片的转录组进行测序,获得了较为完善的转录本信息。GO和KEGG分析表明,核酸结合转录因子、氨基酸生物合成和光合作用代谢等均参与响应低温胁迫过程。同时,也检测到植物激素信号传导基因和转录因子。此外,ABA响应蛋白和MYB转录因子的qRT-PCR表达谱和转录组测序的表达变化一致,是否可以推测在东乡野生稻苗期遭受低温信号刺激时,植物激素信号传导途径相关基因与转录因子相关调控基因被上调诱导表达,从而提高了东乡野生稻的抗寒能力,在其响应低温胁迫的过程中起着关键作用。
| [1] |
ZHAO J L, ZHANG S H, YANG T F, et al. Global transcriptional profiling of a cold-tolerant rice variety under moderate cold stress reveals different cold stress response mechanisms[J]. Physiol Plant, 2015, 154(3): 381-394. DOI:10.1111/ppl.12291 |
| [2] |
LIU J Y, YAN S Y, ZHANG R X, et al. Low temperature tolerance of rice: A review[J]. Chin Sci Bull, 2020, 36(27): 1-5. 刘静妍, 闫双勇, 张融雪, 等. 水稻耐低温研究重要进展[J]. 中国农学通报, 2020, 36(27): 1-5. |
| [3] |
LIU C T, WANG W, MAO B G, et al. Cold stress tolerance in rice: Physiological changes, molecular mechanism, and future prospects[J]. Hereditas, 2018, 40(3): 171-185. 刘次桃, 王威, 毛毕刚, 等. 水稻耐低温逆境研究: 分子生理机制及育种展望[J]. 遗传, 2018, 40(3): 171-185. DOI:10.16288/j.yczz.18007 |
| [4] |
QIU Z H, HU B L, XIAO Y Q, et al. Research advances on important stress tolerant traits and its molecular mechanisms in Dongxiang wild rice[J]. China Rice, 2017, 23(5): 12-18. 邱在辉, 胡标林, 肖叶青, 等. 东乡野生稻重要耐逆性状及其分子机制研究进展[J]. 中国稻米, 2017, 23(5): 12-18. DOI:10.3969/j.issn.1006-8082.2017.05.003 |
| [5] |
MAO D H, YU L, CHEN D Z, et al. Multiple cold resistance loci confer the high cold tolerance adaptation of Dongxiang wild rice (Oryza rufipogon) to its high-latitude habitat[J]. Theor Appl Genet, 2015, 128(7): 1359-1371. DOI:10.1007/s00122-015-2511-3 |
| [6] |
ZHAO J, QIN J J, SONG Q, et al. Combining QTL mapping and expression profile analysis to identify candidate genes of cold tolerance from Dongxiang common wild rice (Oryza rufipogon Griff.)[J]. J Inter Agric, 2016, 15(9): 1933-1943. DOI:10.1016/S2095-3119(15)61214-X |
| [7] |
LUO X D, DAI L F, CAO J F, et al. Identification and molecular cytology analysis of cold tolerance introgression lines derived from Oryza sativa L. mating with O. rufipogon Griff[J]. Euphytica, 2012, 187(3): 461-469. DOI:10.1007/s10681-012-0769-y |
| [8] |
SHEN C X, LI D, HE R H, et al. Comparative transcriptome analysis of RNA-seq data for cold-tolerant and cold-sensitive rice genotypes under cold stress[J]. Plant Biol, 2014, 57(6): 337-348. DOI:10.1007/s12374-014-0183-1 |
| [9] |
XIAO N, HUANG W N, LI A H, et al. Fine mapping of the qLOP2 and qPSR2-1 loci associated with chilling stress tolerance of wild rice seedlings[J]. Theor Appl Genet, 2015, 128(1): 173-185. DOI:10.1007/s00122-014-2420-x |
| [10] |
SHI D H, WANG J Y, BAI Y, et al. Transcriptome analysis of Chinese Ficus tikoua[J]. J Trop Subtrop Bot, 2020, 28(4): 317-328. 石登红, 王继玥, 白禹, 等. 地果的转录组学分析[J]. 热带亚热带植物学报, 2020, 28(4): 317-328. DOI:10.11926/jtsb.4164 |
| [11] |
LUO X D, LIU J, ZHAO J, et al. Rapid mapping of candidate genes for cold tolerance in Oryza rufipogon Griff. by QTL-seq of seedlings[J]. J Integr Agric, 2018, 17(2): 265-275. DOI:10.1016/S2095-3119(17)61712-X |
| [12] |
JOO J, LEE Y H, SONG S I. OsbZIP42 is a positive regulator of ABA signaling and confers drought tolerance to rice[J]. Planta, 2019, 249(5): 1521-1533. DOI:10.1007/s00425-019-03104-7 |
| [13] |
HARTMANN L, PEDROTTI L, WEISTE C, et al. Crosstalk between two bZIP signaling pathways orchestrates salt-Induced metabolic reprogramming in Arabidopsis roots[J]. Plant Cell, 2015, 27(8): 22442260. DOI:10.1105/tpc.15.00163 |
| [14] |
SUN X L, LI Y, CAI H, et al. The Arabidopsis AtbZIP1 transcription factor is a positive regulator of plant tolerance to salt, osmotic and drought stresses[J]. J Plant Res, 2012, 125(3): 429-438. DOI:10.1007/s10265-011-0448-4 |
| [15] |
PAN Y L, HU X, LI C Y, et al. SlbZIP38, a Tomato bZIP family gene downregulated by abscisic acid, is a negative regulator of drought and salt stress tolerance[J]. Genes, 2017, 8(12): 402. DOI:10.3390/genes8120402 |
| [16] |
JIA C L, ZHANG Y, ZHU L, et al. Application progress of transcripttome sequencing technology in biological sequencing[J]. Mol Plant Breed, 2015, 13(10): 2388-2394. 贾昌路, 张瑶, 朱玲, 等. 转录组测序技术在生物测序中的应用研究进展[J]. 分子植物育种, 2015, 13(10): 2388-2394. DOI:10.13271/j.mpb.013.002388 |
| [17] |
JIANG K Y, ZHOU M B. Recent advances in bamboo molecular biology[J]. J Trop Subtrop Bot, 2014, 22(6): 632-642. 姜可以, 周明兵. 竹子分子生物学研究进展[J]. 热带亚热带植物学报, 2014, 22(6): 632-642. DOI:10.11926/j.issn.1005-3395.2014.06.012 |
| [18] |
ZHANG J, TANG L, RAN Q F, et al. Advances in RNA sequencing in response to low temperature stress in plants[J]. Mol Plant Breed, 2020, 18(6): 1849-1866. 张健, 唐露, 冉启凡, 等. 植物响应低温胁迫转录组测序研究进展[J]. 分子植物育种, 2020, 18(6): 1849-1866. DOI:10.13271/j.mpb.018.001849 |
| [19] |
ZHU L, YUAN M, GAO H X, et al. Transcriptomic analysis of rice seedling responsive to low temperature[J]. Acta Agric Boreali-Sin, 2018, 33(5): 40-51. 朱琳, 袁梦, 高红秀, 等. 水稻苗期低温应答转录组分析[J]. 华北农学报, 2018, 33(5): 40-51. |
| [20] |
GUO H, LI S X, SUN P Y, et al. Transcriptomes analysis of the cold tolerance difference of rice seedlings[J]. Mol Plant Breed, 2020, 18(6): 1731-1739. 郭慧, 李树杏, 孙平勇, 等. 水稻苗期耐冷性差异的转录组分析[J]. 分子植物育种, 2020, 18(6): 1731-1739. DOI:10.13271/j.mpb.018.001731 |
| [21] |
ZHANG T, ZHAO X Q, WANG W S, et al. Comparative transcriptome profiling of chilling stress responsiveness in two contrasting rice genotypes[J]. PLoS One, 2012, 7(8): e43274. DOI:10.1371/journal.pone.0043274 |
| [22] |
LIU F. Effects of low temperature on physiological traits and transcriptome of Casuarina cunninghamiana Miq. clones[D]. Changsha: Central South University of Forestry & Technology, 2015: 5-7. 刘芬. 低温胁迫对细枝木麻黄无性系生理指标和转录组的影响[D]. 长沙: 中南林业科技大学, 2015: 5-7. |
| [23] |
JAIN M, KHURANA J P. Transcript profiling reveals diverse roles of auxin-responsive genes during reproductive development and abiotic stress in rice[J]. FEBS J, 2009, 276(11): 3148-3162. DOI:10.1111/j.1742-4658.2009.07033.x |
| [24] |
ZHAO Y D, DAI X H, BLACKWELL H E, et al. SIR1, an upstream component in auxin signaling identified by chemical genetics[J]. Science, 2003, 301(5636): 1107-1110. DOI:10.1126/science.1084161 |
| [25] |
ZHU J K. Abiotic stress signaling and responses in plants[J]. Cell, 2016, 167(2): 313-324. DOI:10.1016/j.cell.2016.08.029 |
| [26] |
ZHANG L, LI X Y, LI D K, et al. CARK1 mediates ABA signaling by phosphorylation of ABA receptors[J]. Cell Discov, 2018, 4(1): 30. DOI:10.1038/s41421-018-0029-y |
| [27] |
SAH S K, REDDY K R, LI J X. Abscisic acid and abiotic stress tolerance in crop plants[J]. Front Plant Sci, 2016, 7: 571. DOI:10.3389/fpls.2016.00571 |
| [28] |
LIU Q Q, ZHENG L, HE F, et al. Transcriptional and physiological analyses identify a regulatory role for hydrogen peroxide in the lignin biosynthesis of copper-stressed rice roots[J]. Plant Soil, 2015, 387(1/2): 323-336. DOI:10.1007/s11104-014-2290-7 |
| [29] |
WANG L N. The WRKY family gene and its response to cold stress in grape[D]. Wuhan: Wuhan Botanical Garden, Chinese Academy of Sciences, 2014: 10-18. 王莉娜. WRKY基因家族的鉴定及其在葡萄冷胁迫中的功能分析[D]. 武汉: 中国科学院研究生院, 2014: 10-18. |
| [30] |
JOO J, LEE Y H, SONG S I. OsbZIP42 is a positive regulator of ABA signaling and confers drought tolerance to rice[J]. Planta, 2019, 249(5): 1521-1533. DOI:10.1007/s00425-019-03104-7 |
| [31] |
HARTMANN L, PEDROTTI L, WEISTE C, et al. Crosstalk between two bZIP signaling pathways orchestrates salt-induced metabolic reprogramming in Arabidopsis roots[J]. Plant Cell, 2015, 27(8): 22442260. DOI:10.1105/tpc.15.00163 |
| [32] |
SUN X L, LI Y, CAI H, et al. The Arabidopsis AtbZIP1 transcription factor is a positive regulator of plant tolerance to salt, osmotic and drought stresses[J]. J Plant Res, 2012, 125(3): 429-438. DOI:10.1007/s10265-011-0448-4 |
| [33] |
PAN Y L, HU X, LI C Y, et al. SlbZIP38, a tomato bZIP family gene downregulated by abscisic acid, is a negative regulator of drought and salt stress tolerance[J]. Genes, 2017, 8(12): 402. DOI:10.3390/genes8120402 |
| [34] |
HUANG J, WANG M M, JIANG Y, et al. Expression analysis of rice A20/AN1-type zinc finger genes and characterization of ZFP177 that contributes to temperature stress tolerance[J]. Gene, 2008, 420(2): 135-144. DOI:10.1016/j.gene.2008.05.019 |
| [35] |
RAMAMOORTHY R, JIANG S Y, KUMAR N, et al. A comprehensive transcriptional profiling of the WRKY gene family in rice under various abiotic and phytohormone treatments[J]. Plant Cell Physiol, 2008, 49(6): 865-879. DOI:10.1093/pcp/pcn061 |
 2021, Vol. 29
2021, Vol. 29



