2. 仲恺农业工程学院园艺园林学院, 广州 510225
2. College of Horticulture and Landscape Architecture, Zhongkai University of Agriculture and Engineering, Guangzhou 510225, China
无心菜属(Arenaria L.)隶属于石竹科(Caryo- phyllaceae),传统界定的无心菜属超过300种,均为草本,主要分布于北温带至寒带地区[1]。在传统分类中,该属基于花萼、花瓣、花柱及果实等一系列形态特征被分为10个亚属[2]。然而近期分子系统学研究结果揭示无心菜属并非单系,因此对其进行了新的范围划定,传统处理中的众多亚属也被提升至属的地位[3‒5]。
Sadeghian等[5]在2015年开展了无心菜属的系统发育关系研究,认为该属齿瓣亚属[subg. Odon-tostemma (G. Don) Williams]与单花亚属(subg. Solitaria McNeill)各自所形成的分支均与无心菜属模式种占据着不同的系统位置,因此恢复了齿瓣亚属的属级分类等级,即齿瓣无心菜属(Odontostemma Benth ex G. Don),并将单花亚属提升为新属,即独缀草属[Solitaria (McNeill) Sadeghian & Zarre]。然而,Pusalkar等[6]于2015年比Sadeghian等[5]早3个月将单花亚属在属级地位的属名Shivparvatia Pusalkar & D. K. Singh正式发表。遵循植物命名法规中的优先级原则,独缀草属的学名应为Shivpar- vatia[7‒8]。在以形态证据为主的传统分类处理中, 桃色无心菜(Arenaria melandryoides Edgew.)被置于无心菜属齿瓣亚属。考虑到传统的齿瓣亚属已被提升至属级地位,Rabeler等[9]于2016年将传统齿瓣亚属中未进行名称处理的其他63个分类群全部组合至齿瓣无心菜属中,其中包括了桃色无心菜[O. melandryoides (Edgew.) Rabeler & W. L. Wagner]。随后, Chandra等于2017年在研究中国-喜马拉雅(Sino- Himalayan)地区的独缀草属时,基于对馆藏标本的研究,又将桃色无心菜纳入独缀草属的范畴, 并做出了新组合: Shivparvatia melandryoides (Edgew.) Satish Chandra and D. S. Rawat[7]。然而这两次对桃色无心菜在属级水平分类地位归属的处理都是基于形态学证据,缺乏分子系统学证据的支持。因此,对于近年来属级地位归属多次发生变动的桃色无心菜,其合理属级地位的归属以及其准确物种学名的确定,还有待进行深入研究加以澄清,尤其需要结合分子系统学证据深入论证。
桃色无心菜在中国分布于西藏东部、南部以及云南西北部地区,本研究对其开展分子系统学研究,结合形态特征分析结果,对其属级地位归属及物种学名等问题进行了探讨。
1 材料和方法 1.1 类群取样及分子序列数据对曾被置于无心菜属齿瓣亚属的桃色无心菜(凭证标本:LiuJQ-09XZ-ML031, KUN)、漆姑无心菜[Arenaria saginoides Maxim. = Odontostemma sagi-noides (Maxim.) Rabeler & W. L. Wagner] (YangYPQ-2181, KUN)、匙叶无心菜(A. spathulifolia C. Y. Wu ex L. H. Zhou = O. spathulifolium (C. Y. Wu ex L. H. Zhou) Rabeler & W. L. Wagner] (Yangqe 2366, KUN)、云南无心菜[A. yunnanensis Franch. = O. yunnanense (Franch.) Rabeler & W. L. Wagner] (MY-187, KUN)、玉龙山无心菜[A. fridericae Hand.-Mazz. = O. fridericae (Hand.-Mazz.) Sadeghian & Zarre] (LiuJQ 11XZ068, KUN), 以及无心菜属单花亚属的红花无心菜[A. rhodantha Pax et Hoffm. = Shivparvatia rhodantha (Pax & K. Hoffmann) Rabeler] (LiuJQ11XZ235, KUN)的硅胶干燥材料取样,基于改良后的CTAB法[10]提取总DNA; 参考White等[11]提供的ITS序列通用引物对取样类群参考Yao等[12]的方法进行聚合酶链式反应(poly- merase chain reaction, PCR)扩增;采用Geneious v. 7.1.4软件[13]对序列进行检查与拼接。考虑到目前所界定的齿瓣无心菜属与独缀草属均隶属于石竹科繁缕族(Alsineae),因此对该族类群进行扩大取样, 以无心菜属的模式种无心菜(A. serpyllifolia L.)为外类群,所有类群的选择参考Greenberg等[3]提供的石竹科系统发育框架。本研究共取样11属56种(含外类群),其中繁缕族10属55种,外类群1属1种,新获得ITS序列6条(表 1)。
| 表 1 取样物种和ITS序列GenBank编号 Table 1 List of taxa sampled and GenBank accession No. (GAN) of ITS sequences |
在Geneious v. 7.1.4软件[13]中采用默认参数对所有取样个体的ITS序列进行比对,构建ITS序列矩阵;在jModelTest v. 2.1.7软件[14]中按照赤池信息量准则(akaike information criterion, AIC)筛选出序序列矩阵最佳碱基替代模型为SYM+I+Γ; 再采用贝叶斯法(bayesian inference, BI)及最大似然法(maximum likelihood, ML)分别对ITS序列矩阵进行系统发育树构建,两种建树方法及详细步骤参照作者近期对苋科(Amaranthaceae)的系统发育关系研究[15]。
2 结果和分析ITS序列矩阵全长641 bp。贝叶斯法与最大似然法所构建的系统树拓扑结构基本一致,以下对结果的描述及讨论将主要基于最大似然法所构建系统树的拓扑结构展开(图 1)。结果表明,繁缕族类群形成2个主要分支,其中繁缕属(Stellaria L.)、卷耳属(Cerastium L.)、三柱卷耳属[Dichodon (Bartl. ex Rchb.) Rchb.]、硬骨草属(Rabelera M. T. Sharples & E. A. Tripp)、假孩儿参属(Hartmaniella M. L. Zhang & Rabeler)和硬骨繁缕属(Rabelera M. T. Sharples & E. A. Tripp)共同形成1个分支[最大似然法分析自展支持率(bootstrap, BS)=70%, 贝叶斯分析后验概率值(posterior probability, PP)=0.98],而独缀草属、齿瓣无心菜属、孩儿参属(Pseudostellaria Pax)与薄蒴草属(Lepyrodiclis Fenzl)则形成另1个分支,但该分支支持率很低(ML BS=68%, BI PP=0.88) (图 1)。在后一个分支中,薄蒴草属最先分出,独缀草属与由孩儿参属和齿瓣无心菜属形成的分支成姐妹群关系,但这些属间关系支持率均很低。
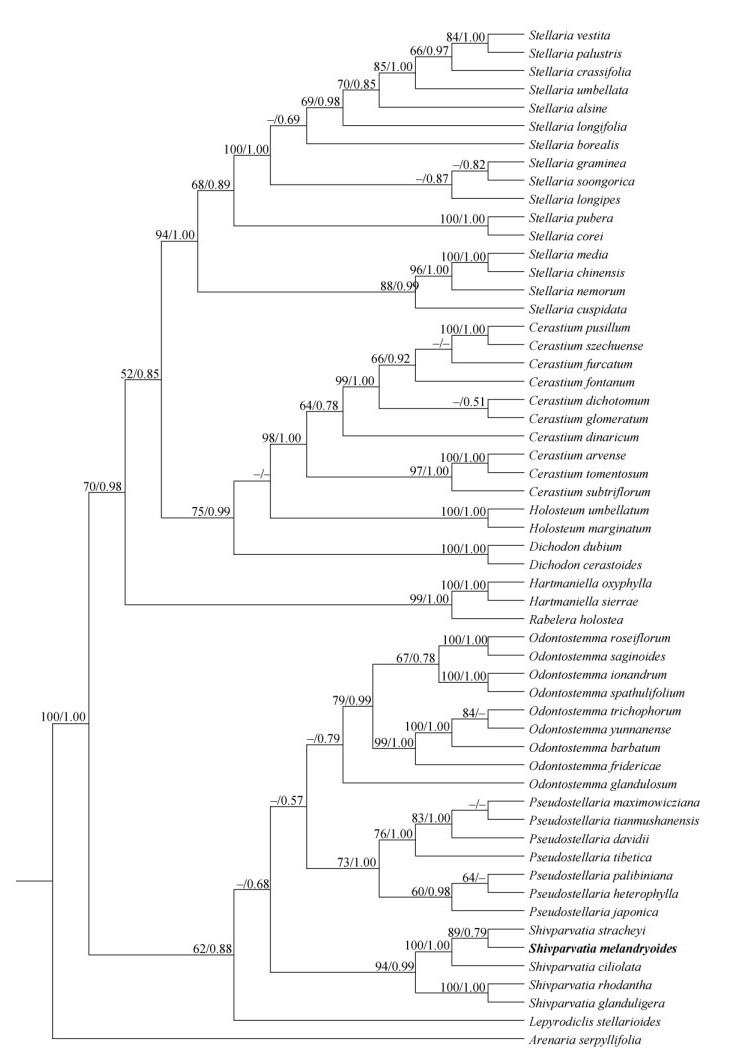
|
图 1 基于核基因ITS序列的繁缕族系统发育关系。分支节点数据代表最大似然树自展支持率/贝叶斯树后验概率; -: 最大似然树自展支持率不足50%或贝叶斯树后验概率不足0.50或该节点未能获得支持。 Fig. 1 Phylogenetic relationship of Alsineae inferred from the nrITS sequence. Data upon each node indicate bootstrap support value/posterior probability. -: Phylogenetic node associated was not supported or the BS value is < 50% in ML analyses or PP < 0.50 in BI. |
本研究所取齿瓣无心菜属物种除了桃色无心菜以外,其他9种形成1个支持率很低的单系(ML BS=34%, BI PP=0.79),其中除了基部最先分出的柔软无心菜(Odontostemma glandulosum Benth. ex G. Don)之外,其他8种形成一个较好的单系分支(ML BS=79%, BI PP=0.99);本研究完成测序的漆姑无心菜、匙叶无心菜、云南无心菜及玉龙山无心菜在该属之中的系统位置均得到解决(图 1)。其中漆姑无心菜与粉花无心菜[O. roseiflorum (Sprague) Sade-ghian & Zarre]、匙叶无心菜与[O. ionandrum (Diels) Sadeghian & Zarre]成支持率很高的姐妹群关系(ML BS=100%, BI PP=1.00),而云南无心菜则与具毛无心菜[O. trichophorum (Franch.) Sadeghian & Zarre]、髯毛无心菜[O. barbatum (Franch.) Sadeghian & Zarre]共同形成一个获得很高支持的分支(ML BS= 100%, BI PP=1.00),该分支再与玉龙山无心菜成支持率很高的姐妹群关系(ML BS=99%, BI PP=1.00)。
桃色无心菜与4种独缀草属物种形成1个获得高度支持的单系分支(ML BS=94%, BI PP=0.99), 并与藏西无心菜[Shivparvatia stracheyi (Edgew.) Pusalkar & D. K. Singh]成姐妹群关系(ML BS=89%, BI PP=0.79);本研究完成测序的红花无心菜则与小腺无心菜[S. glanduligera (Edgew.) Pusalkar & D. K. Singh]形成支持率很高的姐妹群关系(ML BS= 100%, BI PP=1.00)。
3 讨论 3.1 齿瓣无心菜属的分类学问题齿瓣无心菜属基于模式种柔软无心菜(Odon- tostemma glandulosum Benth. ex G. Don; Fig. 2: B)而建立。在Sadeghian等[5]的研究中,分别基于核基因ITS序列和叶绿体基因rps16序列分析的系统发育结果均表明传统无心菜属齿瓣亚属并非单系: 齿瓣亚属中除柔软无心菜(在其系统树中以异名Arenaria debilis Hook. f.出现)以外的其他物种与传统无心菜属单花亚属、孩儿参属共同形成1个分支,该分支再与柔软无心菜成姐妹群关系,但相关系统发育节点所得支持率均很低。尽管齿瓣亚属其他物种未能与柔软无心菜形成单系分支,但Sadeghian等[5]依然将齿瓣亚属所有物种纳入齿瓣无心菜属的范畴,不过考虑到研究取样的情况,仅对6种齿瓣亚属物种给予了新的组合名称。然而,Rabeler等[9]在未对相关物种进行任何形态特征分析或分子系统学研究的情况下,直接将传统齿瓣无心菜亚属其他数十物种全部移入齿瓣无心菜属并进行了新组合。本研究基于ITS序列的系统发育关系揭示出柔软无心菜与齿瓣亚属中除桃色无心菜以外的其他8种形成1个单系,但支持率依然很低。这说明齿瓣无心菜属模式种柔软无心菜的系统位置目前还很不稳定,因此有必要增加序列片段对该种及其近缘分支类群开展深入的分子系统学研究,相关研究结果将决定传统齿瓣亚属中除柔软无心菜以外所有其他物种的属级地位归属,若柔软无心菜与传统齿瓣亚属其他物种确实占据着不同的系统位置,齿瓣亚属其他物种则不能归入齿瓣亚属的范畴,有必要对其进行新的分类处理。
3.2 桃色无心菜等物种的属级分类地位归属Sadeghian等[5]基于分子系统学研究结果, 将传统无心菜属齿瓣亚属和单花亚属分别提升为齿瓣无心菜属和独缀草属,同时也提供了二者的形态特征描述,但基本来自于原亚属水平的特征描述。花序类型、萼片顶端形态、花柱数目等特征可用于齿瓣无心菜属与独缀草属的鉴别性状,其中前者花序类型多样(花单生,或1~3朵,或形成聚伞花序等不同类型)、花萼顶端平截、花瓣顶端齿裂、花柱常2枚(稀3枚),而后者花单生于枝端(Fig. 2: A)、花萼顶端钝至渐尖、花柱3枚。但实际上,齿瓣无心菜属部分物种花萼顶端明显具尖而并非平截,这与独缀草属物种相似;而独缀草属部分物种花柱虽常为3枚,但同时也存在具2枚花柱的个体,其中缘毛无心菜[Shivparvatia ciliolata (Edgew.) Pusalkar & D. K. Singh]便是如此[7]。因此,从花序形态、花萼顶端形态和花柱数目等特征来看,目前所界定的齿瓣无心菜属部分物种与独缀草属存在明显的形态特征重叠,其中花单生于枝端、花萼顶具尖以及花柱2枚(稀3枚)的桃色无心菜(图 3)便是典型代表,这也正是Rawat[7]将该种移入独缀草属的重要原因。本研究的系统发育研究结果进一步支持了Rawat[7]对该种的处理, 即S. melandryoides (Edgew.) Satish Chandra and D. S. Rawat为桃色无心菜的正确学名。

|
图 2 独缀草属模式种小腺无心菜(J. D. Hooker, s.n., GH00353890) (A)和齿瓣无心菜属模式种柔软无心菜(R. Blinkworth s.n., Wallich Cat. 645, K001111464) (B)的模式标本 Fig. 2 Type specimen of Shivparvatia glanduligera (Edgew.) Pusalkar & D. K. Singh (J. D. Hooker, s.n., GH00353890) (A) and Odontostemma glandulosum Benth. ex G. Don (R. Blinkworth s.n., Wallich Cat. 645, K001111464) (B) |

|
图 3 桃色无心菜模式标本(J. D. Hooker s.n., GH00037640) Fig. 3 Type specimen of Shivparvatia melandryoides (Edgew.) Satish Chandra and D. S. Rawat (J. D. Hooker s.n., GH00037640) |
Rabeler等[9]曾将无心菜属齿瓣亚属中数十物种组合至齿瓣无心菜属,漆姑无心菜、匙叶无心菜和云南无心菜这3物种归并至齿瓣无心菜属的分类处理在本研究中首次从分子系统学角度获得了支持,其系统位置也得到了很好解决。形态特征上, 这3物种花序均为聚伞花序,与齿瓣无心菜属模式种一致(Fig. 2: B),不过匙叶无心菜与云南无心菜花萼顶端具尖,又与独缀草属相似,这说明Sadeghian等[5]将花萼顶端是否平截作为齿瓣无心菜属与独缀草属的鉴别特征还有待商榷。另外,曾被置于无心菜属单花亚属的红花无心菜基于形态特征被归入独缀草属之中[8],该分类处理也在此首次从分子系统学角度获得了进一步支持。
另一方面,除了桃色无心菜以外,传统无心菜属齿瓣亚属之中目前已被Rabeler等[9]归入齿瓣无心菜属的紫红无心菜[Odontostemma rockii (Diels) Rabeler & W. L. Wagner]、波密无心菜[O. bomiense (L. H. Zhou) Rabeler & W. L. Wagner]等物种,其花同样单生于枝端、花萼顶端非平截,这些综合特征与独缀草属相似,而与齿瓣无心菜属其他众多物种区别明显,将这些物种置于齿瓣无心菜属是否合理还有待深入研究,尤其需要分子系统学证据的支持。因此,后期有必要对目前所界定的齿瓣无心菜属和独缀草属进行深入的形态特征分析并结合分子系统学研究结果对其进行全面的分类学修订,划定其界定范围,明确其形态特征变异式样,澄清其物种组成。
致谢 承蒙中国西南野生生物种质资源库提供实验材料, 以及哈佛大学植物标本馆(GH)与英国皇家植物园邱园植物标本馆(K)提供标本图片及数据,特此致谢。
| [1] |
WU Z Y, ZHOU L H, WAGNER W L. Arenaria[M]//WU Z Y, RAVEN P H. Flora of China, Vol. 6. Beijing: Science Press & St. Louis: Missouri Botanical Garden Press, 2001: 40-66.
|
| [2] |
MCNEILL J. Taxonomic studies in the Alsinoideae. Ⅰ. Generic and infra-generic groups[J]. Notes R Bot Gard Edinb, 1962, 24(2): 79-155. |
| [3] |
GREENBERG A K, DONOGHUE M J. Molecular systematics and character evolution in Caryophyllaceae[J]. Taxon, 2011, 60(6): 1637-1652. DOI:10.1002/tax.606009 |
| [4] |
HARBAUGH D T, NEPOKROEFF M, RABELER R K, et al. A new lineage-based tribal classification of the family Caryophyllaceae[J]. Int J Plant Sci, 2010, 171(2): 185-198. DOI:10.1086/648993 |
| [5] |
SADEGHIAN S, ZARRE S, RABELER R K, et al. Molecular phylogenetic analysis of Arenaria (Caryophyllaceae: Tribe Arenarieae) and its allies inferred from nuclear DNA internal transcribed spacer and plastid DNA rps16 sequences[J]. Bot J Linn Soc, 2015, 178: 648-668. DOI:10.1111/boj.12293 |
| [6] |
PUSALKAR P K, SINGH D K. Taxonomic rearrangement of Arenaria (Caryophyllaceae) in Indian Western Himalaya[J]. J Jpn Bot, 2015, 90: 77-91. |
| [7] |
CHANDRA S, RAWAT D S. New combinations in Sino-Himalayan genus Shivparvatia (Caryophyllaceae)[J]. Webbia, 2017, 72(2): 177-179. DOI:10.1080/00837792.2017.1332801 |
| [8] |
RABELER R K. New combinations and typification in Shivparvatia (Alsineae, Caryophyllaceae)[J]. Phytotaxa, 2017, 303(3): 293-296. DOI:10.11646/phytotaxa.303.3.11 |
| [9] |
RABELER R K, WAGNER W L. New combinations in Odontostemma (Caryophyllaceae)[J]. PhytoKeys, 2016, 63: 77-97. DOI:10.3897/phytokeys.63.8181 |
| [10] |
DOYLE J J, DOYLE J. A rapid DNA isolation procedure for small quantities of fresh leaf tissue[J]. Phytochem Bull, 1987, 19: 11-15. |
| [11] |
WHITE T J, BRUNS T, LEE S, et al. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics[M]//INNIS M A, GELFAND D H, SNINSKY J J, et al. PCR Protocols: A Guide to Methods and Applications. New York: Academic Press, 1990: 315-322.
|
| [12] |
YAO G, DREW B T, YI T S, et al. Phylogenetic relationships, character evolution and biogeographic diversification of Pogostemon s.l. (Lami-aceae)[J]. Mol Phylogenet Evol, 2016, 98: 184-200. DOI:10.1016/j.ympev.2016.01.020 |
| [13] |
KEARSE M, MOIR R, WILSON A, et al. Geneious basic: An inte-grated and extendable desktop software platform for the organization and analysis of sequence data[J]. Bioinformatics, 2012, 28: 1647-1649. DOI:10.1093/bioinformatics/bts199 |
| [14] |
POSADA D. jModelTest: phylogenetic model averaging[J]. Mol Biol Evol, 2008, 25: 1253-1256. DOI:10.1093/molbev/msn083 |
| [15] |
HUANG J X, CHEN W N, LI Y L, et al. Phylogenetic relationship of Amaranthaceae s.l. based on multiple plastid DNA fragments[J]. Chin Bull Bot, 2020, 55(4): 457-467. DOI:10.11983/CBB19228 |
 2021, Vol. 29
2021, Vol. 29



