2. 温州科技职业学院农业与生物技术学院, 浙江 温州 325006
2. College of Agriculture and Biotechnology, Wenzhou Vocational College of Science and Technology, Wenzhou 325006, Zhejiang, China
GRAS基因是植物特有的一类转录因子,由GAI、RGA和SCR等3个基因家族成员的特征字母命名而来[1-2]。GRAS家族通常包含SAW、亮氨酸重复序列I (LFRI)、NLS、VHIID和亮氨酸重复序列II (LHRII)、PFYRE和LXX-LL等几个典型的结构域,可分为SHR、SCL、SCR、RGA、RGL (RGA- like)、GAI和PAT1等8个分支[3]。目前,GRAS转录因子已在拟南芥(Arabidopsis thaliana)、水稻(Oryza sativa)、白菜(Brassica pekinensis)、蓖麻(Ricinus communis)、玉米(Zea mays)和番茄(Lycopersicon esculentum)等植物中克隆出来[4-9]。
GRAS家族成员数量众多,在植物逆境胁迫应答中发挥着重要作用。Torres-Galea等的研究表明干旱、盐、低温等非生物胁迫可促进拟南芥AtSCL13的表达[1]。李雪燕等报道柽柳(Tamarix hispida)的GRAS转录因子启动子含有多个逆境相关元件,推测其可参与植物的逆境胁迫响应[10]。过表达水稻中的OsGRAS23基因和甘蓝型油菜中的GRAS基因,可提高植株对干旱胁迫的耐受性[11]。
青花菜(Brassica olreacea var. italica)为十字花科(Brassicaceae)芸薹属一二年生草本植物。水分是青花菜生长发育过程中重要的调控因子,水分过多会严重影响生长,甚至造成整株死亡。因此对青花菜渍水胁迫的响应机理进行研究,对于青花菜生产具有重要的现实意义。
本试验从青花菜中克隆得到1个BoSCL3基因, 对其编码的BoSCL3蛋白进行生物信息学分析,并采用荧光定量PCR技术检测BoSCL3基因在渍水胁迫下的表达特性,为揭示青花菜BoSCL3基因响应渍水胁迫的分子机制提供理论依据。
1 材料和方法 1.1 材料供试材料为青花菜(Brassica olreacea var. ita- lica ‘WN12-95’)。种子催芽后,播种到营养钵中, 放置于光照培养箱内进行培养。昼夜温度为25℃/ 18℃, 光照周期为16 h/8 h。待植株长至五叶期时开始渍水胁迫处理,每处理3次重复。分别在处理后0、2和6 d采集叶片,液氮冷冻后置于超低温冰箱保存。
1.2 总RNA提取及cDNA合成参照TaKaRa Mini BEST Plant RNA Extraction Kit试剂盒(TaKaRa公司)的操作说明提取青花菜叶片的总RNA。总RNA质量检测合格后,使用Prime Script 1st Strand cDNA Synthesis Kit反转录试剂盒(大连TaKaRa公司)反转录合成cDNA第一条链。
1.3 基因的克隆根据转录组unigene库中青花菜BoSCL3基因设计全长克隆引物,F:5'-TCGCTTTGATATGGTGGTT- ATGT-3';R:5'-TCATTCACTTCTTGCATCTCCA-3'。PCR反应体系包括1 μL cDNA,上、下游引物各1 μL、10 μL Taq mixture,7 μL ddH20。扩增程序为:94℃预变性5 min;94℃变性30 s,60℃复性50 s,72℃延伸1 min,36个循环;72℃延伸10 min。PCR产物回收后进行TA克隆,选取阳性克隆测序。
1.4 生物信息学分析采用InterPro软件(http://www.ebi.ac.uk/interpro/)进行结构域分析;采用在线软件ProtParamtool (http://web.expasy.org/protparam/)预测氨基酸多肽链的氨基酸组成、等电量、相对分子量、分子式等[12-14]; 使用DNAMAN软件预测多肽链的亲/疏水性; 采用GOR4工具(https://npsa-prabi.ibcp.fr/cginbin/npsa_automat.pl?page=npsa_gor4.html)对编码蛋白的二级结构进行预测。蛋白序列同源性比对和进化树的构建采用Mega 6.0软件[15]。
1.5 BoSCL3基因的表达分析根据获得青花菜BoSCL3基因的cDNA序列,设计定量引物,F: 5'-CTTAACGCAACTCAGACAA-3'和R: 5'-CCATCTTCTCACCTTCCA-3'。以actin基因(引物分别为5'-GACAACTTACAACTCCATCAT-3'和5'-CTCATACGGTCAGCGATA-3')为内参基因。使用SYBR Premix Ex Taq (大连TaKaRa公司)试剂盒在ABI Prism 7500定量扩增仪上进行实时荧光定量分析。反应体系为:cDNA 1 μL,上、下游引物各1 μL, SYBR 10 μL,ddH2O 12 μL。反应程序为94℃预变性2 min;94℃变性20 s,60℃退火30 s,72℃延伸40 s,40个循环,最后72℃延伸10 min。每个反应设3次重复,采用2-ΔΔCT法计算基因的相对表达量。
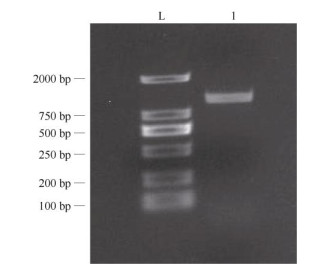
2 结果和分析 2.1 BoSCL3基因的克隆及序列特征分析采用特异引物,经PCR扩增后,从青花菜叶片中克隆了BoSCL3基因(图 1)。序列分析表明该基因cDNA全长1 355 bp,推导编码446个氨基酸(图 2)。InterPro分析表明,该基因具有保守的GRAS结构域,属于GRAS基因家族。
|
图 1 青花菜BoSCL3基因的PCR扩增。L: DL 2000 Marker; 1: BoSCL3。 Fig. 1 PCR amplification of BoSCL3 gene in Brassica olreacea var. italica. L: DL 2000 Marker; 1: BoSCL3. |

|
图 2 BoSCL3基因的全长序列和推测的氨基酸序列。下划线为引物序列。 Fig. 2 Full-length sequence of BoSCL3 gene and deduced amino acid sequence. Primer sequence is marked with underline. |
青花菜BoSCL3蛋白的分子量为49.96 kDa,理论等电点为6.14;带负电残基总数为51,带正电残基总数为45;疏水性最大值为3.04,亲水性最大值为2.38,同时疏水性氨基酸多于亲水性氨基酸,说明该蛋白为疏水性蛋白。
通过GOR4工具对蛋白氨基酸序列的二级结构进行分析, 结果表明,青花菜BoSCL3蛋白的二级结构包括无规则卷曲(Cc)(44.62%)、延伸链(Ee) (12.33%)和α-螺旋(Hh)(43.05%)。
2.3 系统进化树利用BLAST对BoSCL3蛋白的氨基酸序列同源性进行分析,结果表明,BoSCL3基因编码的氨基酸序列与甘蓝(Brassica oleracea)、油菜(B. napus)、芜菁(B. rapa)、白菜、萝卜(Raphanus sativus)、拟南芥中的SCL3氨基酸序列的同源性分别是100%、98%、98%、98%、91%和90%, 与烟草、长叶马先蒿、可可、毛果杨的相似性也都在60%以上。
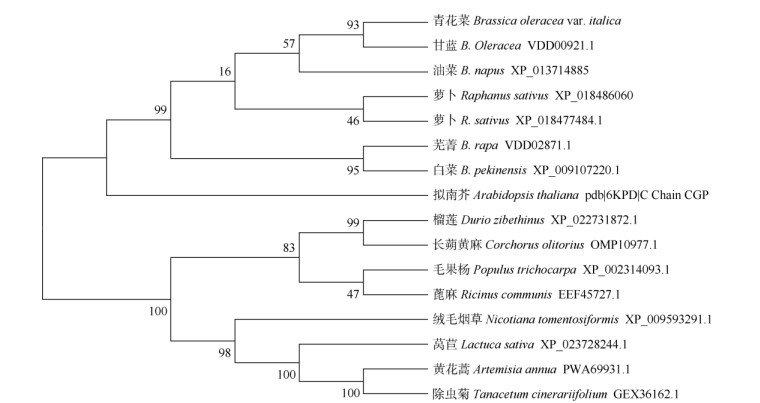
将青花菜BoSCL3蛋白的氨基酸序列与甘蓝、油菜、芜菁、白菜、萝卜、拟南芥(Arabidopsis lyrata subsp. lyrata)、榴莲(Durio zibethinus)、长蒴黄麻(Corchorus olitorius)、黄花蒿(Artemisia annua)、除虫菊(Tanacetum cinerariifolium)、莴苣(Lactuca sativa)、绒毛烟草(Nicotiana tomentosiformis)、蓖麻(Ricinus communis)、毛果杨(Populus trichocarpa)等14种植物的SCL3氨基酸序列进行多重比对并构建进化树(图 3),可见,青花菜的BoSCL3与甘蓝、油菜的SCL3亲缘关系较近。同属于十字花科的甘蓝、油菜、芜菁、萝卜、白菜和拟南芥聚在同一分支上,表明同科植物的SCL3蛋白具有较高的同源性。
|
图 3 基于SCL3蛋白氨基酸序列构建的系统进化树 Fig. 3 Phylogenetic trees based on amino acid sequence of SCL3 protein |
利用实时荧光定量PCR技术检测青花菜BoSCL3渍水处理不同时间的表达特征。结果表明(图 4),青花菜BoSCL3基因在渍水胁迫处理2 d时的表达受到抑制,相对表达量仅为0.11。其后随着渍水胁迫时间的延长,表达量呈现上升的趋势,处理6 d的相对表达量为1.18。

|
图 4 渍水胁迫下BoSCL3基因的表达 Fig. 4 Expression of BoSCL3 gene under waterlogging stress |
本研究从青花菜中成功克隆到BoSCL3基因, 对其编码蛋白的亲/疏水性、结构域、二级结构和同源性进行了预测分析并构建了系统进化树。对渍水胁迫下青花菜BoSCL3基因的表达进行了分析,为进一步揭示青花菜BoSCL3基因响应渍水胁迫的应答机制提供了一定的理论依据。
转录因子对于增强植物对逆境的抵抗力和适应能力具有重要作用[16-17]。本试验克隆得到1个青花菜BoSCL3基因,其编码的蛋白具有GRAS家族的保守结构域,为疏水性蛋白。石瑞等[18]报道的佛手(Citrus medica var. sarcodactylis)的GRAS和陈裕坤等[19]报道的龙眼(Dimocarpus longan)胚性愈伤组织DlGRAS4与DlGRAS54的结构、性质相似。将BoSCL3蛋白的氨基酸序列与荠蓝、油菜、萝卜、白菜、拟南芥等植物中GRAS家族的SCL3的氨基酸序列进行比对,表明约有80%以上的同源性,推测SCL3在功能上存在一定的保守性。
逆境胁迫是限制作物生长的重要因子,严重时可导致植株形态发生变化。GRAS基因可在植物受到逆境胁迫时,产生应答机制来提高植物的抗逆性,直接表现为表达量的变化。本研究中,渍水胁迫处理可对BoSCL3基因的表达量产生影响,说明BoSCL3基因可能参与了青花菜的渍水胁迫响应。前人的研究表明,水稻OsGRAS1的表达量在受到盐、干旱和外源ABA的胁迫时表达上调[20]。郭华军等的研究也表明拟南芥13个GRAS基因在干旱和渗透胁迫处理下的表达均比对照显著上升[21]。本试验中青花菜BoSCL3基因的表达量随着渍水胁迫时间的延长,呈先急剧下降后快速上升的变化趋势,表明BoSCL3基因可能在青花菜渍水胁迫调控过程中具有重要作用。
| [1] |
Torres-Galea P, Hirtreiter B, Bolle C. Two GRAS proteins, SCARE-CROW-LIKE21 and PHYTOCHROME A SIGNAL TRANS-DUCTION1, function cooperatively in phytochrome a signal trans-duction[J]. Plant Physiol, 2013, 161(1): 291-304. DOI:10.1104/pp.112.206607 |
| [2] |
ZHOU L J, YANG Z M, ZHANG F C, et al. Expression analysis and cloning of GRAS transcription factor gene from Halostachys caspica[J]. Acta Bot Boreali-Occid Sin, 2013, 33(6): 1091-1097. 周莲洁, 杨中敏, 张富春, 等. 新疆盐穗木GRAS转录因子基因克隆及表达分析[J]. 西北植物学报, 2013, 33(6): 1091-1097. DOI:10.3969/j.issn.1000-4025.2013.06.004 |
| [3] |
PYSH L D, WYSOCKA-DILLER J W, CAMILLERI C, et al. The GRAS gene family in Arabidopsis: Sequence characterization and basic expression analysis of the SCARECROW-LIKE genes[J]. Plant J, 1999, 18(1): 111-119. DOI:10.1046/j.1365-313X.1999.00431.x |
| [4] |
HOU M Y. Cloning, transformation and functional analysis of GRAS transcription factor family genes in rice[D]. Jilin: Jilin University, 2013: 9. 侯梦筠. 水稻GRAS转录因子家族基因克隆、遗传转化与功能分析[D]. 吉林: 吉林大学, 2013: 9. |
| [5] |
TIAN C G, WAN P, SUN S H, et al. Genome-wide analysis of the GRAS gene family in rice and Arabidopsis[J]. Plant Mol Biol, 2004, 54(4): 519-532. DOI:10.1023/b:plan.0000038256.89809.57 |
| [6] |
SONG X M, LIU T K, DUAN W K, et al. Genome-wide analysis of the GRAS gene family in Chinese cabbage (Brassica rapa ssp. pekinensis)[J]. Genomics, 2014, 103(1): 135-146. DOI:10.1016/j.ygeno.2013.12.004 |
| [7] |
HAN W Y, LI G R, FENG L, et al. Genome-wide analysis of GRAS transcription factors in Ricinus communis and response to abiotic stresses[J]. J Plant Genet Resour, 2020, 21(1): 252-259. 韩雯毓, 李国瑞, 风兰, 等. 蓖麻GRAS转录因子家族的全基因组分析及逆境胁迫响应[J]. 植物遗传资源学报, 2020, 21(1): 252-259. DOI:10.13430/j.cnki.jpgr.20190521002 |
| [8] |
YIN L F, WANG Z Y, WU Z Y, et al. Cloning and functional analysis of ZmGRAS31 gene in maize[J]. Acta Agron Sin, 2019, 45(7): 1029-1037. 殷龙飞, 王朝阳, 吴忠义, 等. 玉米ZmGRAS31基因的克隆及功能研究[J]. 作物学报, 2019, 45(7): 1029-1037. DOI:10.3724/SP.J.1006.2019.83070 |
| [9] |
REN L J. Cloning and functional analysis of tomato GRAS family gene SlFSR[D]. Chongqing: Chongqing University, 2014: 12. 任丽军. 番茄GRAS转录因子家族基因SlFSR的克隆及其功能研究[D]. 重庆: 重庆大学, 2014: 12. |
| [10] |
LI X Y, JIN J J, ZHAO Y L, et al. Cloning and expression analysis of promoter of GRAS transcription factor from Tamarix hispida[J]. Chin Agric Sci Bull, 2016, 32(2): 28-32. 李雪燕, 金胶胶, 赵玉琳, 等. 柽柳GRAS转录因子基因启动子克隆和表达分析[J]. 中国农学通报, 2016, 32(2): 28-32. |
| [11] |
XU K, CHEN S J, LI T F, et al. OsGRAS23, a rice GRAS transcription factor gene, is involved in drought stress response through regulating expression of stress-responsive genes[J]. BMC Plant Biol, 2015, 15(1): 141. DOI:10.1186/s12870-015-0532-3 |
| [12] |
PEI X L, JING Z G, TANG Z, et al. Cloning and expression analysis of a pollen development gene MF21 in broccoli[J]. Biotechnol Bull, 2015, 31(3): 102-107. 裴徐梨, 荆赞革, 唐征, 等. 青花菜花粉发育基因MF21的克隆及表达特征分析[J]. 生物技术通报, 2015, 31(3): 102-107. DOI:10.13560/j.cnki.biotech.bull.1985.2015.04.014 |
| [13] |
LI X N, XIAO H Z, WAN S L, et al. Cloning and expression analysis of HbP450 gene in Hevea brasiliensis Muell. Arg.[J]. Chin J Trop Crops, 2017, 38(11): 2100-2105. 李晓娜, 肖厚贞, 万三连, 等. 巴西橡胶树HbCYP450基因克隆与表达分析[J]. 热带作物学报, 2017, 38(11): 2100-2105. DOI:10.3969/j.issn.1000-2561.2017.11.017 |
| [14] |
CAO X S, WANG J, ZHANG Y Y, et al. Cloning and bioinformatic analysis of MK gene from Cinnamomum camphora[J]. Chin J Trop Crops, 2017, 38(12): 2302-2309. 曹先爽, 王进, 张瑶瑶, 等. 香樟MK基因的克隆与生物信息学分析[J]. 热带作物学报, 2017, 38(12): 2302-2309. DOI:10.3969/j.issn.1000-2561.2017.12.017 |
| [15] |
GUO Y Y. Gene cloning and functional analysis of the GRAS trans-cription factor family from Maize (Zea mays L.)[D]. Taian: Shandong Agricultural University, 2015: 21. 郭玉玉. 玉米GRAS转录因子家族基因克隆与功能验证[D]. 泰安: 山东农业大学, 2015: 21. |
| [16] |
LI A Y, LIU H, LI X Y, et al. Cloning and subcellular localization analysis of two GRAS genes from Poncirus trifoliata[J]. Genom Appl Biol, 2012, 31(3): 240-248. 李阿英, 刘洪, 李晓颖, 等. 枳两个GRAS基因cDNA全长的克隆及其亚细胞定位分析[J]. 基因组学与应用生物学, 2012, 31(3): 240-248. DOI:10.3969/gab.031.000240 |
| [17] |
NIU Y L. Bioinformatic analysis and identification of some resistance-associated genes of GRAS gene family in tomato[D]. Harbin: North-east Agricultural University, 2017: 1. 牛义岭. 番茄GRAS基因家族生物信息学分析及部分抗性相关基因鉴定分析[D]. 哈尔滨: 东北农业大学, 2017: 1. |
| [18] |
SHI R, CAO Y B, CHEN W R, et al. On cDNA cloning and expression analysis of GRAS gene in fingered citron[J]. J Zhejiang Norm Univ (Nat Sci), 2011, 34(4): 446-451. 石瑞, 曹诣斌, 陈文荣, 等. 佛手GRAS基因的克隆及表达分析[J]. 浙江师范大学学报(自然科学版), 2011, 34(4): 446-451. DOI:10.3969/j.issn.1001-5051.2011.04.016 |
| [19] |
CHEN Y K, LIN Y L, TIAN Q L, et al. Cloning and expression analysis of DlGRAS4 and DlGRAS54 from embryogenic callus of Dimocarpus longan Lour.[J]. Acta Bot Boreali-Occid Sin, 2014, 34(2): 215-224. 陈裕坤, 林玉玲, 田奇琳, 等. 龙眼胚性愈伤组织DlGRAS4与DlGRAS54基因的克隆及表达分析[J]. 西北植物学报, 2014, 34(2): 215-224. DOI:10.7606/j.issn.1000-4025.2014.02.0215 |
| [20] |
DING X E, LIU H Y, LUO L J. Cloning and diversity analysis of the OsGRAS1 promoter in rice[J]. Acta Agric Shanghai, 2010, 26(4): 8-14. 丁雪峰, 刘鸿艳, 罗利军. 水稻OsGRAS1启动子的克隆及多样性分析[J]. 上海农业学报, 2010, 26(4): 8-14. DOI:10.3969/j.issn.1000-3924.2010.04.003 |
| [21] |
GUO H J, JIAO Y N, DI C, et al. Discovery of Arabidopsis GRAS family genes in response to osmotic and drought stresses[J]. Chin Bull Bot, 2009, 44(3): 290-299. 郭华军, 焦远年, 邸超, 等. 拟南芥转录因子GRAS家族基因群响应渗透和干旱胁迫的初步探索[J]. 植物学报, 2009, 44(3): 290-299. DOI:10.3969/j.issn.1674-3466.2009.03.005 |
 2021, Vol. 29
2021, Vol. 29


