植物的形态、生理和生化反应由一系列基因控制,这些基因的表达会受到多种因素的诱导,包括病菌、干旱、高盐、寒冷等。根据基因的表达模式和对环境胁迫的反应,可以分为不同类别,即冷诱导响应基因COR、低温响应基因LTI和脱水诱导早期响应基因ERD等[1]。拟南芥(Arabidopsis thaliana)受脱水胁迫1 h内可诱导ERD基因表达,已鉴定的16个ERD基因cDNA属于不同的基因家族,作用于不同的代谢途径,以增强拟南芥的抗旱性[2],这些基因除了具备快速应答干旱胁迫外,还具备应答冷、高盐、衰老、ABA (脱落酸)等多种逆境胁迫信号,表现出多样化生化功能并存在于不同的亚细胞[3-5],ERD1编码叶绿体ATP依赖蛋白酶,防御叶绿体损伤;ERD13是植物特异性硫代谷胱甘肽转移酶,能与BAK1相互作用;ERD16是一种泛素化延伸蛋白[6-8]。拟南芥中ERD15基因受各种生物因子和非生物因子的诱导表达,是ABA反应和依赖水杨酸(SA)防御途径的重要调节器[9-10]。
基因组学研究证实,ERD15属于一个高度保守的植物特有基因家族,ERD15蛋白具有典型的与PABP [poly(A) binding protein]作用的基序PAM2 (PABP-interacting motif 2),其他高度保守基序PAE1 (PAM2 associated element 1)和QPR (amino acid sequence isoleucine-glutamine/histidine-glutamine- proline-arginine)的功能还有待研究[11]。含有PAM2基序的蛋白称为PACs (PAM2-containing proteins)家族蛋白,该家族蛋白参与mRNA代谢,同时也参与调控生物周期节律以及泛素化降解等[12-13]。这提示ERD15可能在基因表达的转录后调控中发挥作用。目前对于ERD15的研究主要是与胁迫响应有关,大豆(Glycine max)在渗透胁迫下,ERD15蛋白结合并激活NRP-B启动子表达,调控引发细胞程序性死亡的相关基因[14]。低温下葡萄(Vitis vinifera)的ERD15可以保护细胞膜,提高光合效率并促进可溶性物质的积累和抑制脂质过氧化来增强植物抵抗外部应激的能力[15]。Saeed等[16]报道桑树(Morus alba)的ERD15蛋白能在酵母中驱动报告基因表达,作为转录调控因子发挥功能,其中第70~100位氨基酸是转录激活的关键区域,可能含有ERD15的DNA结合结构域。对拟南芥中的研究表明,ERD15可以瞬时被各种胁迫以及和胁迫相关的植物激素所诱导,如脱水、低温、外界损伤、ABA、SA、高光和植物病原菌等[7, 9-10, 17]。在非生物胁迫响应中, ERD15是ABA信号的负调控因子,过量表达ERD15会降低植物对ABA的敏感性,从而降低植株对干旱和寒冷的耐受性;相反,通过RNAi沉默导致ERD15表达缺失使得植株对ABA超敏感, 从而提高耐旱性和抗冷性[9, 18]。胡萝卜(Daucus carota) ERD15基因过表达诱导水杨酸调控基因的应答,增加了对软腐欧文氏菌的抵抗力[9]。拟南芥中ERD15负调控ABA信号途径,阻止植物在生物胁迫时快速应答,从而加剧对植物的损害; 但过表达ERD15可以提高葡萄和烟草(Nicotiana tabacum)的存活率, 减少脂质过氧化,提高渗透压从而增强对干旱和冷胁迫的耐受性[15, 19]。因此,不同植物中ERD15可能具有不同的功能[16, 20-21]。
ERD15响应各种逆境胁迫,是多种信号调节的枢纽,为深入探究ERD15基因的功能,本研究以拟南芥T-DNA插入纯合突变体erd15为材料,进行表型观察分析,揭示了ERD15基因参与调控拟南芥生殖生长过程,为进一步研究该基因的功能和分子调控机制奠定基础。
1 材料和方法 1.1 材料和培养条件拟南芥(Arabidopsis thaliana)突变体erd15 (SALK_ 202985)购自拟南芥生物资源中心ABRC (Arabidopsis Biological Resource Center),经筛选鉴定后的纯合突变体用于本试验。野生型(Col-0)由华南师范大学生态与环境科学重点实验室保存。
拟南芥种子在黑暗中4℃春化3 d,用30% (V/V)次氯酸钠消毒10 min,无菌水反复洗涤8~10次后播种在固体1/2MS培养基上。培养温度22℃,相对湿度60%~70%,16 h/8 h的光暗周期。幼苗长出2~4片真叶后,移栽到培养土中(营养土:蛭石= 2:1),覆盖保鲜膜保湿3 d后揭膜,相同温度和光照条件继续培养。
1.2 纯合突变体DNA水平的鉴定幼苗生长3周,剪取叶片采用CTAB法[22]提取基因组总DNA,以总DNA为模板进行PCR扩增。PCR所用引物序列从http://signal.salk.edu/上获取。采用基因特异引物LP、RP和T-DNA通用引物LB1.3 (表 1)进行PCR扩增,筛选出erd15纯合突变体,并确定T-DNA的插入位点。反应体系共20 μL:模板1 μL, 引物各1 μL,2×Taq PCR Master Mix 10 μL (上海生物工程有限公司),加ddH2O至20 μL,充分混匀后进行PCR扩增。PCR反应程序为:94℃变性4 min,94℃变性30 s,55℃退火30 s,72℃延伸1 min,共30个循环。PCR产物经1.2%琼脂糖凝胶电泳检测后于凝胶成像系统成像。
| 表 1 PCR引物序列 Table 1 Primer sequences for PCR |
取1周龄幼苗约100 mg为材料,采用Trizol法提取总RNA后用反转录试剂盒(Prime ScriptTM reagent Kit with gDNA Eraser)进行反转录,得到的cDNA进行RT-PCR,分析基因表达水平。以Actin2 (AT3G18780)为内参基因,对目的基因的表达水平进行标准化。反应体系及反应程序同DNA水平筛选实验,PCR扩增产物经1.0%琼脂糖凝胶电泳检测后于凝胶成像系统成像。
1.4 植株的表型观测随机选取20株拟南芥野生型Col-0和突变体erd15植株,观测并记录莲座叶数目、花序形态、果荚长度等性状。使用体式荧光显微镜(徕卡, M205FA)和佳能相机(Canon EOS D70)拍摄记录表型,使用Photoshop和Image J软件对采集的图像进行处理和测量分析。统计4周龄生长状况良好植株的莲座叶数目;以种植日到第1朵花露白的天数为开花时间[23];选择发育到第14阶段左右的花,用解剖针小心去除萼片和花瓣,观察拍摄雌蕊表型; 在植株花序下约10 cm处测量主茎直径;花发育至第17阶段时,取不同基因型角果各30个,测量果荚长度,并徒手横切,观察果荚内部的种子腔[24-25],按照Alvarez-Buylla等的方法划分花发育阶段[25]。收集成熟但未开裂的长角果,完全浸入脱色液中(乙醇:乙酸体积比=3:1),脱色24 h使长角果透明, 便于观察种子表型和败育情况。植株高度以主茎的最终长度为准,生长50 d时进行测量。在同一固体1/2MS培养基上,划分区域均匀点播野生型和突变体种子,每区域播种50粒,共10个重复,6 d后统计种子萌发率。
1.5 叶绿素荧光参数的测定选择生长4周左右已成熟的莲座叶,用CF Imager叶绿素荧光快速成像系统(Chlorophyll Fluo- rescence Imager System, Technologica公司, 英国)进行叶绿素荧光参数测定。Fv/Fm代表PSII潜在的最大光能转化效率;Fv'/Fm'反映光合反应中心PSII实际捕获能量的传递效率;Fq'/Fm'为PSII实际的光化学量子效率,是反映植物光合能力的重要指标之一;NPQ为非光化学淬灭系数,反映植物热耗散能力[27-28]。测定前,将拟南芥幼苗暗处理30 min后测定初始荧光(Fo),在6 164 μmol/(m2·s)光照强度下测定Fv/Fm,然后打开光照强度为100 μmol/(m2·s)的测量光,光适应5 min后测定Fv'/Fm'、Fq'/Fm'和NPQ等参数。
1.6 数据处理和分析方法使用软件SPSS 23对数据进行显著性分析,以P < 0.05表示差异显著;P < 0.01表示差异极显著。
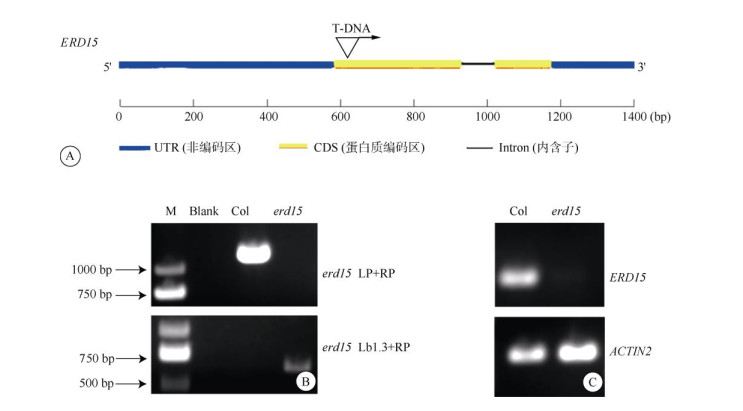
2 结果和分析 2.1 纯合突变体的鉴定从图 1: A可见,ERD15 (AT2G41430)由2个外显子和1个内含子组成,erd15 (SALK_202985)突变体的T-DNA插入位点位于第一个外显子上, 约627 bp处。
|
图 1 纯合突变体的鉴定。A: ERD15基因结构和T-DNA插入位点; B: erd15突变体基因水平检测; C: erd15突变体转录水平的检测。 Fig. 1 Identification of homozygous mutant. A: ERD15 gene structure and T-DNA insertion site; B: Detection of erd15 mutant at gene level; C: Detection of erd15 gene at expression level. |
用ERD15基因的特异性引物LP与RP进行PCR扩增,野生型中扩增出1条带,条带位置与目的基因(1 385 bp)大小一致,而与此对应的erd15突变体中未扩增出条带,而用T-DNA特异性引物LB1.3和RP进行扩增,erd15突变体中扩增出1条特异条带,条带大小为500~750 bp,与引物的预期扩增结果一致,而野生型中未能扩增出此条带(图 1: B)。证明本试验所使用ERD15基因的T-DNA插入erd15突变体在DNA水平上是纯合突变。为确定erd15突变体植株在转录水平是否仍存在ERD15基因的表达,从基因转录水平对erd15进行了转录水平鉴定,半定量PCR结果表明,以Actin2为内参基因,野生型拟南芥扩增出ERD15基因的CDS片段,而纯合erd15突变体无表达产物(图 1: C),这说明由于T-DNA的插入导致ERD15的转录表达缺失, erd15突变体为功能缺失突变体。
2.2 erd15突变体的表型分析将野生型拟南芥和erd15纯合突变体共同种植进行表型比较分析。从表 2可见,erd15突变体的株高和野生型接近,无显著差异(图 2: A)。植株营养生长后期,突变体的莲座叶数目较野生型多37.97% (图 2: B)。生长28 d的拟南芥野生型和erd15突变体叶片的叶绿素荧光参数Fv/Fm、Fv'/Fm'、Fq'/Fm'和NPQ等均无显著差异(表 3),这一定程度表明ERD15基因突变没有改变erd15突变体的光合作用能力。erd15突变体开花时间较野生型植株提前3~4 d (图 2: C),说明ERD15基因的缺失可能加速了植物由幼龄期向成熟期转变。
| 表 2 erd15突变体和野生型Col-0的表型 Table 2 Phenotype of erd15 mutant and wild type Col-0 |

|
图 2 野生型Col-0与erd15突变体的表型。A:种子萌发60 d的植株; B:莲座叶; C:长日照下erd15突变体开花。 Fig. 2 Phenotype of wild-type Col-0 and erd15 mutant. A: Plant height at 60 days after germination; B: Rosette leaves; C: Early flowering of erd15 mutant under long-day. |
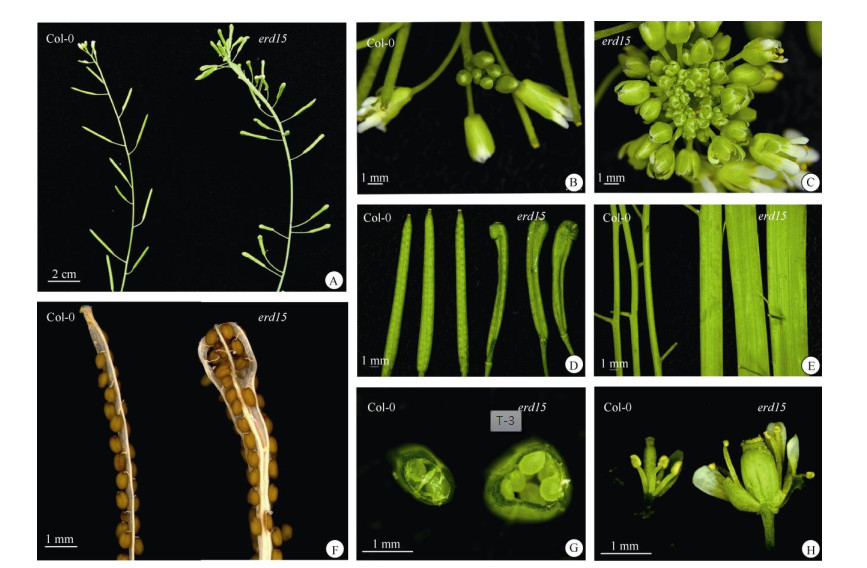
在拟南芥生殖生长期,ERD15基因的缺失导致突变体植株花序表型异常(图 3: A~C);角果表面出现皱褶,顶端膨大成楔形(图 3: D),长度比野生型缩短37.67% (表 2),达显著差异。主茎由细长的圆柱体变成扁平状,比野生型的直径增加75.97% (图 3: A, E,表 3)。野生型Col-0自然开裂的长角果由2个心皮和1个隔膜组成,形成假两室,种子通过株柄呈两列附着在隔膜上;erd15突变体自然开裂的长角果由多心皮发育而成,内部假隔膜部分不融合,有多排种子(图 3: F)。从角果的徒手横切切片可见,野生型的角果内有2个种子腔,erd15突变体长角果顶端膨大区域内无明显种子腔(图 3: G), 并且比野生型的雌蕊异常膨大(图 3: H),这进一步说明erd15的雌蕊可能由多心皮融合而成。野生型长角果的平均种子数为46.94粒,erd15突变体为65.25粒(图 4: A, 表 2),增加了39.01%,可见erd15突变体长角果的平均长度较野生型短,但平均结籽数却显著增加。
|
图 3 野生型Col-0和erd15突变体生殖生长期的表型。A:主茎上的花和果荚; B, C:花序; D:角果; E:主茎; F:去除果皮的角果; G:角果横切; H:雌蕊。 Fig. 3 Phenotype of wild-type Col-0 and erd15 mutant at reproductive stage. A: Flower and silique on main stems; B, C: Inflorescences; D: Silique; E: Main stem; F: Silique removed fruit coat; G: Transection of silique; H: Pistil. |
| 表 3 erd15突变体和野生型Col-0的叶绿素荧光参数 Table 3 Chlorophyll fluorescent parameters of erd15 mutant and wild-type Col-0 |
拟南芥野生型和突变体种子均呈饱满椭圆状、无皱褶、棕色,没有明显差异(图 4: C)。野生型种子的萌发率为96.00%,erd15突变体为95.80% (图 4: B, 表 2),无显著差异。这表明ERD15基因的缺失虽然干扰了拟南芥的生殖生长,但并没有导致育性下降。

|
图 4 野生型Col-0和erd15突变体的种子表型。A:透明角果; B, C:种子。 Fig. 4 Seed phenotype of erd15 mutant and wild-type Col-0. A: Decolorized siliques; B, C: Seeds. |
随着基因组计划的完成,反向遗传学已经是研究基因功能的有效方法之一。反向遗传学是指从已知的基因序列出发,通过对靶基因进行必要的加工和修饰,如定点突变、基因插入、基因置换等,从而获得该基因突变产生的突变体,根据突变体表型的变化研究该基因的功能。目前拟南芥用农杆菌侵染的方法创造出大量T-DNA插入突变体(http://signal.salk.edu/)。本文以拟南芥野生型Col-0和T-DNA插入突变体erd15为材料,进行了表型的初步分析,结果表明,erd15突变体植株在表型上与野生型发生了明显变化且在后代可稳定遗传。本研究获得的erd15突变体,erd15基因表达缺失,表明该突变体可以用于对ERD15基因功能的研究。
通过观察,erd15突变体的莲座叶数目增多, 出现早花现象,暗示着该基因的缺失表达使得植物过早地从营养生长转向了生殖生长[23]。Bravo等通过半定量PCR检测不同组织中ERD15基因的转录水平,结果表明ERD15在莲座叶、茎、花和未成熟的长角果中均有表达[12],这与该基因缺失后突变体与野生型出现差异表型的部位相一致,揭示ERD15基因对于拟南芥植株正常的生长发育起重要作用。当植物从营养生长向生殖生长过渡时,茎顶分生组织会转化成初生花序分生组织,后者直接产生花芽[29], 并且有研究表明,拟南芥dellaP突变体出现主茎增长,花原基数目增多与花序分生组织活性增加有关[23]。本研究中观察到erd15突变体的主茎比野生型更宽,单个花序的花数比野生型更多,这与dellaP突变体的表型相似,推测ERD15基因的缺失可能会增强花序分生组织的活性。此外,erd15突变体角果缩短,出现褶皱且顶端膨大成楔形,心皮数增多,无明显分隔的种子腔,且平均结籽数比野生型多,但野生型和erd15突变体叶片珠叶绿素荧光参数,包括PSII实际捕获能量的传递效率Fv'/Fm'、PSII实际的光化学量子效率Fq'/Fm'和植物热耗散能力指标NPQ等没有显著差异,这表明ERD15基因的缺失加速了拟南芥开花,影响生长,但不是通过影响植株的光合作用来改变的。下一步可使用扫描电镜观察比较野生型和突变体花序分生组织活性和雌蕊发育过程,深入探究ERD15基因影响拟南芥生长发育的分子机制。
| [1] |
ZIAF K, MUNIS M F H, SAMIN G, et al. Characterization of ERD15 gene from cultivated tomato (Solanum lycopersicum)[J]. Pak J Agric Sci, 2016, 53(1): 27-33. DOI:10.21162/PAKJAS/16.3695 |
| [2] |
KIYOSUE T, YAMAGUCHI-SHINOZAKI K, SHINOZAKI K. Cloning of cDNAs for genes that are early-responsive to dehydration stress (ERDs) in Arabidopsis thaliana L.:Identification of three ERDs as HSP cognate genes[J]. Plant Mol Biol, 1994, 25(5): 791-798. DOI:10.1007/BF00028874 |
| [3] |
RAI A N, TAMIRISA S, RAO K V, et al. Retracted article:Brassica RNA binding protein ERD4 is involved in conferring salt, drought tolerance and enhancing plant growth in Arabidopsis[J]. Plant Mol Biol, 2016, 90(4-5): 375-387. DOI:10.1007/s11103-015-0423-x |
| [4] |
RAI A, SUPRASANNA P, D'SOUZA S F, et al. Membrane topology and predicted RNA-binding function of the 'early responsive to dehydration (ERD4)' plant protein[J]. PLoS One, 2012, 7(3): e32658. DOI:10.1371/journal.pone.0032658 |
| [5] |
LIU Y H, WANG T Y, LI Y. Review of early responsive genes expressed during dehydration in Arabidopsis sp.[J]. J China Agric Univ, 2009, 14(3): 7-11. 刘颖慧, 王天宇, 黎裕. 拟南芥脱水诱导早期应答基因研究进展[J]. 中国农业大学学报, 2009, 14(3): 7-11. DOI:10.3321/j.issn:1007-4333.2009.03.003 |
| [6] |
RYU H Y, KIM S Y, PARK H M, et al. Modulations of AtGSTF10 expression induce stress tolerance and BAK1-mediated cell death[J]. Biochem Biophys Res Commun, 2009, 379(2): 417-422. DOI:10.1016/j.bbrc.2008.11.156 |
| [7] |
DUNAEVA M, ADAMSKA I. Identification of genes expressed in response to light stress in leaves of Arabidopsis thaliana using RNA differential display[J]. Eur J Biochem, 2001, 268(21): 5521-5529. DOI:10.1046/j.1432-1033.2001.02471.x |
| [8] |
KIYOSUE T, YAMAGUCHI-SHINOZAKI K, SHINOZAKI K. Characterization of two cDNAs (ERD10 and ERD14) corresponding to genes that respond rapidly to dehydration stress in Arabidopsis thaliana[J]. Plant Cell Physiol, 1994, 35(2): 225-231. DOI:10.1093/oxfordjournals.pcp.a078588 |
| [9] |
KARIOLA T, BRADER G, HELENIUS E, et al. Early responsive to dehydration 15, a negative regulator of abscisic acid responses in Arabidopsis[J]. Plant Physiol, 2006, 142(4): 1559-1573. DOI:10.1104/pp.106.086223 |
| [10] |
KIYOSUE T, YAMAGUCHI-SHINOZAKI K, SHINOZAKI K. ERD15, a cDNA for a dehydration-induced gene from Arabidopsis thaliana[J]. Plant Physiol, 1994, 106(4): 1707. DOI:10.1104/pp.106.4.1707 |
| [11] |
AALTO M K, HELENIUS E, KARIOLA T, et al. ERD15:An attenuator of plant ABA responses and stomatal aperture[J]. Plant Sci, 2012, 182: 19-28. DOI:10.1016/j.plantsci.2011.08.009 |
| [12] |
BRAVO J, AGUILAR-HENONIN L, OLMEDO G, et al. Four distinct classes of proteins as interaction partners of the PABC domain of Arabidopsis thaliana poly(A)-binding proteins[J]. Mol Genet Genom, 2005, 272(6): 651-665. DOI:10.1007/s00438-004-1090-9 |
| [13] |
XIE J W, KOZLOV G, GEHRING K. The "tale" of poly(A) binding protein:The MLLE domain and PAM2-containing proteins[J]. Biochim Biophys Acta Gene Regul Mech, 2014, 1839(11): 1062-1068. DOI:10.1016/j.bbagrm.2014.08.001 |
| [14] |
ALVES M S, REIS P A B, DADALTO S P, et al. A novel transcription factor, ERD15(early responsive to dehydration 15), connects endoplasmic reticulum stress with an osmotic stress-induced cell death signal[J]. J Biol Chem, 2011, 286(22): 20020-20030. DOI:10.1074/jbc.M111.233494 |
| [15] |
YU D D, ZHANG L H, ZHAO K, et al. VaERD15, a transcription factor gene associated with cold-tolerance in Chinese Wild Vitis amurensis[J]. Front Plant Sci, 2017, 8: 297. DOI:10.3389/fpls.2017.00297 |
| [16] |
SAEED B, KHURANA P. Transcription activation activity of ERD15 protein from Morus indica[J]. Plant Physiol Biochem, 2017, 111: 174-178. DOI:10.1016/j.plaphy.2016.11.020 |
| [17] |
TIMMUSK S, WAGNER E G H. The plant-growth-promoting rhizobacterium Paenibacillus polymyxa induces changes in Arabidopsis thaliana gene expression:A possible connection between biotic and abiotic stress responses[J]. Mol Plant Microbe Interact, 1999, 12(11): 951-959. DOI:10.1094/MPMI.1999.12.11.951 |
| [18] |
SUKWEENADHI J, KIM Y J, CHOI E S, et al. Paenibacillus yonginensis DCY84T induces changes in Arabidopsis thaliana gene expression against aluminum, drought, and salt stress[J]. Microbiol Res, 2015, 172: 7-15. DOI:10.1016/j.micres.2015.01.007 |
| [19] |
ZIAF K, LOUKEHAICH R, GONG P J, et al. A multiple stress-responsive gene ERD15 from Solanum pennellii confers stress tolerance in tobacco[J]. Plant Cell Physiol, 2011, 52(6): 1055-1067. DOI:10.1093/pcp/pcr057 |
| [20] |
SHAO H H, CHEN S D, ZHANG K, et al. Isolation and expression studies of the ERD15 gene involved in drought-stressed responses[J]. Genet Mol Res, 2014, 13(4): 10852-10862. DOI:10.4238/2014.december.19.6 |
| [21] |
KAUSHAL M. Microbes in cahoots with plants:Mist to hit the jackpot of agricultural productivity during drought[J]. Int J Mol Sci, 2019, 20(7): 1769. DOI:10.3390/ijms20071769 |
| [22] |
WU X B, WANG Q N, LING Q P, et al. Molecular identification and resistance evaluation to smut and mosaic disease with BC1 of Saccharum×Erianthus arundinaceus[J]. J Trop Subtrop Bot, 2019, 27(1): 45-52. 吴小斌, 王勤南, 凌秋平, 等. 甘蔗与斑茅BC1分子鉴定、抗黑穗病和花叶病初步评价[J]. 热带亚热带植物学报, 2019, 27(1): 45-52. DOI:10.11926/jtsb.3926 |
| [23] |
WANG W, ZHANG J, QIN Q Q, et al. The six conserved serine/threonine sites of repressor of ga1-3 protein are important for its functionality and stability in gibberellin signaling in Arabidopsis[J]. Planta, 2014, 240(4): 763-779. DOI:10.1007/s00425-014-2113-3 |
| [24] |
FUENTES S, LJUNG K, SOREFAN K, et al. Fruit growth in Arabidopsis occurs via DELLA-dependent and DELLA-independent gibberellin responses[J]. Plant Cell, 2012, 24(10): 3982-3996. DOI:10.1105/tpc.112.103192 |
| [25] |
ALVAREZ-BUYLLA E R, BENÍTEZ M, CORVERA-POIRÉ A, et al. Flower development[J]. Arabidopsis Book, 2010, 8: e0127. doi: 10.1199/tab.0127.
|
| [26] |
LU Y, LONG H. The effect of leaf number-altered mutants in Arabidopsis thaliana[J]. Chin Bull Bot, 2015, 50(3): 331-337. 卢阳, 龙鸿. 拟南芥叶片数目变化突变体对营养生长时相转变的影响[J]. 植物学报, 2015, 50(3): 331-337. |
| [27] |
ZHANG S R. A discussion on chlorophyll fluorescence kinetics parameters and their significance[J]. Chin Bull Bot, 1999, 16(4): 444-448. 张守仁. 叶绿素荧光动力学参数的意义及讨论[J]. 植物学通报, 1999, 16(4): 444-448. DOI:10.3969/j.issn.1674-3466.1999.04.021 |
| [28] |
GUO H, LIU P, DENG L, et al. Effect of light intensity on growth and chlorophyll fluorescence parameters of Belosynapsis ciliata[J]. J Trop Subtrop Bot, 2018, 26(3): 255-261. 郭微, 刘萍, 邓磊, 等. 不同光照强度对假紫万年青生长和叶绿素荧光参数的影响[J]. 热带亚热带植物学报, 2018, 26(3): 255-261. DOI:10.11926/jtsb.3816 |
| [29] |
DENAY G, CHAHTANE H, TICHTINSKY G, et al. A flower is born:An update on Arabidopsis floral meristem formation[J]. Curr Opin Plant Biol, 2017, 35: 15-22. DOI:10.1016/j.pbi.2016.09.003 |
 2020, Vol. 28
2020, Vol. 28



