2. 海南医学院基础医学与生命科学学院, 海口 571199
2. School of Basic Medicine and Life Science, Hainan Medical College, Haikou 571199, China
随着全球经济与科技的不断发展,人类对石油等化石燃料的需求不断增加,造成全球气候变暖、环境污染等问题日趋严重,寻求更加安全,更加环保的能源已经成为各国关注的焦点[1]。生物柴油的生产主要是通过动植物油脂与甲醇(或乙醇)经过酯交换反应得到的脂肪酸甲酯或者脂肪酸乙酯,是有望替代石油的可再生清洁燃料[2]。微藻因其生长速度快,生长周期短,含油量高,不占用耕地等优点,在生产生物柴油方面有着无法比拟的优越性[3]。
微藻生物柴油研究的热点是“产油微藻的筛选”、“微藻油高产策略”、“缺氮诱导微藻油脂”。目前,提高微藻产油率的方法主要有优化微藻的培养条件[4]、改变微藻的培养方式[5],微藻诱变[6]和基因工程技术定向改造微藻代谢途径[7],有研究表明对微藻细胞内的一些代谢关键酶基因进行改造可以促进细胞内油脂的积累[8-10],因此对微藻油脂合成及调控机理研究,对基因改造微藻具有重要的意义。
生物体内蛋白质降解的主要途径除了溶酶体途径,还有泛素-蛋白酶体途径(ubiquitin-proteasome pathway, UPP)。UPP水解过程由蛋白泛素化和蛋白质水解两部分组成。26S蛋白酶体是真核细胞内降解泛素化底物的一个ATP依赖型水解复合体, 是细胞内蛋白质水解的主要部位。在结构上可以分为20S核心颗粒(core protease, CP)和19S调节颗粒(regulatory particle, RP),CP是26S蛋白酶体的水解中心,由两个外环α和两个内环β堆叠而成的一个空心圆柱体样结构,每层环由7个亚基组成, CP圆柱体空心结构中心的2个β环是其活性位点, 含有胰蛋白酶、糜蛋白酶和谷氨酰样肽水解活性, 能够使大多数肽键断裂[11]。研究证实蛋白酶体与多种疾病有密切的关系,其可以通过水解,间接调控细胞分裂、细胞凋亡、信号转导,以及蛋白跨膜定位[12-14]。
大量文献报道,在能源和碳源充足的情况下, 逆境条件诱导微藻油脂的积累,其中这些条件有营养元素的缺乏,例如氮、硫、磷、铁、硅等,温度升高等[15-17]。其中缺氮诱导中性脂积累最为显著。其主要原因是氮是酶、膜等细胞大分子的重要组成部分,氮胁迫会影响细胞的分裂和增值,同时营养缺陷促使非脂类物质向脂类转化、微藻油脂积累。微藻的三酰甘油(TAG)途径与高等植物十分相似[18], 可以为微藻的初始研究提供了一个相对成熟的代谢通路框架, 但是微藻作为单细胞生物和高等植物有很大的区别,其次在藻细胞中也存在很多的旁路代谢途径[18-19],对于微藻油脂合成及调控相关基因的挖掘对于揭示微藻油脂代谢网络具有重要意义。因此,本实验室通过莱茵衣藻(Chlamydomonas rein- hardtii)氮胁迫基因数字表达谱挖掘筛选油脂代谢调控相关的基因,莱茵衣藻在缺氮培养下CrPOA1 mRNA水平大幅度降低[20],而莱茵衣藻在缺氮诱导下中性油脂积累增加,莱茵衣藻POA1基因表达的异常是否与油脂的积累有关?本试验通过RNAi干涉技术和亚细胞定位技术研究POA1与莱茵衣藻油脂积累之间的关系,为高产油微藻基因工程改造提供科学证据。
1 材料和方法 1.1 材料试验藻株 莱茵衣藻(Chlamydomonas reinhardtii)细胞壁缺失型藻株CC425购于中国科学院水生生物研究所;培养条件:24℃, 光照强度110 μmol/(m2·s), 用Tris-Acetate-Phosphate (TAP)固体培养基继代培养,液体培养时采用50 mL Sueoka高盐培养基(HSM), 在24℃, 光照强度110 μmol/(m2·s)下200 r/min振荡培养。大肠杆菌DH5α (本实验室保存)用于载体构建,采用LB培养基于37℃, 220 r/min下振荡培养。
抗生素终浓度 100 μg/mL氨苄青霉素(ampi- cillin);50 μg/mL硫酸卡娜霉素(kanamycin mono- sulfate);5 μg/mL巴龙霉素(paromomycin);5 mmol/L 5-氟吲哚(5-fluoroindol);1.5 mmol/L L-色氨酸; 10 μg/mL尼罗红(nile red)。
载体 RNAi干涉载体pMaa7/XIR (美国Duke大学衣藻中心)、pT282载体(本实验室构建)、pMD18-T [宝生物工程(大连)有限公司]。
试剂和试剂盒 M-MLV反转录酶、荧光定量试剂、限制性内切酶均购置于宝生物工程(大连)有限公司;总RNA提取试剂(Trizol)、Taq DNA聚合酶、T4 DNA连接酶、PCR产物纯化试剂盒、质粒小量抽提试剂盒、胶回收试剂盒等均购自生工生物工程(上海)股份有限公司。
1.2 莱茵衣藻缺氮培养下CrPOA1表达分析莱茵衣藻培养至对数生长期,于3 500×g下离心5 min收集藻细胞,HSM-N培养基洗藻细胞3次, 重新悬浮后接种于HSM-N培养基连续培养4 d, 取样液氮速冻研磨后按照Trizol总RNA抽提试剂盒抽提总RNA。按照MMLV反转录试剂盒合成cDNA。按照SYBR®Premix Ex TaqTM II试剂盒说明书进行基因表达量检测。以莱茵衣藻18S rRNA基因作为内参基因,采用2△△CT法进行相对定量计算,采用SPSS 22.0进行显著性分析, 用Origin 2019 64bit作图。
1.3 CrPOA1基因扩增和RNAi载体构建将莱茵衣藻CC425接种到50 mL TAP培养基, 培养2~3 d至对数生长期, 于3 500×g下离心5 min收集藻细胞, 莱茵衣藻总RNA的抽提参照改良Trizol法[21]。参照M-MLV反转录试剂合成cDNA。根据Phytozome Chlamydomonas reinhardtii V5.5数据库公布的CrPOA1 (Cre17.g705400)序列,设计干涉片段的特异性引物,以cDNA为模板,PCR扩增干涉片段。插入pMD18-T载体, 得到载体pMD- CrPOA1。RNAi载体构建参照李兴涵等[22]的方法, 分别用Hind III/BamH I和Xba I/Sal I双酶切质粒pMD-CrPOA1,得到CrPOA1正向片段和反向片段, 先将正向片段插入pT282载体Hind III/BamH I位点,再将反向片段插入含有正向片段的pT282载体, 到含有CrPOA1正反片段的pT282载体。EcoR I酶切含有CrPOA1正反片段的pT282载体,回收产物连接EcoR I酶切并去磷酸化的pMaa7IR/XIR载体, 得到CrPOA1 RNAi干涉载体Maa7IR/CrPOA1IR。
1.4 RNAi转基因藻株构建用HSM培养基把莱茵衣藻CC425培养至对数生长期,于3 500×g下离心5 min收集藻细胞,采用Glass Beads的方法[23]转化莱茵衣藻。转化后将藻株转移至50 mL无菌离心管中,于24℃黑暗温育过夜。在3 500×g下离心5 min收集藻细胞,添加500 μL TAP培养基重悬,涂布于含L-色氨酸和paromomycin的TAP固体平板,吹干后置于光照培养箱中培养6~ 8 d。挑取单克隆,接种到含5-Fluoroindole的TAP固体平板上复筛。采用CTAB法提取基因组DNA, 根据载体正反向重复序列骨架,用Primer 5设计特异性引物,PCR扩增,检测目标片段是否插入莱茵衣藻基因组DNA中。选取CC425、maa7转基因藻株和3个CrPOA1 RNAi转基因藻株于HSM培养基培养4 d,Trizol试剂法提取藻株总RNA,反转录成cDNA,以18S rRNA基因做内参,分析藻株CrPOA1 mRNA水平。采用2–△△CT法进行相对定量计算,用SPSS 22.0进行显著性分析。
| 表 1 用于PCR扩增的引物 Table 1 Primers for PCR amplification |
在HSM培养基中将莱茵衣藻CC425培养至对数生长期,于3 500×g下离心5 min收集藻细胞, 重悬后接种于6瓶1 L的HSM培养基中,通气光照培养。取不同生长时长的莱茵衣藻CC425,用酶标仪测定490 nm处的吸光值(OD),取1 L藻液离心收集,烘干后称量,绘制OD490与细胞干质量的标准曲线。选取3个转基因藻株和CC425,接种于50 mL HSM培养基,每天测定OD490,绘制莱茵衣藻生长曲线。
将三酰甘油标样梯度稀释,用尼罗红染色,测定575 nm处的吸光值,绘制OD575与三酰甘油浓度的标准曲线,选取3个转基因藻株和CC425,接种于50 mL HSM培养基,尼罗红染色后测定OD575, 绘制莱茵衣藻油脂积累曲线。取培养3~4 d的藻液, 用尼罗红染色10 min,在荧光显微镜下观察藻细胞生长状态及油脂情况。
1.6 CrPOA1-GFP融合表达载体的构建及亚细胞定位根据Phytozome数据库公布的CrPOA1 (Cre17. g705400)序列设计引物,PCR扩增全长基因。回收产物,在CrPOA1 ORF两端分别设计含有Nco I和Spe I位点的引物,PCR扩增CrPOA1全长基因(去除终止密码子),PCR产物经Nco I和Spe I双酶切后连接pCAMBIA1302,转化大肠杆菌DH5α,得到CrPOA1-GFP融合表达载体。基因枪法转化洋葱表皮[24],黑暗培养16 h后在激光共聚焦显微镜蓝光488 nm激发光下观察CrPOA1-GFP。
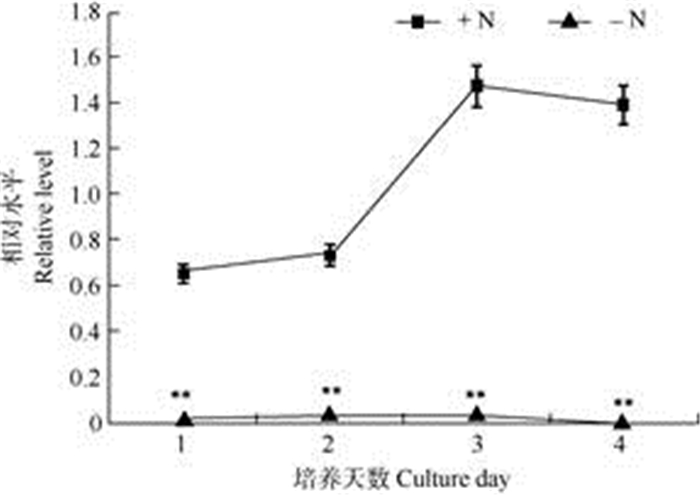
2 结果和分析 2.1 莱茵衣藻缺氮培养下CrPOA1表达分析从图 1可见,随着莱茵衣藻CC425在HSM-N连续培养时间的增加,CrPOA1 mRNA水平先升高后降低,与对照HSM+N相比,莱茵衣藻在HSM-N培养下的CrPOA1 mRNA水平显著降低(P < 0.01), 这说明CrPOA1可以响应低氮胁迫,与本实验室数字表达谱结果一致。
|
图 1 莱茵衣藻缺氮培养下CrPOA1表达分析 Fig. 1 Level of CrPOA1 mRNA of Chlamydomonas reinhardtii CC425 on HSM-N medium. **: P < 0.01. |
克隆的CrPOA1基因全长750 bp, 经生物信息学分析, CrPOA1编码含249个氨基酸残基的蛋白质, CrPOA1分子式为C1238H1947N329O371S4, 分子量(MW)为27.51 kDa,功能预测为20S蛋白酶体的α亚基(20S proteasome α subunit A, POA1),理论等电点(PI)为6.87,不稳定系数(II)为24.91,属稳定蛋白质。脂肪系数84.10,属亲水性蛋白质, 无跨膜结构区,亚细胞定位于细胞核中。
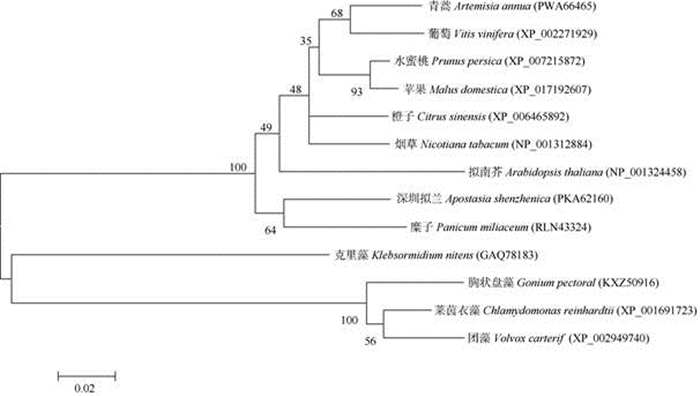
2.3 CrPOA1干涉片段的扩增扩增的莱茵衣藻20S蛋白酶体α亚基同源基因POA1在Phytozome 12 Chlamydomonas reinhardtiiV 5.5数据库(https://phytozome.jgi.doe.gov/pz/portal. html#!info?alias=Org_Creinhardtii)的登录号为Cre 17.g705400;GenBank序列号为XP_001691723.1。采用MEGA 6.0对POA1进行聚类分析(图 2),结果表明,CrPOA1与已报道的团藻(Volvox carterif, XP_ 002949740)、胸状盘藻(Gonium pectoral, KXZ 50916)的POA1进化关系较近。DNAMAN氨基酸同源性分析表明,CrPOA1与团藻和胸状盘藻的POA1氨基酸同源性分别为95.60%和94.00%。
2.4 RNAi载体构建及转基因藻株筛选按照图 3: A首先构建含有CrPOA1正反向干涉片段的pT282中间载体,将克隆的CrPOA1干涉片段(图 3: B)插入pMD18-T,得到pMD-CrPOA1, 通过Hind III、BamH I双酶切pMD-CrPOA1得到正向片段,插入pT282 Hind III和BamH I位点,通过Xba I和Sal I双酶切pMD-CrPOA1得到反向片段, 插入含正向片段T282载体的Xba I和Sal I位点, 得到含有正反向干涉片段的中间载体pT282, 通过EcoR I酶切含正反向片段的pT282载体获得正反向结构片段,插入pMaa7IR/XIR EcoR I位点,得到CrPOA1干涉载体pMaa7IR/CrPOA1IR (图 3: C)。
采用玻璃珠转化法转化莱茵衣藻CC425,转化藻株长出后接种于5-Fluoroindole TAP培养基进行复筛,提取基因组DNA,PCR检测表明pMaa7IR/ CrPOA1IR已经成功整合到莱茵衣藻CC425基因组中(图 3: D)。
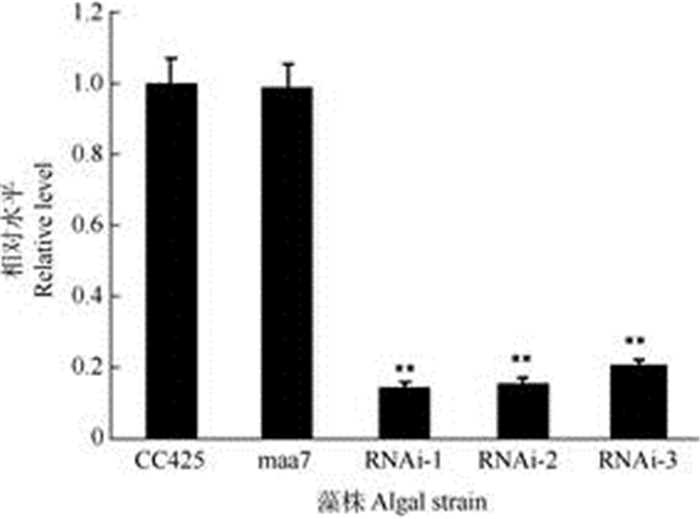
2.5 转基因藻株的基因表达CrPOA1 mRN水平直接说明了RNA干扰的效果,从图 4可见,3个RNAi转基因藻株RNAi-1、RNAi-2和RNAi-3的CrPOA1 mRNA水平比对照maa7显著降低,分别降低了85.35%、84.214%和79.36%,均达极显著差异(P < 0.01), 说明RNAi载体对CrPOA1沉默效果良好。
|
图 2 CrPOA1与其他物种POA1的聚类分析 Fig. 2 Cluster analysis of CrPOA1 and POA1 in other species |

|
图 3 干涉载体构建和转基因莱茵衣藻筛选。A: pT282载体和pMaa7IR/XIR载体, E: EcoR I, H: Hind III, B: BamH I, S: Sal I, X: Xba I; B: M: Marker DL 2000, 1: CrPOA1干涉片段; C: M: Marker DL 15000, 1: pMaa7 IR/CrPOA1IR EcoR I酶切; D: M: Marker DL 2000, 1~17:藻株。 Fig. 3 Construction of CrPOA1 RNAi interference vector and screening of transgenic Chlamydomonas reinhardtii. A: pT282 vector and pMaa7IR/XIR vector, E: EcoR I, H: Hind III, B: M: Marker DL 2000, 1: Interference fragment of CrPOA1; C: M: Marker DL 15000, 1: C: Digestion pMaa7 IR/CrPOA1IR by EcoR I; D: M: Marker DL2000, 1-17: Algal strains. |
|
图 4 转基因藻株的CrPOA1 mRNA水平. **: P < 0.01。 Fig. 4 CrPOA1 mRNA level of transgenic alga. **: P < 0.01. |
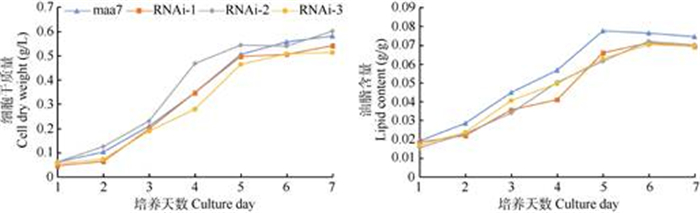
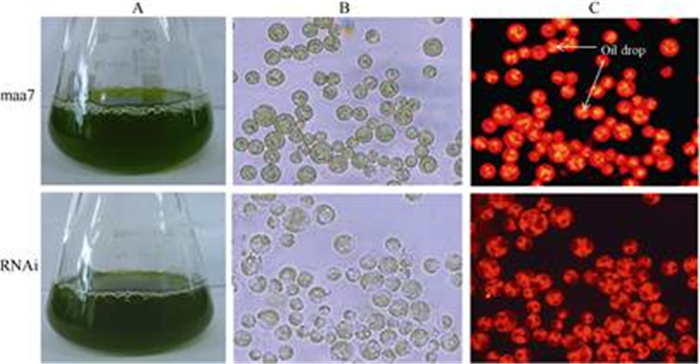
从图 5可见,连续培养7 d的CrPOA1 RNAi转基因藻株细胞干质量随培养时间的延长而增加, 第1~2天增加缓慢,第3~5天增加迅速,第6天趋于稳定,符合微生物培养生长的“S”型曲线,与对照maa7相比无显著差异。随着培养时间的延长, 细胞内油脂逐渐积累,CrPOA1 RNAi转基因藻株的油脂积累比对照maa7缓慢,培养3~5 d与对照的差异达显著水平(P < 0.05),转基因藻株培养第5天的油脂含量达最大值,比对照下降了6.38%~ 24.63%,这说明CrPOA1对莱茵衣藻油脂积累具有促进作用。莱茵衣藻在连续培养过程中,随着油脂的积累,先形成小油体,油体数目逐渐增加,体积增大,然后,小油体融合形成大油体。培养5 d的转基因藻细胞,经尼罗红染色10 min后在荧光显微下观察, 对照maa7藻细胞中有大量油体存在,而RNAi转基因藻细胞内油体数量较少(图 6)。
|
图 5 CrPOA1 RNAi转基因藻株的细胞干质量和油脂含量 Fig. 5 Cell dry weight and lipid content of CrPOA1 RNAi transgenic strains |
|
图 6 CrPOA1 RNAi转基因藻株培养和显微镜检。A:培养状态; B:明场(40x10); B:暗场(荧光40x10);棕黄色代表油体。 Fig. 6 CrPOA1 RNAi transgenic algae after cultured 5 days under microscope. A: Culture state; B: Bright field (40x10); C: Dark field (fluorescence 40x10); Yellow was oil drop. |
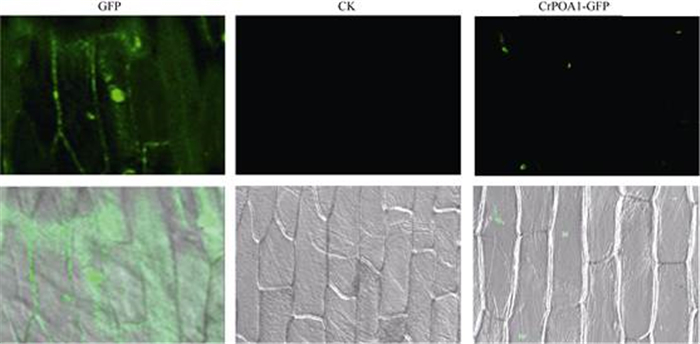
蛋白质的功能与其亚细胞位置有着密切的关系,蛋白质只有被运送到特定的细胞器中,才能发挥正常的生物学功能。洋葱(Allium cepa)表皮细胞因遗传背景清晰、便于操作、相对植物蛋白定位准确度高等优点,被广泛用于植物蛋白的亚细胞定位研究。为研究CrPOA1的亚细胞定位,将构建的CrPOA1-GFP融合表达载体,用基因枪法转化洋葱表皮细胞,以pCAMBIA1302空载体做阳性对照(GFP),以蒸馏水做阴性对照(CK)。从图 7可见, 阳性对照细胞质和细胞核中都有GFP的表达,说明35S CaMV启动子能够驱动GFP基因的表达,阴性对照细胞核、细胞质都未见绿色荧光,转化CrPOA1-GFP的细胞核有荧光信号,细胞质未见荧光信号,说明CrPOA1-GFP位于细胞核中。这与生物信息预测的结果一致。
|
图 7 CrPOA1在洋葱表皮中的瞬时表达(40x10)。GFP: pCAMBIA1302; CK:蒸馏水; CrPOA1-GFP: pCAMBIA1302-CrPOA1-GFP。 Fig. 7 Transient expression of CrPOA1 in onion epidermis (40x10). FPG: pCAMBIA1302; CK: Distilled water; CrPOA1-GFP: pCAMBIA1302-CrPOA1-GFP. |
UPP是目前已知最有效、最具可行性的蛋白质降解途径,参与细胞多项生命活动。植物生长发育的诸多方面和胁迫响应等过程都受到该途径的调控[25]。大量研究报道UPP中主要的组成,如E2泛素结合酶、E3泛素连接酶和蛋白酶体等对低氮胁迫等环境胁迫做出响应[26]。植物在高温、高pH (pH > 7)、低氮、低磷等环境因子胁迫下,UPP途径加强以抵抗这些非生物胁迫,NLA基因(E3)在细胞核内与AtUBC8互作共同调控拟南芥(Arabidopsis thaliana)对氮环境的适应性反应[27]。本研究结果表明,低氮胁迫下,CrPOA1下调表达,进一步影响了UPP, 导致UPP对异常蛋白的降解。
3.2 UPP参与油脂的代谢调控UPP参与藻类等植物的非生物胁迫响应应答。而大量研究表明,大多数微藻在低氮、低硫、低磷、低铁、低硅等和高温、高pH[17, 28-30]等环境因子胁迫下油脂大幅度增加, 以缺氮诱导最为显著。因此, 我们判断UPP参与莱茵衣藻油脂的代谢调控。目前, UPP如何参与莱茵衣藻油脂代谢的调控鲜有报道, 但是,大量研究报道UPP的主要组成成员,如E2泛素结合酶、E3泛素连接酶和蛋白酶体等,参与调控藻类和其他植物的脂类代谢。Fei等[31]的研究表明, 莱茵衣藻油脂含量和E2泛素结合酶基因CrUBC2存在正相关。Lü等[32]研究了CER9 E3泛素连接酶调控拟南芥的油脂合成机理,认为CER9基因功能缺失突变体cer9-2的种子含油率比Col-0野生型增加了17%,通过酵母双杂交并筛选出其互作蛋白基因编码内质网溶血磷脂酰基转移酶LPAT2。Luo等[33]研究了U-box E3泛素连接酶基因家族,认为有效沉默CrPUB5和CrPUB14后莱茵衣藻油脂积累增加了9.8%~45.0%和14.4%~61.8%。van Loon等[34]的研究表明E3泛素连接酶的失活可减轻小鼠饮食诱导的肥胖和代谢功能紊乱。Oliva等[35]对注射蛋白酶体抑制剂PS-341的大鼠肝脏进行了芯片分析, 表明SREBP-1C mRNA表达显著下降, 其下游脂类合成相关酶,如脂肪酸合成酶(FAS)和乙酰辅酶A羧化酶(ACC)活性显著降低,同时降低了参与三酰甘油(TAG)合成的酰基甘油-3-磷酸酰基转移酶(AGPAT)和二酰甘油酰基转移酶(DGAT)基因的表达。
本研究通过RNA干扰技术对CrPOA1进行沉默,结果藻细胞油脂的积累降低。CrPOA1沉默必然影响26S蛋白酶体的组装,UPP对异常蛋白的降解作用被大大削弱,这可能影响油脂合成关键酶基因的表达,如FAS、ACC、AGPAT、DGAT[35]。微藻三酰甘油合成与植物的类似,大量研究表明通过基因工程的手段降低脂类的分解代谢以及抑制脂类流向蛋白质和碳水化合物,可以提高油脂的产率[36]。沉默脂类代谢的酰基CoA氧化酶、酰基CoA合成酶、肉碱酰基转移酶I、脂酰基CoA脱氢酶基因, 可有效提高藻细胞内脂类的含量[37]。莱茵衣藻中ADP-葡萄糖焦磷酸化酶(AGPase)活性丧失,细胞内淀粉合成受阻,脂类含量增加10倍[38]。UPP对异常蛋白质降解的减弱,也可能引起藻细胞内碳代谢异常,使藻细胞内碳向淀粉、蛋白质等物质的转化,从而油脂含量降低。
综上所述,本研究为揭示UPP对藻类油脂的调控提供了一定的证据,CrPOA1响应低氮胁迫,其可能是莱茵衣藻在低氮胁迫下通过UPP响应来抵抗胁迫。CrPOA1沉默后UPP对异常蛋白的降解减弱,可能引起油脂合成关键酶基因和底物降低,从而导致藻细胞油脂含量的降低。UPP对异常蛋白的降低也可能是CrPOA1参与调控脂类的代谢调控, 促进脂类向糖类、蛋白质等物质的转化,从而引起藻细胞内油脂的降低。因此,后续应从POA1对莱茵衣藻碳流分配的影响以及POA1互做蛋白的筛选等方面开展工作。
| [1] |
ZHANG D H, WANG J Q, LIN Y G, et al. Present situation and future prospect of renewable energy in China[J]. Renew Sust Energ Rev, 2017, 76: 865-871. DOI:10.1016/j.rser.2017.03.023 |
| [2] |
USMANOV R A, GABITOV R R, BIKTASHEV S A, et al. Pilot unit for permanent transesterification of vegetable oils in supercritical methanol or ethanol media[J]. Russ J Phys Chem B, 2011, 5(8): 1216-1227. DOI:10.1134/S1990793111080112 |
| [3] |
MA C, LIU B F, REN H Y, et al. A review on biodiesel production:Breeding technologies of microalgae containing rich lipid[J]. Appl Mechan Mat, 2014, 472: 759-763. DOI:10.4028/www.scientific.net/AMM.472.759 |
| [4] |
GIM G H, RYU J, KIM M J, et al. Effects of carbon source and light intensity on the growth and total lipid production of three microalgae under different culture conditions[J]. J Ind Microbiol Biotechnol, 2016, 43(5): 605-616. DOI:10.1007/s10295-016-1741-y |
| [5] |
JUAN S, KEHOU P. Effects of different culture conditions and growth phases on lipid of microalgae[J]. Mar Fish Res, 2004, 25(6): 79-85. |
| [6] |
ANTHONY J, RANGAMARAN V R, GOPAL D, et al. Ultraviolet and 5' fluorodeoxyuridine induced random mutagenesis in Chlorella vulgaris and its impact on fatty acid profile:A new insight on lipid-metabolizing genes and structural characterization of related proteins[J]. Mar Biotechnol, 2015, 17(1): 66-80. DOI:10.1007/s10126-014-9597-5 |
| [7] |
SUN P P, FAN C M, CHEN Y H, et al. The research advances on microalgae lipid synthesis and regulation by biochemical and genetic engineering[J]. Mol Plant Breed, 2017, 15(7): 2628-2635. 孙配配, 范成明, 陈宇红, 等. 微藻油脂合成与调控的生化及基因工程研究进展[J]. 分子植物育种, 2017, 15(7): 2628-2635. DOI:10.13271/j.mpb.015.002628 |
| [8] |
GUSCHINA I A, HARWOOD J L. Lipids and lipid metabolism in eukaryotic algae[J]. Prog Lipid Res, 2006, 45(2): 160-186. DOI:10.1016/j.plipres.2006.01.001 |
| [9] |
DENG X D, LI Y J, FEI X W. The mRNA abundance of pepc2 gene is negatively correlated with oil content in Chlamydomonas reinhardtii[J]. Biomass Bioenerg, 2011, 35(5): 1811-1817. DOI:10.1016/j.biombioe.2011.01.005 |
| [10] |
BAURAIN D, DINANT M, COOSEMANS N, et al. Regulation of the alternative oxidase Aox1 gene in Chlamydomonas reinhardtii:Role of the nitrogen source on the expression of a reporter gene under the control of the Aox1 promoter[J]. Plant Physiol, 2003, 131(3): 1418-1430. DOI:10.1104/pp.013409 |
| [11] |
LI Y, LIANG J Y, LIU X L, et al. Research progress on 26S protea-some inhibitors[J]. Acta Pharm Sin, 2017, 52(4): 524-530. 李媛, 梁俊玉, 刘鑫龙, 等. 26S蛋白酶体抑制剂研究进展[J]. 药学学报, 2017, 52(4): 524-530. DOI:10.16438/j.0513-4870.2016-1082 |
| [12] |
LECKER S H, SOLOMON V, MITCH W E, et al. Muscle protein breakdown and the critical role of the ubiquitin-proteasome pathway in normal and disease states[J]. J Nutr, 1999, 129(1): 227S-237S. DOI:10.1093/jn/129.1.227s |
| [13] |
TU Y Q, CHEN C, PAN J R, et al. The ubiquitin proteasome pathway (UPP) in the regulation of cell cycle control and DNA damage repair and its implication in tumorigenesis[J]. Int J Clin Exp Pathol, 2012, 5(8): 726-738. |
| [14] |
SHANG F, TAYLOR A. Roles for the ubiquitin-proteasome pathway in protein quality control and signaling in the retina:Implications in the pathogenesis of age-related macular degeneration[J]. Mol Aspects Med, 2012, 33(4): 446-466. DOI:10.1016/j.mam.2012.04.001 |
| [15] |
WANG B, LI Y Q, WU N, et al. CO2 bio-mitigation using microalgae[J]. Appl Microbiol Biot, 2008, 79(5): 707-718. DOI:10.1007/s00253-008-1518-y |
| [16] |
HOLCOMB R E, MASON L J, REARDON K F, et al. Culturing and investigation of stress-induced lipid accumulation in microalgae using a microfluidic device[J]. Anal Bioanal Chem, 2011, 400(1): 245-253. DOI:10.1007/s00216-011-4710-3 |
| [17] |
LONGWORTH J, WU D Y, HUETE-ORTEGA M, et al. Proteome response of Phaeodactylum tricornutum, during lipid accumulation induced by nitrogen depletion[J]. Algal Res, 2016, 18: 213-224. DOI:10.1016/j.algal.2016.06.015 |
| [18] |
BELLOU S, BAESHEN M N, ELAZZAZY A M, et al. Microalgal lipids biochemistry and biotechnological perspectives[J]. Biotechnol Adv, 2014, 32(8): 1476-1493. DOI:10.1016/j.biotechadv.2014.10.003 |
| [19] |
LEE J W, SHIN S Y, KIM H S, et al. Lipid turnover between membrane lipids and neutral lipids via inhibition of diacylglyceryl N, N, N-trimethylhomoserine synthesis in Chlamydomonas reinhardtii[J]. Algal Res, 2017, 27: 162-169. DOI:10.1016/j.algal.2017.09.001 |
| [20] |
LI Y J, FEI X W, DENG X D. Digital gene expression profile of Chlamydomonas reinhardtii grown under nitrogen starvation[J]. Chin J Trop Agric, 2012, 32(11): 66-70. 李亚军, 费小雯, 邓晓东. 莱茵衣藻氮胁迫基因数字表达谱分析[J]. 热带农业科学, 2012, 32(11): 66-70. DOI:10.3969/j.issn.1009-2196.2012.11.015 |
| [21] |
LI Y J, FEI X W, DENG X D. Two protocols for total RNA extraction from microalgae[J]. Chin J Trop Agric, 2011, 31(5): 24-27. 李亚军, 费小雯, 邓晓东. 2种微藻总RNA提取方法的比较[J]. 热带农业科学, 2011, 31(5): 24-27. DOI:10.3969/j.issn.1009-2196.2011.05.007 |
| [22] |
LI X H, FEI X W, DENG X D. Construction of phosphofructokinase RNAi vectors and its impact on lipid accumulation in Chlamydomonas reinhardtii[J]. Guangdong Agric Sci, 2014, 41(10): 155-159. 李兴涵, 费小雯, 邓晓东. 莱茵衣藻磷酸果糖激酶RNAi载体构建及其对莱茵衣藻油脂积累的影响[J]. 广东农业科学, 2014, 41(10): 155-159. DOI:10.3969/j.issn.1004-874X.2014.10.035 |
| [23] |
KINDLE K L. High-frequency nuclear transformation of Chlamydo-monas reinhardtii[J]. Proc Natl Acad Sci USA, 1990, 87(3): 1228-1232. DOI:10.1073/pnas.87.3.1228 |
| [24] |
YU Y F, ZHU X B, GE H M, et al. Subcellular localization of plants based on transient expression of green fluorescent protein[J]. Jiangsu Agric Sci, 2014, 42(12): 58-61. 于一帆, 朱小彬, 葛会敏, 等. 基于绿色荧光蛋白瞬时表达的植物亚细胞定位方法[J]. 江苏农业科学, 2014, 42(12): 58-61. DOI:10.15889/j.issn.1002-1302.2014.12.017 |
| [25] |
XU D D, SUN F, WANG Y X, et al. Biological function insights into the ubiquitin/26S proteasome pathway in rice[J]. J Agric Sci Technol, 2018, 20(1): 25-33. 徐丹丹, 孙帆, 王银晓, 等. 泛素/26S蛋白酶体途径在水稻中的生物学功能研究进展[J]. 中国农业科技导报, 2018, 20(1): 25-33. DOI:10.13304/j.nykjdb.2017.0144 |
| [26] |
WANG J L, SHI S Q, JIA L Q, et al. Progress on functions of ubiquitin-conjugating enzyme (E2) in plants[J]. Biotechnol Bull, 2010, 20(4): 7-10. 王金利, 史胜青, 贾利强, 等. 植物泛素结合酶E2功能研究进展[J]. 生物技术通报, 2010, 20(4): 7-10. |
| [27] |
PENG M S, HANNAM C, GU H L, et al. A mutation in NLA, which encodes a RING-type ubiquitin ligase, disrupts the adaptability of Arabidopsis to nitrogen limitation[J]. Plant J, 2007, 50(2): 320-337. DOI:10.1111/j.1365-313X.2007.03050.x |
| [28] |
HOUNSLOW E, KAPOORE R V, VAIDYANATHAN S, et al. The search for a lipid trigger:The effect of salt stress on the lipid profile of the model microalgal species Chlamydomonas reinhardtii for biofuels production[J]. Curr Biotechnol, 2016, 5(4): 305-313. DOI:10.2174/2211550105666160322234434 |
| [29] |
CHAISUTYAKORN P, PRAIBOON J, KAEWSURALIKHIT C. The effect of temperature on growth and lipid and fatty acid composition on marine microalgae used for biodiesel production[J]. J Appl Phycol, 2018, 30(1): 37-45. DOI:10.1007/s10811-017-1186-3 |
| [30] |
PHILIPPS G, HAPPE T, HEMSCHEMEIER A. Nitrogen deprivation results in photosynthetic hydrogen production in Chlamydomonas reinhardtii[J]. Planta, 2012, 235(4): 729-745. DOI:10.1007/s00425-011-1537-2 |
| [31] |
FEI X W, LI X H, LI P, et al. Involvement of Chlamydomonas DNA damage tolerence gene UBC2 in lipid accumulation[J]. Algal Res, 2017, 22: 148-159. DOI:10.1016/j.algal.2016.12.019 |
| [32] |
LÜ S Y, ZHAO H Y, MARAIS D L D, et al. Arabidopsis ECERI FERUM9 involvement in cuticle formation and maintenance of plant water status[J]. Plant Physiol, 2012, 159(3): 930-944. DOI:10.1104/pp.112.198697 |
| [33] |
LUO Q L, LI Y J, WANG W Q, et al. Genome-wide survey and expression analysis of Chlamydomonas reinhardtii U-box E3 ubiquitin ligases (CrPUBs) reveal a functional lipid metabolism module[J]. PLoS One, 2015, 10(11): e0142996. DOI:10.1371/journal.pone.0122600 |
| [34] |
van LOON N M, OTTENHOFF R, KOOIJMAN S, et al. Inactivation of the E3 ubiquitin ligase IDOL attenuates diet-induced obesity and metabolic dysfunction in mice[J]. Arterioscle Thrombosis Vascul Biol, 2018, 38(8): 1785-1795. DOI:10.1161/ATVBAHA.118.311168 |
| [35] |
OLIVA J, FRENCH S W, LI J, et al. Proteasome inhibitor treatment reduced fatty acid, triacylglycerol and cholesterol synthesis[J]. Exp Mol Pathol, 2012, 93(1): 26-34. DOI:10.1016/j.yexmp.2012.03.006 |
| [36] |
YU W L, ANSARI W, SCHOEPP N G, et al. Modifications of the meta-bolic pathways of lipid and triacylglycerol production in microalgae[J]. Microb Cell Fact, 2011, 10(1): 91. DOI:10.1186/1475-2859-10-91 |
| [37] |
HE S S, WANG Y L, GAO B Y, et al. Biosynthetic pathway of triacyl-glycerol in microalgae and its latest research progress[J]. Chin Bull Life Sci, 2014, 26(9): 979-990. 何思思, 王元丽, 高保燕, 等. 微藻三酰甘油合成途径及其最新研究进展[J]. 生命科学, 2014, 26(9): 979-990. DOI:10.13376/j.cbls/2014141 |
| [38] |
LI Y T, HAN D X, HU G R, et al. Chlamydomonas starchless mutant defective in ADP-glucose pyrophosphorylase hyper-accumulates triacyl-glycerol[J]. Metab Eng, 2010, 12(4): 387-391. DOI:10.1016/j.ymben.2010.02.002 |
 2020, Vol. 28
2020, Vol. 28


