琴叶风吹楠(Horsfieldia pandurifolia)是肉豆蔻科(Myristicaceae)高大乔木,濒危种,属于典型的热带雨林树种[1-3],种子含油率高,且脂肪酸成分主要为肉豆蔻酸,可用于工业、化妆品等,是珍稀的油料资源植物。据《中国植物志》和《云南植物志》记载,琴叶风吹楠分布于澜沧江流域以及与缅甸、老挝、越南接壤的国境线一带。吴裕等[4]对分布在云南省内的琴叶风吹楠野生资源进行了调查,其分布区零散,且现存的资源种群数量小,地理跨度大,呈斑块状分布。
对珍稀濒危植物遗传多样性进行研究是了解其濒危机制的基础[5],在保护濒危植物种群数量的同时,还需保护其遗传多样性和进化潜力,根据遗传基础制定科学合理的保护措施,因此,应对珍稀濒危植物进行居群遗传多样性特征和遗传结构研究。目前,利用分子生物学手段,如扩增片段长度多态性(AFLP, amplified fragment length polymorphism)、简单重复序列(SSR, simple sequence repeat)、核基因、叶绿体基因片段等,对濒危植物进行保护遗传学研究[6-9],在探明这些濒危植物遗传多样性的基础上, 提出了科学合理的保护措施;此外,应用分子生物学方法还分析了物种资源的遗传多样性、谱系地理及亲缘关系等[10-15]。
琴叶风吹楠已处于濒危状态[3],了解其遗传基础并制定合理的保护措施,降低灭绝风险和速度, 延续物种多样性显得十分重要,但现阶段对琴叶风吹楠的研究主要集中在分类学、油脂成分分析、资源调查和形态变异等方面。对居群间变异式样的研究少见报道,吴裕等[16-17]对不同居群间单株的果实和种子形态变异进行了分析,结果表明,琴叶风吹楠果实和种子形态以及种仁含油率和脂肪酸成分, 在居群内和居群间存在不同程度的变异。而有关琴叶风吹楠分子遗传学方面的研究报道甚少,仅毛常丽等对其叶绿体基因组进行了测序[18]。本研究采用AFLP标记方法,对云南省分布的琴叶风吹楠居群进行遗传多样性及遗传结构分析,为琴叶风吹楠的开发利用及可持续发展奠定基础,并提出适宜的保护策略。
1 材料和方法 1.1 材料从云南省西双版纳州景洪市、勐腊县、临沧市双江县的8个琴叶风吹楠(Horsfieldia pandurifolia)居群(表 1),采集56株树的嫩叶或嫩树皮,液氮保存。
| 表 1 琴叶风吹楠8个居群信息 Table 1 Informations of 8 Horsfieldia pandurifolia populations |
用QIAGEN试剂盒改良法提取琴叶风吹楠基因组DNA,用NanoDrop2000超微量分光光度计测定DNA浓度和OD值,质量合格的DNA样品送生工生物工程(上海)股份有限公司进行荧光AFLP分析。
1.3 数据分析获得的AFLP分析数据进行人工校对后用POPGENE vesion 1.32软件[19-20]计算观测等位基因数(Na)、有效等位基因数(Ne)、Nei’s多样性指数(H)、Shannon多样性指数(Ⅰ)、遗传一致度等遗传多样性指标。基于popgene计算得到的遗传一致度数据, 用NTSYSpc-2.10e软件的Clustering中SAHN程序对8个琴叶风吹楠居群进行UPGMA聚类[21]。用“经纬度计算距离软件”将各居群的经纬度换算成地理距离,用TFPGA1.3软件进行mantel相关性检验。Winamova软件进行AMOVA分析。
2 结果和分析 2.1 荧光AFLP扩增多态性用QIAGEN试剂盒改良法提取的56份琴叶风吹楠基因组DNA能够满足本试验要求。从64对组合引物中选出6对多态性引物,分别为H-AAC/ M-CAG、H-ACA/M-CTC、H-ACG/M-CAA、H-AAC/ M-CAC、H-AGC/M-CAG、H-AGG/M-CAA。采用ABI377测序仪进行条带读取,每对引物读取216条带,6对引物共扩增出1 296条带,平均多态性位点162个,多态性百分率为75.16% (表 2)。
| 表 2 6对引物扩增结果 Table 2 Amplification results on the 6 pairs of primers |
用POPGENE vesion 1.32软件对8个居群的遗传参数进行计算(表 3)。可以看出,琴叶风吹楠居群内的观测等位基因数(Na)为1.217 2~1.532 5,有效等位基因数(Ne)为1.044 9~1.248 9,Nei’s多样性指数(H)为0.073 7~0.151 9,Shannon信息指数(Ⅰ)为0.236 1~0.112 0。各居群扩增的多态性位点百分率以NBH最大,SJ最小,其余各遗传参数的结果和多态性位点百分率显示结果一致,均以NBH最高,SJ最低。
| 表 3 琴叶风吹楠居群的遗传多样性 Table 3 Genetic diversity of Horsfieldia pandurifolia populations |
对琴叶风吹楠居群间的遗传分化分析可见(表 4),琴叶风吹楠的遗传参数在不同引物组合中的表现不同,总遗传多样性(Ht)为0.124 2~0.184 1, 居群内遗传多样性(Hs)为0.096 2~0.123 3,居群间遗传分化系数(Gst)为0.175 4~0.386 6,居群间遗传多样性(Dst)为0.026 2~0.071 2,Ht、Hs、Gst和Dst的平均值分别为0.149 5、0.112 8、0.238 6和0.036 7;物种水平的Ht、Hs、Gst和Dst分别为0.140 6、0.102 8、0.226 5和0.030 6。居群内的遗传变异占总变异的75.45%,居群间的占24.55%, 说明琴叶风吹楠的遗传变异主要存在于居群内。另外,不同引物组合的基因流(Nm)不同,为1.350 8~1.933 0,变化幅度不大,物种水平的基因流为1.707 5。
| 表 4 琴叶风吹楠居群的遗传结构参数 Table 4 Genetic structure parameters of Horsfieldia pandurifolia populations |
AMOVA分析表明,各个居群间的遗传差异达显著水平(P < 0.001),居群内的遗传变异为94.26%,居群间的为5.74%,物种遗传变异主要来自于群体内,这和Nei’s基因多样性、多态性位点百分率、Shannon信息指数的分析结果一致。
2.4 居群间遗传距离通过NTSYS软件计算琴叶风吹楠居群间的遗传距离和遗传一致度(表 5)。琴叶风吹楠居群的遗传距离为0.019 6~0.099 7,NBH与SL居群的遗传距离最小,为0.019 6,WTS与SJ居群的遗传距离最大,为0.099 7。琴叶风吹楠的遗传一致度为0.909 4~ 0.980 7, NBH与SL居群的遗传一致度最高,为0.980 7, WTS与SJ居群的遗传一致度最低,为0.909 4。
| 表 5 8个居群的Nei’s遗传一致度(右上角)和遗传距离(左下角) Table 5 Nei's genetic consistency (upper right corner) and genetic distance (lower left corner) among 8 populations |
将表 1中各居群的经纬度用“经纬度计算距离软件”换算成地理距离, 将地理距离与遗传距离进行mantel相关性检验,结果表明,地理距离和遗传距离之间存在一定的相关性(r=0.119 7,P=0.321 0), 但相关性不显著。
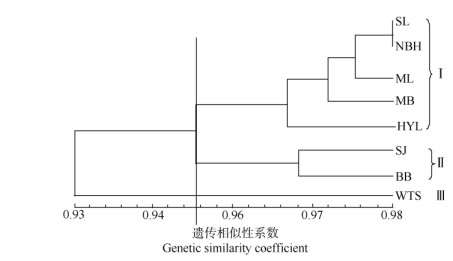
2.6 聚类分析对琴叶风吹楠居群进行UPGMA聚类分析(图 1),结果表明,当遗传相似性系数为0.951时,琴叶风吹楠居群可分为3大支,首先,地理距离较近的SL和NBH居群聚在一起,ML、MB和HYL等3个居群的地理距离较近,和SL及NBH居群聚在一起形成第Ⅰ支,SJ和BB居群地理距离较远,但遗传距离较近,两居群聚在一起形成第Ⅱ支,最后是WTS居群单独形成第Ⅲ支。
|
图 1 琴叶风吹楠的UPGMA聚类图 Fig. 1 UPGMA dendrogram of Horsfieldia pandurifolia |
物种的遗传多样性受多种因素影响,包括生态环境、生境周围的人类活动、演化史、种子散播机制等,同时,遗传多样性的高低决定了其适应环境的能力。本研究采用6对AFLP引物共扩增得到1 296条带, 平均每对引物扩增的多样性位点为162。在物种水平上,琴叶风吹楠的平均多态性百分率为75.16%,表明其具有丰富的遗传多样性;在居群水平上,平均多态性位点为100,平均多态性百分率为36.20%,比物种的遗传多样性低,推测远古时期琴叶风吹楠在云南地区可能成连续分布, 且分布范围广泛,遗传基础丰富,但受云贵高原隆起影响, 分布范围逐渐缩小,使琴叶风吹楠生境片断化,该物种在不断的地质变化中,各个相对隔离的居群受自然灾害等因素影响,使居群的数量减少,遗传多样性较整个物种降低。吴裕等[4]对云南省内琴叶风吹楠资源的调查结果也表明,其自然繁衍对生境的需求和人为导致濒危生境是造成种群数量锐减的主要原因,其居群内数量逐渐减少,即使具有祖先丰富的遗传多样性, 但由于现在分布区域的不连续性,导致居群规模不断缩小,其后果就是各个居群内遗传多样性下降。同时, 后代成活数量的差异也是导致有效居群数量下降的原因之一[22]。在对琴叶风吹楠进行调查的过程中,一些居群生长在密林, 周围还有更上层树种遮盖,这样的生长环境中,幼苗生长成大树的几率很低,而一些“开天窗”或林缘处的居群,幼苗能接收到的光、温、水适宜,其居群内幼树能成长为大树的几率较高。在这两种生长环境下,其后代的成活数量是有差异的,长此以往,有效居群大小发生变化,遗传变异度也在长期的繁殖及进化中发生改变。
基因的相互交流可引起居群内的遗传变异增加,减少居群间的分化[23-24]。遗传结构分析表明, 琴叶风吹楠物种的基因流为1.707 5,居群间的遗传分化系数为0.226 5,居群内遗传变异大于居群间的遗传变异,居群内变异是其变异的主要来源,变异式样和濒危植物思茅木姜子(Litsea szemaois)[8]及西双版纳地区分布的油料树种硬核(Scleropyrum wallichianum)[25]相似, 但与双子叶植物的遗传变异水平(Gs=0.273)对比[26],琴叶风吹楠的遗传分化程度稍低。
通过聚类分析,对琴叶风吹楠各个居群进行分组, 表明并不是所有居群均按地理距离远近聚类。地理距离和遗传距离之间存在不显著的正相关性, 但总体上还是遵循遗传距离随地理距离增大而增大的规律。距离较近的NBH和SL两个居群首先聚在一起,ML、MB和HYL地理距离较近,3个居群聚在一起。但是BB居群从地理距离看,与MB和HYL居群很近,但聚类时却和SJ居群聚在一起, 这可能和其自然居群的大小相关,SJ也属于小居群, 小居群趋向于更低的遗传多样性。
物种的遗传结构是决定其对自然或人工选择响应能力的基础,同时是制定该物种合理保护和利用策略的关键[27]。保护物种的遗传结构就是尽可能多地保护遗传多样性。因此,对琴叶风吹楠的保护,不能采用单一的保护措施,而应该采取综合保护策略。首先,应该制止人类对居群内树木的砍伐,特别是接近农耕区的居群;其次,本研究结果表明, ML和NBH居群具有丰富的遗传多样性,且这2个居群的种群数量较大,生境遭人为破坏较少,应该就地保存;在保持原有居群数量的同时,可以人工播种繁殖,以扩大现有居群大小,达到丰富居群内遗传多样性的目的。
| [1] |
Delectis Florae Reipublicae Popularis Sinicae, Agendae Academiae Sinicae Edita. Florae Reipublicae Popularis Sinicae, Tomus 30(2)[M]. Beijing: Science Press, 1979: 194-205. 中国植物志编辑委员会. 中国植物志, 第30卷第2分册[M]. 北京: 科学出版社, 1979: 194-205. |
| [2] |
Yunnan Institute of Botany. Floral of Yunnan, Vol. 1[M]. Beijing: Science Press, 1977: 8-13. 云南省植物研究所. 云南植物志, 第1卷[M]. 北京: 科学出版社, 1977: 8-13. |
| [3] |
WANG S, XIE Y. China Species Red List, Vol. 1 Red List[M]. Beijing: Higher Education Press, 2004: 330. 汪松, 解焱. 中国物种红色名录, 第1卷红色名录[M]. 北京: 高等教育出版社, 2004: 330. |
| [4] |
WU Y, DUAN A A, TIAN Y H, et al. Investigation of wild resource of a rare oil tree Horsfieldia pandurifolia in Yunnan[J]. Guihaia, 2011, 31(2): 217-221, 216. 吴裕, 段安安, 田耀华, 等. 云南野生珍稀油料树种琴叶风吹楠资源调查[J]. 广西植物, 2011, 31(2): 217-221, 216. DOI:10.3969/j.issn.1000-3142.2011.03.015 |
| [5] |
JOSHI S P, GUPTA V S, AGGARWAL P K, et al. Genetic diversity and phylogenetic relationship as revealed by inter simple sequence repeat (ISSR) polymorphism in the genus Oryza[J]. Theor Appl Genet, 2000, 100(8): 1311-1320. DOI:10.1007/s001220051440 |
| [6] |
WANG Y, TANG S Q, LI X K. The genetic diversity of the endangered plant Abies yuanbaoshanensis[J]. Biodiv Sci, 2004, 12(2): 269-273. 王燕, 唐绍清, 李先琨. 濒危植物元宝山冷杉的遗传多样性研究[J]. 生物多样性, 2004, 12(2): 269-273. DOI:10.3321/j.issn:1005-0094.2004.02.007 |
| [7] |
LI X L. Genetic diversity and endangered mechanism of Camellia azalea Wei[D]. Beijing: Chinese Academy of Forestry, 2012: 59-73. 李辛雷.杜鹃红山茶遗传多样性及其濒危机制[D].北京: 中国林业科学研究院, 2012: 59-73. |
| [8] |
CI X Q. Studies on conservation genetics of endangered species Litsea szemaois (Lauraceae)[D]. Beijing: Graduate University of Chinese Academy of Sciences, 2007: 1-56. 慈秀芹.樟科濒危植物思茅木姜子(Litsea szemaois)的保护遗传学研究[D].北京: 中国科学院研究生院, 2007: 1-56. |
| [9] |
GUAN B C. Conservation genetics and molecular phylogeography of Dysosma versipellis (Berberidaceae), an endemic and endangered plant from China[D]. Hangzhou: Zhejiang University, 2008: 70-91. 管毕财.特有濒危植物八角莲保护遗传学和分子亲缘地理学[D].杭州: 浙江大学, 2008: 70-91. |
| [10] |
PANICZ R, NAPORA-RUTKOWSKI Ł, KESZKA S, et al. Genetic diversity in natural populations of noble crayfish (Astacus astacus L.) in north-western Poland on the basis of combined SSR and AFLP data[J]. PeerJ, 2019, 7: e7310. DOI:10.7717/peerj.7301 |
| [11] |
LI X L, WANG J, YIN H F, et al. Analysis on genetic diversity and genetic structure of Camellia nitidissima by AFLP technology[J]. J Ecol Rural Environ, 2019, 35(1): 63-68. 李辛雷, 王洁, 殷恒福, 等. 金花茶遗传多样性和遗传结构AFLP分析[J]. 生态与农村环境学报, 2019, 35(1): 63-68. DOI:10.19741/j.issn.1673-4831.2018.0236 |
| [12] |
LI X Y, LIANG D J, JI C Y, et al. AFLP analysis of Populus alba, P. maximowiczii and their hybrid offsprings[J]. J Liaoning For Sci Technol, 2019(4): 1-4, 74. 李晓宇, 梁德军, 纪纯阳, 等. 银白杨×马氏杨杂交子代及亲本的AFLP分析[J]. 辽宁林业科技, 2019(4): 1-4, 74. |
| [13] |
CHEN Z H, SHAN R Y, YOU X M, et al. Constructing fingerprints and analyzing genetic diversity of 43 tea cultivars in Fujian province[J]. J Trop Subtrop Bot, 2017, 25(6): 579-586. 陈志辉, 单睿阳, 游小妹, 等. 43个福建省茶树品种指纹图谱构建及遗传多样性分析[J]. 热带亚热带植物学报, 2017, 25(6): 579-586. DOI:10.11926/jtsb.3743 |
| [14] |
LI Y T, DONG Y F, WANG Z M, et al. Genetic diversity analysis of Catalpa bungee germplasm with AFLP markers[J]. J CS Univ For Technol, 2019, 39(7): 37-43. 李永涛, 董玉峰, 王振猛, 等. 楸树优良种质资源的AFLP遗传多样性分析[J]. 中南林业科技大学学报, 2019, 39(7): 37-43. DOI:10.14067/j.cnki.1673-923x.2019.07.005 |
| [15] |
YAN S Y, ZHU P, GONG W, et al. Studies on genetic diversity of Juglans cultivar germplasms in Sichuan based on RAD-SNPs analysis[J]. J Trop Subtrop Bot, 2019, 27(1): 19-28. 闫思宇, 朱鹏, 龚伟, 等. 基于RAD-SNPs分析的四川核桃良种资源的遗传多样性研究[J]. 热带亚热带植物学报, 2019, 27(1): 19-28. DOI:10.11926/jtsb.3906 |
| [16] |
WU Y, XU Y L, TIAN Y H, et al. Morphological variations of fruits and seeds in rare oil tree Horsfieldia pandurifolia Hu[J]. Bull Bot Res, 2011, 31(1): 4-8. 吴裕, 许玉兰, 田耀华, 等. 珍稀油料树种琴叶风吹楠果实和种子形态变异[J]. 植物研究, 2011, 31(1): 4-8. DOI:10.7525/j.issn.1673-5102.2011.01.002 |
| [17] |
XU Y L, WU Y, ZHANG K Y, et al. Oil content and fatty acid components in seeds of Horsfieldia pandurifolia (a rare oil tree species) in Yunnan[J]. Guizhou Agric Sci, 2010, 38(7): 163-166. 许玉兰, 吴裕, 张夸云, 等. 珍稀油料树种琴叶风吹楠种子含油量及脂肪酸成分分析[J]. 贵州农业科学, 2010, 38(7): 163-166. DOI:10.3969/j.issn.1001-3601.2010.07.051 |
| [18] |
MAO C L, ZHANG F L, LI X Q, et al. The complete chloroplast genome sequence of Horsfieldia pandurifolia (Myristicaceae)[J]. Mitochond DNA Part B Resour, 2019, 4(1): 949-950. DOI:10.1080/23802359.2018.1561224 |
| [19] |
NEI M. Molecular Population Genetics and Evolution[M]. Amsterdam, New York: North Holland, 1975: 13-17. DOI:10.1111/j.1558-5646.1976.tb00902.x
|
| [20] |
YEH F C, YANG R C, BOYLE T B J, et al. POPGENE, the User Friendly Shareware for Population Genetic Analysis[M]. Edmonton: University of Alberta, Canada, 1997: 1-10.
|
| [21] |
ROHLF F J. NTSYS-pc:Numerical Taxonomy and Multi-variate Analysis System[M]. New York: Exeter Software, Setauker, 1992: 1-11.
|
| [22] |
LIU Z L, ZHAO G F. Population genetics and its implications for conservation of rare and endangered plants[J]. Chin Biodiv, 1999, 7(4): 340-346. 刘占林, 赵桂仿. 居群遗传学原理及其在珍稀濒危植物保护中的应用[J]. 生物多样性, 1999, 7(4): 340-346. DOI:10.3321/j.issn:1005-0094.1999.04.014 |
| [23] |
WHITLOCK M C, MCCAULEY D E. Indirect measures of gene flow and migration:FST≠1/(4Nm+1)[J]. Heredity, 1999, 82(2): 117-125. DOI:10.1038/sj.hdy.6884960 |
| [24] |
THOMAS L, THOMAS G, DENIS B, et al. Evaluating gene flow using selected markers:A case study[J]. Genetics, 1998, 149(3): 1383-1392. |
| [25] |
MAO C L, WU Y, ZHANG F L, et al. Genetic diversity of Scleropyrum wallichianum based on AFLP markers[J]. J Trop Subtrop Bot, 2019, 27(1): 29-35. DOI:10.11926/jtsb.3908 |
| [26] |
HAMRICK J L, GODT M J W. Allozyme diversity in plant species[M]//BROWN A H D, CLEGG M T, KAHLER A L, et al. Plant Population Genetics. Breeding, and Genetic Resources. Sunderland: Sinauer, 1990: 43-63.
|
| [27] |
REIS A M M, GRATTAPAGLIA D. RAPD variation in a germplasm collection of Myracrodruon urundeuva (Anacardiaceae), an endangered tropical tree:Recommendations for conservation[J]. Genet Resour Crop Evol, 2004, 51(5): 529-538. DOI:10.1023/B:GRES.0000024159.48644.e6 |
 2020, Vol. 28
2020, Vol. 28



