2. 中国科学院大学, 北京 100049;
3. 鼎湖山国家级自然保护区管理局, 广东 肇庆 526070;
4. 广东省应用植物学重点实验室, 广州 510650
2. University of Chinese Academy of Sciences, Beijing 100049, China;
3. Administration of Dinghushan National Nature Reserve, South China Botanical Garden, Chinese Academy of Sciences, Zhaoqing 526070, Guangdong, China;
4. Guangdong Provincial Key Laboratory of Applied Botany, Guangzhou 510650, China
种群是生态学研究各个层次中最重要的一个层次, 它是群落结构和功能的最基本单位, 也是物种适应的单位。环境条件显著地影响着种群内个体的表现特征,同时起到环境筛的效果。因此深入研究种群内个体与物理环境相互作用、以及调节机理是了解群落和生态系统的结构、功能、动态和调控的基础[1–2]。植物由于固着生长的特性,发展出以表型可塑性为主的反应策略应对环境变化,可塑性极大的扩展了植物物种的生态幅和分布范围,使种群中个体的分布具有很大的随机性[3], 同时由于不同基因型对种群的贡献,从而使得植物种群的分布特征变得非常复杂。因此,要真正了解植物种群分布格局的形成和调节机制以及其适应性进化的动态规律,必须选择能综合反映遗传基础和可塑性调节的信息指标,才能详实的反映种群特征。
生物的表型和生理特征对环境的响应(植物表现为表型、生理等方面的可塑性)来源于其对环境刺激信号敏感DNA位点的表达调控。目前已有充分的研究表明可塑性特征受DNA二级结构所影响, 并显著影响所在DNA区域的遗传变异速率[4–5]。DNA的甲基化作为一种重要的表观遗传修饰,广泛存在于动植物中,并在维持基因组稳定性[6]、调节基因表达[7]等方面起着重要作用。植物基因组中, 大约有超过30%的胞嘧啶发生甲基化,同一植物的DNA甲基化特征存在显著的时空差异[8]。相比于常用的以生理代谢、形态结构、个别基因功能等指标对物种的适应机制进行研究的方法[9],DNA甲基化变异分析,如甲基敏感扩增片段多态性(methylation sensitive amplified polymorphism, MSAP)不但敏感性强, 而且由于对试验条件要求不苛刻且蕴含的信息全面,非常适合于多个体的群体行为研究[10]。为弥补目前对DNA甲基化在小尺度,特别是种群内土壤环境反应机制研究的不足,我们以鼎湖山20 hm2大样地锥(Castanopsis chinensis)种群为材料, 在充分了解其遗传基础的情况下[11],对其DNA甲基化进行分析,探讨影响种群表观遗传变异的因素,为研究植物表型遗传变异对微生境的适应机制提供科学依据。
1 材料和方法 1.1 采样地概况鼎湖山20 hm2大样地位于广东省肇庆市鼎湖山国家级自然保护区内,属于南亚热带常绿阔叶林(以下简称DHS),样地海拔230~470 m,坡度30°~ 50°。土壤以酸性赤红壤为主,海拔300 m以上的局部分布着山地黄壤。样地于2005年参照CTFS (Centre for Tropical Forest Science)森林动态样地建设技术规范建立,位于保护区核心区,东西跨度400 m, 南北跨度500 m,划分成500个20 m×20 m的小样方[12]。该样地依据史料记载可以明显划分为2个不同演替阶段的群落,成熟林在1955年遭受过严重的砍伐破坏,林分的演替年龄在60 a左右, 处于群落演替的中后期;过熟林自明朝以来属于庆云寺的风水林,植被保护完好,至少有400 a的保护历史,属于群落演替的顶级阶段(图 1)[13]。

|
图 1 鼎湖山大样地取样分布图 Fig. 1 Sample individual distribution of large sample plot in Dinghushan |
在鼎湖山大样地以20 m×20 m的小样方为单位, 每个样方内随机选择1株锥(Castanopsis chinensis), 采集新鲜、无病虫害、成熟度一致的叶片,液氮保存, 带回实验室提取DNA,共采集381株,308株来自成熟林,73株来自过熟林。2个演替阶段林分在土壤理化因子和地形因子上存在显著差异(表 1)。锥所在小样方的土壤理化性质、地形因子来源于2005年样方建立时测量的数据,并经过2015年的土样分析校正[14]。
| 表 1 成熟林和过熟林土壤理化性质和地形概况 Table 1 Soil physiochemical characters and topographic status of mature forest and over-mature forest |
用改良的经典CTAB法来提取锥基因组DNA。MSAP是在AFLP (扩增片段长度多态性, amplified fragment length polymorphism)的基础上用对甲基化敏感性不同的两种限制性内切酶Hap ⅡMsp Ⅰ代替Mse Ⅰ,与对甲基化不敏感的内切酶EcoR I对基因组DNA进行双酶切;再将酶切产物连接接头, 设计筛选引物进行预扩增和选择性扩增,在选择性扩增阶段使用荧光引物,经由毛细管电泳得到结果。锥的F-MSAP扩增体系参考刘孟等[15]的优化体系。
1.4 数据处理为保证选取片段的质量,每对引物在100~500 bp长度内选择25~30个片段,各对引物的位点数分别为E3-H/M2:26、E5-H/M2:34、E6-H/M1:26、E8-H/ M1:27、E8-H/M5:29、E9-H/M2:34,共176个片段。用R语言包“msap”分析MSAP矩阵,分析6对引物的扩增片段多态性,依据对EcoRI-HpaⅡ和EcoRI- MspⅠ酶切后的表征片段有无的0, 1数据表所构成的二进制数据矩阵,进一步统计甲基化敏感位点(MSL)和不敏感位点(NML)的数量、多态率、香浓多样性指数,并对两种位点的香农多样性指数进行Wilcoxon秩检验,比较表观遗传多样性的差异。结合选择性扩增条带的特征,将MSAP矩阵按照DNA甲基化模式进行分类,检测到4种条带类型(表 2),其中类型Ⅳ不在本研究的范围,只研究未甲基化、全甲基化、半甲基化3种模式[16]。甲基化率的计算公式分别为:半甲基化率(%)=Ⅱ型/(Ⅰ型+Ⅱ型+Ⅲ型);基化率(%)=Ⅲ型/(Ⅰ型+Ⅱ型+Ⅲ型);总的甲基化率(%)=(Ⅱ型+Ⅲ型)/(Ⅰ型+Ⅱ型+Ⅲ型)。统计3种类型扩增条带的数量,计算锥种群不同模式、不同林分的甲基化水平。利用GenALEX软件进行5种生境的ANOVA分析, 并进行显著性检验。使用距离矩阵用Mega 6进行UPGMA聚类分析。所有显著性分析时先检验样本的正态性,当样本都服从正态分布时,运用独立样本t检验,样本不服从正态分布时,运用非参数Mann-Whitney U检验,显著性水平以P < 0.05为标准。
| 表 2 不同林分锥的MSAP条带类型和甲基化水平 Table 2 Methylation levels of Castanopsis chinensis in different stand |
为了量化环境因子对甲基化水平变异的解释, 用方差分解的方法分析土壤和地形因子及其交互作用对锥甲基化率的解释。个体的甲基化率作为反应变量,锥所在样方的土壤含水率、容重、pH、有机质含量、总氮、速效氮、总磷、有效磷、总钾和有效钾作为土壤变量,运算时用正态转换过的数据;样本所在样方的凹凸度、坡度、朝向、海拔的一到三阶函数作为地形变量[14];方差分解前先用“packfor”程序包[17]对各组变量进行前向选择,筛选出与甲基化率显著相关的变量,再将显著变量按土壤、地形、两类进行方差分解,使用R语言的“vegan”程序包[18]。
2 结果和分析 2.1 甲基化水平表 2为整个样地和不同林分锥个体的甲基化水平以及3种甲基化类型的条带数。锥种群中Ⅰ型条带最多,约为80%,表示大部分片段未发生甲基化现象, Ⅱ型(7.46%~8.46%)略低于Ⅲ型(11.30%~12.42%), 发生全甲基化的片段比例高于半甲基化。鼎湖山锥的总体甲基化率为19.97%,全甲基化率为11.60%, 半甲基化率为8.36%。经Kolmogorov-Smirnov检验,大部分环境因子和甲基化水平不服从正态分布,经Mann-Whitney U检验,两林分间的总甲基化率、全甲基化率、半甲基化率均无显著差异(P > 0.05)。成熟林锥个体的平均总甲基化率(19.94%)比过熟林(20.06%)略低;成熟林的平均全甲基化率(11.38%)比过熟林(12.52%)低,平均半甲基化率(8.56%)比过熟林(7.54%)高,但两林分间的甲基化水平差异不显著。
2.2 相关性分析Spearman秩相关分析表明,很多环境因子对甲基化率产生影响(表 3)。整体而言,土壤的相对含水量(RSW)、含氧量(TO)、全氮(TN)、全磷(TP)和地形的凹凸度和海拔与锥的甲基化水平呈负相关关系; 土壤的容重(VW)、酸碱度(pH)、速效钾(AK)、全钾(TK)与锥的甲基化水平呈正相关关系,即土壤养分和光照越丰富,锥的甲基化水平越低。具体来讲RSW显著负向影响成熟林锥的全甲基化水平和显著正向影响过熟林锥的半甲基化水平,TN对两林分的总甲基化率、成熟林的全甲基化率都有负面的显著性影响,TO对过熟林锥的总甲基化率、全甲基化率和TP对过熟林锥的半甲基化率都呈显著负相关。VW和pH对整体的甲基化率无显著影响,但在过熟林中与总甲基化率、全甲基化率呈显著正相关。
| 表 3 土壤因子与甲基化率的相关性 Table 3 Relation between soil factors and methylation rate |
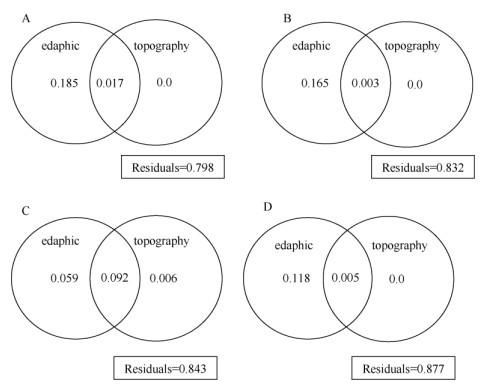
方差分解结果表明, 土壤因子解释了成熟林中20.2%的全甲基化率和16.8%的半甲基化率变异(图 2: A, B)。而在过熟林中土壤因子解释率降低,对全甲基化率和半甲基化率变异的解释分别为15.1%和12.3% (图 2: C, D)。与之相反,地形因子在两个林分之间对全甲基化率变异的解释率由成熟林的1.7%上升为过熟林的9.8%。同时方差分解中“packfor”程序包筛选出显著影响甲基化率的环境因子,成熟林中TN、pH、TO、TP、AK、AN、RSW和VW均对甲基化有显著影响,过熟林中则是TK起主要作用,而AP等微生境因子作用不明显。
|
图 2 锥成熟林和过熟林的甲基化率变异方差分解图。数字表示显著解释率,残差表示未解释部分。A:成熟林甲基化率; B:成熟林半甲基化率; C:过熟林全甲基化率; D:过熟林半甲基化率。 Fig. 2 Variance decomposition map of methylation rate variation in mature and over mature forest of Castanopsis chinensis. The number means significant interpretation rate, and the residual means unexplained part. A: Methylation rate in mature forest; B: Hemi methylation rate in mature forest; C: Methylation rate in over-mature forest; D: Hemi methylation rate in over-mature forest. |
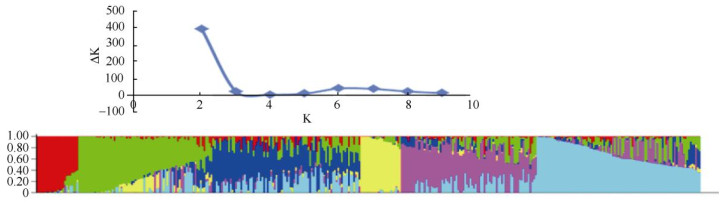
基于MSAP数据,通过Structure软件分析,从K与ΔK的关系图(图 3)可见,当K为6时,ΔK出现峰值,因此377份鼎湖山锥样本可划分为6个亚群体,但除红色和黄色区域外,其余4个区域都表现出高的混杂现象。
|
图 3 Structure软件对377鼎湖山锥样本的聚类分析 Fig. 3 Clustering results of 377 samples in Castanopsis chinensis by structure software |
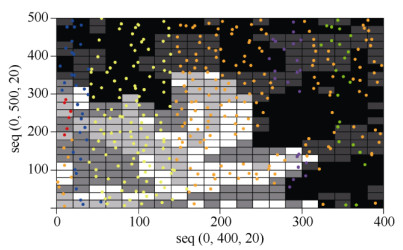
将MSP聚类结果标注到鼎湖山大样地的分布图上(图 4)。背景颜色由浅到深分别代表5种不同的生境,即高山、山脊、高谷、低山、低谷。不同颜色的圆点代表377个样本聚成的不同类群。可以看出,锥种群的表观遗传特征聚类结果与地形特征并不重合。
|
图 4 鼎湖山锥377个样本的甲基化敏感聚类图 Fig. 4 MSP clusters of 377 samples of Castanopsis chinensis in Dinghu Mountain |
从物种的遗传结构入手是长期以来种群生态学研究的一个重要方面[9, 15]。但常用的遗传标记得出的种群结构虽然能显示物种群体之间以及群体内个体间亲缘关系的远近,但并不是物种与环境条件相互作用的直接体现,并且研究结果无法与生理生态学结果直接相衔接。因此一直以来遗传多样性分析偏重于物种扩散过程的研究[16, 19], 而物种个体扩散仅是其实现定居的前期步骤,个体最终定居形成特定分布格局并进一步发生适应性进化与生境选择适应、个体互作密切相关[20–22]。
本研究中随着群落的演替,影响DNA甲基化水平的土壤因子发生了变化。成熟林的土壤含水量、土壤容重、pH值显著高于过熟林,过熟林土壤的总有机质、总钾、有效钾、总磷、总氮、有效氮都显著高于成熟林(P < 0.05),表明随着群落的演替有机质不断积累,土壤营养物质含量增加。这是因为土壤有机质能促进土壤微生物的分解和矿化作用,引起过熟林土壤中的无机营养物质N、P、K等含量的增加;氮沉降造成过熟林的AN和TN都高于成熟林;土壤有机质的增加使土壤变得更疏松透气,从而过熟林的土壤容重和土壤含水率更低, 这与Chen等[14]的结果一致。过熟林pH值更低, 说明随着群落的演替,土壤酸性增强。过熟林N含量高而P含量低于成熟林,氮磷比高于成熟林,说明随着群落发育,P限制进一步加剧,这与酸性土壤受到P限制的研究结果一致。因此所有的这些在成熟林和过熟林之间土壤特征的差异变化符合亚热带常绿阔叶林森林群落土壤演替的一般规律[14]。
对应于锥所在两个演替阶段森林土壤营养条件的不同,几种重要环境因子在两个林分中与锥的DNA甲基化水平相关的显著性不同。RSW与DNA甲基化率的相关性在成熟林中更明显,这可能是因为RSW是群落演替的驱动因子,土壤中的水分与有机元素是植物生长发育的必要条件。充足的土壤水分和养分可以促进锥的生长,降低甲基化率。而过熟林土壤因为TN、TO等有机质积累比过熟林多,有机质越多,pH也越低,土壤越疏松透气,土壤容重越低,越适合植物生长,DNA甲基化率越低。综合分析锥DNA甲基化水平与环境因子的关系可以看出,首先环境因子中相对含量更高的林分,对锥的DNA甲基化水平呈正或负向的影响更显著, 如RSW更高的成熟林中,RSW对DNA甲基化率的影响更明显;TO、TP、VW、pH、凹凸度更高的过熟林中,DNA甲基化率对TO、TP、VW、pH和凹凸度的响应更显著,这证明了甲基化修饰对环境变化的敏感适应性响应。
在两林分中土壤对植株甲基化生理反应变异的贡献差异传递了非常有意义的生态学含义。首先从土壤整体的营养条件对DNA甲基化的影响上看,虽然在半甲基化率变异的贡献上两个样地土壤的贡献率差异不大,但过熟林中土壤对全甲基化率变异的贡献要显著小于成熟林(15.1% < 20.2%),尤其是土壤本身的贡献由成熟林的18.5%降为过熟林的5.9%。这与亚热带常绿阔叶林演替过程中土壤养分增加的规律相对应,由于过熟林中土壤营养限制的减弱和土壤的均一化,致使土壤所引起的表观遗传变异减小。
其次也是更为有意义的是,具体土壤元素对DNA甲基化率变异的影响。在过熟林和成熟林中对甲基化率变异起显著作用的具体土壤元素差异显著,成熟林中几乎所有的元素都对全甲基化率变异有贡献,磷和氮对半甲基化率有影响,而在过熟林中仅钾对全甲基化率有影响,没有土壤元素对半甲基化率有影响。这为我们重新定义不同植物群落中养分限制提供了新的思路。以磷为例,理论预测磷限制应普遍存在于强风化古老土壤,但试验尚未在物种丰富的低地热带森林中检测到对磷添加的一致反应,最新的研究表明,磷限制普遍存在于物种个体水平,未在群落水平得到响应[23]。在本研究中土壤有效磷的变化遵循成土序列上原生演替的经典理论假设,即过熟林显著低于成熟林,但相反土壤有效磷的限制作用却仅发生在成熟林个体上,这表明影响植株对土壤有效磷的需求除了土壤元素本身的浓度限制外,尚有其它重要的限制条件存在,本研究中除了所在微环境有效磷的显著差异, 还存在过熟林中群落内植株密度以及锥的茎级均高于成熟林的差异[14]。由于植株密度大只会加剧植株间对土壤有效磷的竞争行为,而植株胸径大小所表征的生活史阶段不同以及由此引发的植株营养需求的转变可能成为一个合理的解释,也为我们重新定义植物群落中养分限制提供了新的思路。
同时通过方差分解得到的微生境对植株甲基化率变异贡献的结果也解释了MSP的聚类类型并不与5种生境的分布相重合的原因,即微生境特征和分布格局对种群适应机制和分布格局有显著影响,但仅起部分作用(本研究中最高为由成熟林贡献20.2%全甲基化变异率)。
鼎湖山样地内地形复杂多变,而地形是生境异质性的重要来源,会导致很多基本生态因子,如光照、温度、水分和土壤养分等的空间差异,这种差异即使是在空间很小尺度上也可能导致个体间表观遗传分化。另外通过Structure分析划分的6个亚群体之间表现出高的混杂现象,说明影响表观遗传变异的因素较为复杂,仅凭土壤微环境无法给出完整解释,在今后的研究中应进一步关注空间密度、胸径等因素对DNA甲基化的影响。
| [1] |
SEIDLER T G, PLOTKIN J B. Seed dispersal and spatial pattern in tropical trees[J]. PLoS Biol, 2006, 4(11): 2132-2137. DOI:10.1371/journal.pbio.0040344 |
| [2] |
RAVENTóS J, WIEGAND T, DE LUIS M. Evidence for the spatial segregation hypothesis:A test with nine-year survivorship data in a Mediterranean shrubland[J]. Ecology, 2010, 91(7): 2110-2120. DOI:10.1890/09-0385.1 |
| [3] |
FUSCO G, MINELLI A. Phenotypic plasticity in development and evolution:Facts and concepts[J]. Philos Trans Roy Soc B Biol Sci, 2010, 365(1540): 547-556. DOI:10.1098/rstb.2009.0267 |
| [4] |
JOHANNES F, PORCHER E, TEIXEIRA F K, et al. Assessing the impact of transgenerational epigenetic variation on complex traits[J]. PLoS Genet, 2009, 5(6): e1000530. DOI:10.1371/journal.pgen.1000530 |
| [5] |
PAUN O, BATEMAN R M, FAY M F, et al. Stable epigenetic effects impact adaptation in allopolyploid orchids (Dactylorhiza:Orchidaceae)[J]. Mol Biol Evol, 2010, 27(11): 2465-2473. DOI:10.1093/molbev/msq150 |
| [6] |
MATZKE M A, METTE M F, AUFSATZ W, et al. Host defenses to parasitic sequences and the evolution of epigenetic control mechanisms[J]. Genetica, 1999, 107(1/2/3): 271-287. DOI:10.1023/a:1003921710672 |
| [7] |
CHAN S W L, HENDERSON I R, JACOBSEN S E. Gardening the genome:DNA methylation in Arabidopsis thaliana[J]. Nat Rev Genet, 2005, 6(5): 351-360. DOI:10.1038/nrg1601 |
| [8] |
RAPP R A, WENDEL J F. Epigenetics and plant evolution[J]. New Phytol, 2005, 168(1): 81-89. DOI:10.1111/j.1469-8137.2005.01491.x |
| [9] |
KOSMA D K, BOURDENX B, BERNARD A, et al. The impact of water deficiency on leaf cuticle lipids of Arabidopsis[J]. Plant Physiol, 2009, 151(4): 1918-1929. DOI:10.1104/pp.109.141911 |
| [10] |
XIONG L Z, XU C G, SAGHAI M M A, et al. Patterns of cytosine methylation in an elite rice hybrid and its parental lines, detected by a methylation-sensitive amplification polymorphism technique[J]. Mol Genet Genom, 1999, 261(3): 439-446. DOI:10.1007/s004380050986 |
| [11] |
WANG Z F, LIAN J Y, HUANG G M, et al. Genetic groups in the common plant species Castanopsis chinensis and their associations with topographic habitats[J]. Oikos, 2012, 121(12): 2044-2051. DOI:10.1111/j.1600-0706.2012.20483.x |
| [12] |
YE W H, CAO H L, HUANG Z L, et al. Community structure of a 20 hm2 lower subtropical evergreen broadleaved forest plot in Dinghu-shan, China[J]. J Plant Ecol, 2008, 32(2): 274-286. 叶万辉, 曹洪麟, 黄忠良, 等. 鼎湖山南亚热带常绿阔叶林20公顷样地群落特征研究[J]. 植物生态学报, 2008, 32(2): 274-286. DOI:10.3773/j.issn.1005-264x.2008.02.005 |
| [13] |
LIAN J Y, CHEN C, HUANG Z L, et al. Community composition and stand age in a subtropical forest, southern China[J]. Biodiv Sci, 2015, 23(2): 174-182. 练琚愉, 陈灿, 黄忠良, 等. 鼎湖山南亚热带常绿阔叶林不同成熟度群落特征的比较[J]. 生物多样性, 2015, 23(2): 174-182. DOI:10.17520/biods.2014243 |
| [14] |
CHEN J, RUI Y C, ZHOU X, et al. Determinants of the biodiversity patterns of ammonia-oxidizing archaea community in two contrasting forest stands[J]. J Soils Sed, 2016, 16(3): 878-888. DOI:10.1007/s11368-015-1302-4 |
| [15] |
LIU M, OUYANG X J, CHEN J, et al. F-MSAP sampling strategy and genetic diversity analysis of Castanopsis chinensis in South China Botanical Garden[J]. Guihaia, 2017, 37(1): 15-21, 8. 刘孟, 欧阳学军, 陈洁, 等. 华南植物园锥栗F-MSAP采样策略及遗传多样性分析[J]. 广西植物, 2017, 37(1): 15-21, 8. DOI:10.11931/guihaia.gxzw201603024 |
| [16] |
SALMON A, AINOUCHE M L, WENDEL J F. Genetic and epigenetic(Poaceae)consequences of recent hybridization and polyploidy in Spartina[J]. Mol Ecol, 2005, 14(4): 1163-1175. DOI:10.1111/j.1365-294X.2005.02488.x |
| [17] |
BLANCHET F G, LEGENDRE P, BORCARD D. Forward selection of explanatory variables[J]. Ecology, 2008, 89(9): 2623-2632. DOI:10.1890/07-0986.1 |
| [18] |
GILBERT B, BENNETT J R. Partitioning variation in ecological communities:Do the numbers add up?[J]. J Appl Ecol, 2010, 47(5): 1071-1082. DOI:10.1111/j.1365-2664.2010.01861.x |
| [19] |
LUNDEMO S, STENØIEN H K, SAVOLAINEN O. Investigating the effects of topography and clonality on genetic structuring within a large Norwegian population of Arabidopsis lyrata[J]. Ann Bot, 2010, 106(2): 243-254. DOI:10.1093/aob/mcq102 |
| [20] |
HARDESTY B D, HUBBELL S P, BERMINGHAM E. Genetic evidence of frequent long-distance recruitment in a vertebrate-dispersed tree[J]. Ecol Lett, 2006, 9(5): 516-525. DOI:10.1111/j.1461-0248.2006.00897.x |
| [21] |
JACQUEMYN H, WIEGAND T, VANDEPITTE K, et al. Multigene-rational analysis of spatial structure in the terrestrial, food-deceptive orchid Orchis mascula[J]. J Ecol, 2009, 97(1): 206-216. DOI:10.1111/j.1365-2745.2008.01464.x |
| [22] |
COMITA L S, CONDIT R, HUBBELL S P. Developmental changes in habitat associations of tropical trees[J]. J Ecol, 2007, 95(3): 482-492. DOI:10.1111/j.1365-2745.2007.01229.x |
| [23] |
TURNER B L, BRENES-ARGUEDAS T, CONDIT R. Pervasive phosphorus limitation of tree species but not communities in tropical forests[J]. Nature, 2018, 555(7696): 367-370. DOI:10.1038/nature25789 |
 2020, Vol. 28
2020, Vol. 28



