2. 中国科学院大学, 北京 100049;
3. 海南大学热带农林学院, 海口 570228
2. University of Chinese Academy of Sciences, Beijing 100049, China;
3. Institute of Tropical Agriculture and Forestry, Hainan University, Haikou 570228, China
厚藤(Ipomoea pes-caprae)是广泛分布于热带亚热带滨海地区的多年生藤本盐生植物,隶属于旋花科(Convolvulaceae)番薯属[1]。厚藤抗逆性强,对营养物质和水分的利用效率极高,具有良好的固沙能力和耐海水冲刷能力,在岛礁防风固沙、绿化及生态重建等方面发挥重要作用[2]。厚藤作为优异的野生植物资源,具有较大的开发潜力及应用价值,同时也可作为一种研究植物抗逆分子机理、发掘抗逆基因的材料进行深入研究。
脱水素(dehydrin, DHN)是植物胚胎发育晚期丰富蛋白(late embryogenesis abundant proteins, LEA)家族成员,该家族蛋白通常富含较高比例的亲水性氨基酸,是一类高度稳定的亲水性蛋白[3]。研究表明,LEA基因(包括脱水素基因)通常在植物组织遭遇缺水胁迫时广泛诱导表达,在种子发育晚期的脱水过程中,以及植物组织遭受高盐、干旱、冷冻基因ABA处理时表达上调[4]。脱水素可归于LEA_Ⅱ类蛋白[5],目前已经鉴定的植物脱水素蛋白的分子量为9~200 kDa[6-7],且通常含有1~3种具有不同重复的保守结构域。根据各个保守序列在蛋白中的分布数目,脱水素可分为Kn、SKn、KnS、YnKn和YnSKn (n表示重复数)[8]5个亚组。
有研究表明,当植物遭受能导致细胞脱水的相关逆境时,如高渗透压或高盐胁迫以及冷冻时,脱水素基因都能够在细胞内大量表达[9]。脱水素基因针对逆境胁迫的转录调控是该类基因行使其正常的生物学功能的第一步,而基因的转录主要决定于其启动子序列。胁迫诱导的基因启动子通常含有多个/种胁迫调控的顺式作用元件,包括ABA应答元件ABRE (ABA-responsive elements),C-重复/干旱应答/低温应答元件CRT/DRE/LTRE (C-repeat/ drought-responsive/low-temperature-responsive element), 转录因子MYB或MYC结合元件MYBPE/MYCPE (myeloblastosis/myelocytomatosispromoter elements)[8]。如黄瓜(Cucumis sativus)[9]、葡萄(Vitis vinifern)[10-11]、小麦(Triticum aestivum)[12]、大麦(Hordeum vulgare)[13]的脱水素基因启动子中含有多个不同逆境胁迫应答元件,并且这些脱水素基因的表达均受到相应的胁迫环境的调控。综上所述,基因的转录调控受启动子区域的顺式作用元件的影响,而鉴定具有特定逆境胁迫应答元件的启动子在逆境胁迫的植物遗传改良中具有重要的应用潜力。
本实验室在前期研究中已经克隆了厚藤脱水素的cDNA序列,并根据其cDNA序列发掘出该基因对应的基因组序列。实时定量RT-PCR表明IpDHN是一个受胁迫诱导的耐盐耐旱功能基因[14]。据此, 本试验拟通过染色体步移法克隆厚藤IpDHN基因的启动子序列,分析启动子中的顺式作用元件,进一步构建IpDHN-Pro/pBI101.2植物表达载体,并转化拟南芥植株,探讨启动子的表达模式,为启动子序列应用于针对高盐/干旱胁迫的植物转基因育种和培养高盐/干旱抗逆作物提供科学依据。
1 材料和方法 1.1 材料厚藤(Ipomoea pes-caprae)的成年植株取材于中国科学院华南植物园温室群(23°18′75.91″ N, 113° 37′02.38″ E)。拟南芥(Arabidopsis thaliana)生态型为Col-0,栽培温度25℃,相对湿度70%,光照强度为120~150 μmol m–2s–1,光暗周期为16 h/8 h。大肠杆菌(Escherichia coli)菌株DH5α,植物表达载体pBI101.2和农杆菌株GV3101均为本实验室保存。载体构建所需要的克隆载体pGEM T购于Promega (上海)公司,基因侧翼序列扩增所用的试剂盒为Genome Walking Kit (TaKaRa Bio USA)。载体无缝克隆所用的In-Fusion HD Cloning Kit购于Clontech (TaKaRa Bio USA)公司。总RNA的提取依照HiPure Plant RNA Kits (Magen)的说明书进行。cDNA链的合成依照TransScript One-Step gDNA Removal and cDNA Synthesis SuperMix (全式金公司)进行。qRT- PCR反应参照TransStart® Top Green qPCR Super- Mix (全式金公司)进行。其他试剂及仪器均为分子生物学实验室常用试剂及设备。
1.2 方法基因组DNA的提取 取健康生长的厚藤小苗叶片0.1 g,放入研钵中加入液氮研磨至粉末,采用北京天恩泽基因科技有限公司的植物基因组DNA提取试剂盒One-Tube Plant DNAOUT (货号: 60705)提取基因组DNA。采用电泳检测和紫外分光光度计的方法检测基因组DNA的纯度和浓度,并用ddH2O将DNA的浓度调整至100 ng μL–1。
IpDHN基因组序列的获取 根据厚藤IpDHN的cDNA阅读框序列,设计引物对DHNF和DHNR (5′-ATGGCGGAGGAGTGCCACC-3′和5′-TTAATG- GCATTCCCCACCCTT-3′),以厚藤基因组DNA为模板,扩增IpDHN的基因组序列。PCR反应结束后,用1%的琼脂糖凝胶电泳检测,从PCR产物中挑选明亮条带,依照Magen公司HiPure Gel Pure DNA Kits说明书进行琼脂糖凝胶电泳回收,并连接于T载体上。将反应产物转化大肠杆菌DH5α感受态菌株。挑取单克隆,提取质粒,送生物公司进行测序。
IpDHN基因5′侧翼序列的克隆 根据IpDHN基因的基因组序列,参照Genome Walking Kit的操作指南,设计两个特异性引物SP1: 5′-TCCTTATA- CTCCACCCCATG-3′和SP2: 5′-GCAGTGGGGCTT- CTTCTCCT-3′。以厚藤基因组DNA为模板,用SP1、SP2对应染色体步移的第1、2轮随机引物进行染色体步移克隆IpDHN基因5′侧翼序列。两轮PCR结束后,用1%的琼脂糖凝胶电泳检测,从PCR产物中挑选明亮条带,依照Magen公司HiPure Gel Pure DNA Kits说明书进行琼脂糖凝胶电泳回收,并连接于T载体上。将反应产物转化大肠杆菌DH5α感受态菌株。挑取单克隆,提取质粒,送生物公司进行测序, 并保存正确质粒(命名为IpDHN-Pro-pGEM T)备用。
生物信息学分析 采用启动子分析数据库PlantCARE (http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)和植物顺式作用元件分析数据库PLACE (https://www.dna.affrc.go.jp/PLACE/?action=newplace)对IpDHN基因的5′侧翼序列进行启动子功能预测及顺式作用元件进行分析。
IpDHN-Pro::GUS转基因拟南芥的获取 以厚藤基因组DNA为模板,设计引物IpDHNProF: 5′- CGACTCTAGAGGATCCCAGTGGGGCTTCTTCT- CCT-3′和IpDHNProR: 5′-ACCTACCCGGGGATCC- GCTCCTCCGCAGGCTTCTG-3′对厚藤脱水素基因IpDHN的启动子序列进行PCR扩增(下划线表示BamHI酶切位点)。PCR产物进行琼脂糖凝胶电泳回收;同时采用BamHI单酶切处理植物表达载体pBI101.2,回收线性化质粒。回收后IpDHN- PRO启动子PCR片段和线性化pBI101.2质粒经Nanodrop公司紫外分光光度计测定浓度,采用无缝克隆技术进行DNA片段和载体的同源重组连接。按照说明书的方法将反应产物转化大肠杆菌DH5α感受态菌株。挑取单克隆,提取质粒,经测序鉴定为正确的阳性克隆后,命名为IpDHN-Pro/pBI101.2, 保存质粒备用。经测序分析正确后,IpDHN-Pro/ pBI101.2重组质粒采用冻融法转入农杆菌GV3101中。以野生型拟南芥作为转基因材料,采用花序侵染的方法,通过GV3101农杆菌介导转化拟南芥。将所获得的拟南芥转基因T3代种子放于含有50 μg mL–1卡那霉素的MS培养基上进行筛选。获取T3代纯合体拟南芥进行后续分析。
IpDHN-Pro的转录活性验证 取生长10 d的转基因拟南芥T3代植株和开花拟南芥的不同部位, 置于GUS染色反应液中处理3 h以上,采用组织化学法进行染色检测,之后用95%的乙醇退色48 h后利用LEICA DM2500体式显微镜拍照。配制1 mL的GUS染液可以按照以下操作进行,即取1.5 mL的空离心管,首先加入309 μL的ddH2O,再依次加入500 μL 0.2 mol L–1的磷酸钠缓冲液(pH 7.0),1 μL的Triton X- 100,10 μL的50 mmol L–1亚铁氰化钾溶液,10 μL的50 mmol L–1铁氰化钾溶液,20 μL 0.2 mol L–1的EDTA- Na2溶液(pH 7.0),100 μL的甲醇,以及50 μL 10 mg mL–1 X-Gluc溶液(溶于二甲基甲酰胺),并充分混匀。
IpDHN-Pro对胁迫和激素信号的响应 取生长10 d的转基因拟南芥T3代植株(MS培养基平板上),分别加入200 mmol L–1 NaCl、300 mmol L–1甘露醇和0.1 mmol L–1 ABA进行高盐、渗透压和激素处理24 h,以ddH2O处理的幼苗为对照,取幼苗进行GUS组织化学染色并拍照。收集处理的拟南芥小苗各0.5 g提取总RNA,采用两步法以总RNA为模板进行逆转录cDNA。根据罗氏荧光定量PCR LightCycler480使用方法进行Real time RT-PCR,检测GUS基因的表达情况。GUS基因的引物为GUS- RTF: 5′-ACGGGGAAACTCAGCAAGC-3′和GUS- RTR: 5′-TGAGCGTCGCAGAACATTACAT-3′。内参基因为拟南芥肌动蛋白基因AtACT2 (At3g 18780)。AtACT2基因的引物为ACT2-RTF: 5′-GGTAACATT- GTGCTCAGTGGTGG-3′和ACT2-RTR: 5′-AACGA- CCTTAATCTTCATGCTGC-3′。qRT-PCR扩增程序为:95℃预变性30 s;95℃15 s,60℃35 s,40次循环,绘制溶解曲线。采用Excel 2003统计数据,样本的Ct值取3次重复的平均值。
2 结果和分析 2.1 IpDHN启动子IpDHN-Pro的克隆根据厚藤IpDHN的cDNA序列,在起始密码子和终止密码子处设计引物,以基因组DNA为模板,PCR扩增获得IpDHN序列片段。回收片段连接于T载体上测序,并和IpDHN的cDNA序列阅读框进行比较,可知IpDHN序列中含有1个264 bp的内含子序列(图 1)。

|
图 1 IpDHN基因序列。阴影部分为IpDHN基因的内含子;下划线分别为引物SP2和SP1序列。 Fig. 1 Sequence of IpDHN. The sequence in gray is the intron, and the underlined sequences are primers SP2 and SP1. |
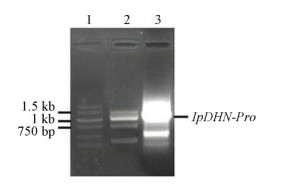
采用染色体步移法设计基因特异引物SP1和SP2, 参考Genome Walking Kit说明书,以厚藤基因组DNA为模板,经两轮PCR扩增获得1条明亮的DNA条带,大小约为1 kb (图 2)。
|
图 2 IpDHN启动子片段的PCR扩增。1:标准DNA; 2:第一轮PCR: 3:第二轮PCR。 Fig. 2 PCR of IpDHN promoter. 1: Standard DNA marker; 2: The first PCR; 3: The second PCR. |
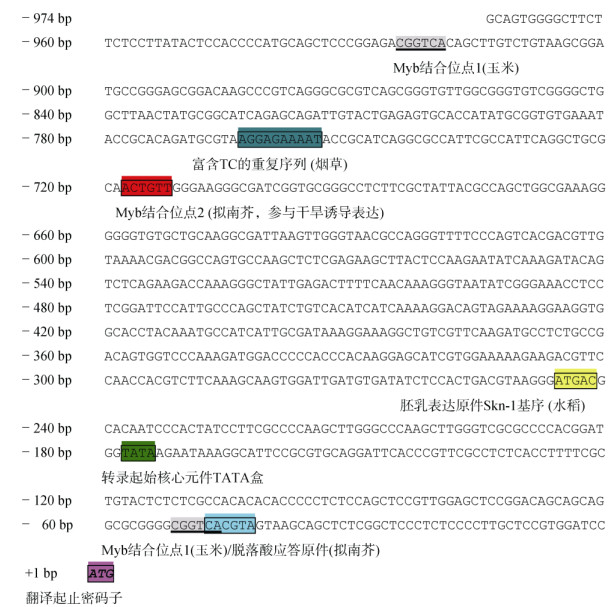
将该片段回收后连接于T载体上测序,即获得长度为974 bp的IpDHN的5′侧翼启动子序列, 将其输入在线分析软件PlantCARE和PLACE, 该序列除了含有真核生物转录启动核心元件TATA-box (TATA盒)和CAAT-box (CAAT盒)外,还存在一些其他的顺式作用元件(图 3)。起始转录必需的TATA- box位于起止密码子ATG上游178个碱基处(–178 bp); 而调控转录起始频率的CAAT-box位于起止密码子ATG上游140个碱基处(–140 bp)。在ATG上游48个碱基处(–48 bp)含有1个脱落酸应答元件(abscisic acid-response element; ABRE; TACGTG);而在ATG上游52、718和925个碱基处(–52、–718和–925 bp)还有3个Myb转录因子结合位点(myeloblastosis- binding sites, MBS1和MBS2; CGGTCA或CAA- CTG);ATG上游767个碱基处(–767 bp)含有植物防御反应相关的富含TC的重复序列(TC-rich repeat regulatory element; ATTTTCTCCA)。此外,在ATG上游246个碱基处(–246 bp)含有一个种子胚乳特异表达的顺式作用元件Skn-1基序(Skn-1 motif; GTCAT)。
|
图 3 启动子IpDHN-Pro序列和顺式作用元件 Fig. 3 Sequence of IpDHN-Pro and the cis-acting elements |
为探明IpDHN-Pro序列是否具有转录活性,将IpDHN-Pro连接至植物启动子表达载体pBI101.2中,并通过农杆菌介导的拟南芥转基因试验,验证其是否在拟南芥中具有启动基因转录的活性。选取经卡那霉素筛选的T3代纯合体转基因拟南芥,分别对其根、茎、叶、花、荚果和幼苗进行GUS染色。结果表明(图 4),GUS阳性信号在拟南芥的主根中最强,在莲座叶的叶中也有一定的表达,而在幼苗的下胚轴中几乎不表达;在成年植株叶片上有显色,且表达量一般;在花序中有较低的表达,在发育角果的基部和顶端有较强的表达。这说明该启动子可能属于根、叶丰富的启动子,在拟南芥中具备转录活性且正常生长条件下启动转录活性较低。

|
图 4 转IpDHN-Pro/pBI101.2拟南芥植株GUS组织染色。A:幼苗; B:真叶; C:主根; D:叶片; E:花序; F:角果。 Fig. 4 GUS staining of transgenic Arabidopsis with IpDHN-Pro/pBI101.2. A: Seedling; B: True leaves; C: Axial root; D: Leaf; E: Inflorescence; F: Silique. |
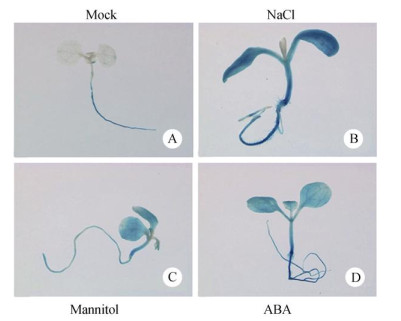
为进一步探索IpDHN-Pro的功能,对转IpDHN- Pro/pBI101.2拟南芥进行高盐、干旱、外源激素ABA等胁迫处理,然后进行GUS染色。在未经处理的对照拟南芥幼苗中,GUS阳性信号较弱, 但经200 mmol L–1 NaCl、300 mmol L–1甘露醇和0.1 mmol L–1 ABA分别处理24 h后,拟南芥幼苗的GUS信号明显增强(图 5),这表明在高盐、干旱和外源激素ABA处理下,IpDHN-Pro启动转录的活性有所增强,造成葡萄糖苷酸酶GUS在植株体内活性增强。
|
图 5 转基因拟南芥幼苗在胁迫处理24 h后的GUS活性。A:对照; B: 200 mmol L–1 NaCl; C: 300 mmol L–1甘露醇; D: 0.1 mmol L–1 ABA。 Fig. 5 GUS activity in transgenic Arabidopsis seedlings under stress for 24 h. A: Control; B: 200 mmol L–1 NaCl; C: 300 mmol L–1 mannitol; D: 0.1 mmol L–1 ABA. |
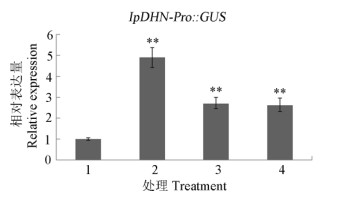
为了证明GUS活性的增强是由于GUS基因的表达上调引起的,我们同时采用qRT-PCR方法分析了胁迫处理后的拟南芥幼苗GUS基因表达情况。在高盐、高渗透压和外源激素ABA处理下GUS基因的表达上调,且表达量分别上调了4、2和2倍(图 6)。这进一步证明,在启动子IpDHN-Pro的控制下, GUS基因的表达受到了高盐、干旱和外源激素ABA等胁迫的诱导,即我们所克隆获得的厚藤脱水素IpDHN基因中974 bp的5′侧翼序列为启动子序列, 具有起始转录的活性,且其调控GUS基因的转录受高盐、干旱、外源激素ABA等胁迫的上调影响。
|
图 6 qRT-PCR分析转基因拟南芥幼苗胁迫处理24 h后GUS基因的表达。1:对照; 2: 200 mmol L–1 NaCl; 3: 300 mmol L–1甘露醇; 4: 0.1 mmol L–1 ABA。**: P < 0.01。 Fig. 6 Expression of GUS in transgenic Arabidopsis seedlings under stress by qRT-PCR. 1: Control; 2: 200 mmol L–1 NaCl; 3: 300 mmol L–1 mannitol; 4: 0.1 mmol L–1 ABA. **: P < 0.01. |
脱水素基因是植物体内受盐、干旱胁迫诱导的抗逆标志基因[3, 8, 15],有大量研究证明该类基因与植物耐盐、耐旱、抗冻等抗逆性密切相关[16]。高盐和干旱是植物生长过程中最常见的逆境胁迫,通常会引起植物失水,造成细胞内水平衡失衡,进而影响植物的生长和发育。对于农作物而言,由于高盐和干旱引起的农作物生长不良,进而造成农作物减产和品质下降是农业生产过程中的常见问题。因此, 在植物基因工程育种中,通过上调一些抗逆基因的表达,提高植物或作物的抗逆性,是一种最为行之有效的方法。
启动子是调控基因表达的主要因素,而启动子序列所包含的顺式作用元件通常能够决定该基因的转录受哪些转录因子调控,并在何种情况下启动转录或改变转录强度。我们的前期研究表明,厚藤IpDHN基因是一个典型的受盐旱等非生物胁迫及ABA诱导的抗逆功能基因[14],结合厚藤这一物种的极端耐盐耐旱性,我们推测IpDHN基因的表达调控可能在厚藤抗逆性中发挥了重要作用。基于以上假设,本研究克隆并分析了IpDHN基因的启动子序列,为深入了解厚藤的抗逆生物学特征奠定基础,也为植物抗逆遗传工程提供了可操作的遗传物质基础。
目前,盐生植物已成为克隆耐盐基因和盐害诱导启动子的主要来源[17]。厚藤是一种典型的热带亚热带盐生植物,我们通过对厚藤IpDHN基因的启动子区域(974 bp)进行鉴定,其含有多个与植物非生物逆境胁迫相关的顺式作用元件,如ABRE、MBS以及富含TC的重复调控元件等。ABRE是ABA信号中关键的基因转录顺式应答元件[18],广泛参与调控植物功能基因应答ABA信号及渗透压胁迫[19-20]。MBS主要通过与转录因子MYB结合,而很多MYB转录因子均与植物应答干旱胁迫和ABA信号密切相关[21-22]。富含TC的重复调控元件主要参与调控植物防御反应和抗逆应答[23],暗示了IpDHN基因的启动子序列IpDHN-Pro具备广泛抗逆应答特征。PlantCARE软件预测结果表明,IpDHN-Pro序列还存在1个胚乳诱导表达元件Skn-1[24],表明IpDHN基因能够在种子发育后期胚乳发育中表达,符合IpDHN基因作为LEA基因家族成员这一基本特征。
本研究还成功获得IpDHN-Pro调控GUS表达的转基因拟南芥。通过对拟南芥不同组织部位的化学染色分析,IpDHN-Pro具有在拟南芥中广泛调控基因表达的特征,但正常生长条件下,GUS表达水平较低,表明我们所克隆的IpDHN-Pro序列具备基本的起始基因转录功能,是一个有活性的启动子; 通过对转基因拟南芥幼苗进行高盐、渗透压胁迫及ABA处理,转基因拟南芥植株的GUS染色活性增强;同时qRT-PCR分析表明GUS的表达显著增强。这表明IpDHN-Pro启动子可驱动外源GUS基因受干旱或高盐和外源激素ABA的诱导。同时,也验证了前期对厚藤IpDHN基因表达研究的结论[14]。
本研究通过对克隆获得的974 bp的IpDHN基因的5′侧翼序列进行初步的生物信息学分析,鉴定出一部分可能的启动子顺式作用元件。通过转基因和GUS组织化学染色研究,阐明了该启动子序列具有起始基因转录的功能,证明IpDHN-Pro序列驱动GUS基因在拟南芥的不同组织中表达有差异, 以下胚轴、主根和叶片中的表达较强,在角果中的表达微弱,而在花序中几乎无表达,是1个根和叶丰富表达的启动子;GUS染色试验进一步证明该启动子能够在高盐、干旱和外源激素ABA等胁迫下增强细胞内GUS活性;qRT-PCR分析表明IpDHN-Pro序列驱动的GUS基因的表达受高盐、干旱和外源激素ABA诱导,均不同程度的上调表达。这为植物抗逆遗传工程育种中利用特定胁迫诱导的启动子调控植物抗逆性奠定理论基础。
| [1] |
MIRYEGANEH M, TAKAYAMA K, TATEISHI Y, et al. Long-distance dispersal by sea-drifted seeds has maintained the global distribution of Ipomoea pes-caprae subsp. brasiliensis (Convolvulaceae)[J]. PLoS One, 2014, 9(4): e91836. DOI:10.1371/journal.pone.0091836 |
| [2] |
OUYANG P Y, LIU N, ZHANG W W, et al. Biological and ecophysiological characteristics of a beach plant Ipomoea pescaprae[J]. J Hunan Univ Sci Technol (Nat Sci), 2011, 26(4): 117-121. 欧阳蒲月, 刘楠, 张伟伟, 等. 海滩植物厚藤(Ipomoea pescaprae)的生物学及生理生态特性[J]. 湖南科技大学学报(自然科学版), 2011, 26(4): 117-121. DOI:10.3969/j.issn.1672-9102.2011.04.025 |
| [3] |
GRAETHER S P, BODDINGTON K F. Disorder and function:A review of the dehydrin protein family[J]. Front Plant Sci, 2014, 5: 576. DOI:10.3389/fpls.2014.00576 |
| [4] |
HAND S C, MENZE M A, TONER M, et al. LEA proteins during water stress:Not just for plants anymore[J]. Annu Rev Physiol, 2011, 73: 115-134. DOI:10.1146/annurev-physiol-012110-142203 |
| [5] |
BIES-ETHÈVE N, GAUBIER-COMELLA P, DEBURES A, et al. Inventory, evolution and expression profiling diversity of the LEA (late embryogenesis abundant) protein gene family in Arabidopsis thaliana[J]. Plant Mol Biol, 2008, 67(1/2): 107-124. DOI:10.1007/s11103-008-9304-x |
| [6] |
CLOSE T J. Dehydrins:Emergence of a biochemical role of a family of plant dehydration proteins[J]. Physiol Plant, 1996, 97(4): 795-803. DOI:10.1111/j.1399-3054.1996.tb00546.x |
| [7] |
OUELLET F, HOUDE M, SARHAN F. Purification, characterization and cDNA cloning of the 200 kDa protein induced by cold acclimation in wheat[J]. Plant Cell Physiol, 1993, 34(1): 59-65. DOI:10.1093/oxfordjournals.pcp.a078400 |
| [8] |
HANIN M, BRINI F, EBEL C, et al. Plant dehydrins and stress tolerance:Versatile proteins for complex mechanisms[J]. Plant Signal Behav, 2011, 6(10): 1503-1509. DOI:10.4161/psb.6.10.17088 |
| [9] |
ZHOU Y, HU L F, XU S Y, et al. Identification and transcriptional analysis of dehydrin gene family in cucumber (Cucumis sativus)[J]. Acta Physiol Plant, 2018, 40(8): 144. DOI:10.1007/s11738-018-2715-7 |
| [10] |
YANG Y Z, HE M Y, ZHU Z G, et al. Identification of the dehydrin gene family from grapevine species and analysis of their responsiveness to various forms of abiotic and biotic stress[J]. BMC Plant Biol, 2012, 12: 140. DOI:10.1186/1471-2229-12-140 |
| [11] |
VAZQUEZ-HERNANDEZ M, ROMERO I, ESCRIBANO M I, et al. Deciphering the role of CBF/DREB transcription factors and dehydrins in maintaining the quality of table grapes cv. autumn royal treated with high CO2 levels and stored at 0℃[J]. Front Plant Sci, 2017, 8: 1591. DOI:10.3389/fpls.2017.01591 |
| [12] |
ZHU W N, ZHANG L S, LÜ H, et al. The dehydrin wzy2 promoter from wheat defines its contribution to stress tolerance[J]. Funct Integr Genomics, 2014, 14(1): 111-125. DOI:10.1007/s10142-013-0354-z |
| [13] |
ABEDINI R, GHANEGOLMOHAMMADI F, PISHKAMRAD R, et al. Plant dehydrins:Shedding light on structure and expression patterns of dehydrin gene family in barley[J]. J Plant Res, 2017, 130(4): 747-763. DOI:10.1007/s10265-017-0941-5 |
| [14] |
ZHANG H, ZHENG J X, SU H X, et al. Molecular cloning and functional characterization of the dehydrin (IpDHN) gene from Ipomoea pes-caprae[J]. Front Plant Sci, 2018, 9: 1454. DOI:10.3389/fpls.2018.01454 |
| [15] |
HARA M. The multifunctionality of dehydrins:An overview[J]. Plant Signal Behav, 2010, 5(5): 503-508. DOI:10.4161/psb.11085 |
| [16] |
KOSOVÁ K, VÍTÁMVÁS P, PRÁŠIL I T. Wheat and barley dehydrins under cold, drought, and salinity:What can LEA-Ⅱ proteins tell us about plant stress response?[J]. Front Plant Sci, 2014, 5: 343. DOI:10.3389/fpls.2014.00343 |
| [17] |
MISHRA A, TANNA B. Halophytes:Potential resources for salt stress tolerance genes and promoters[J]. Front Plant Sci, 2017, 8: 829. DOI:10.3389/fpls.2017.00829 |
| [18] |
HOBO T, ASADA M, KOWYAMA Y, et al. ACGT-containing abscisic acid response element (ABRE) and coupling element 3(CE3) are functionally equivalent[J]. Plant J, 1999, 19(6): 679-689. DOI:10.1046/j.1365-313x.1999.00565.x |
| [19] |
MISHRA S, SHUKLA A, UPADHYAY S, et al. Identification, occurrence, and validation of DRE and ABRE cis-regulatory motifs in the promoter regions of genes of Arabidopsis thaliana[J]. J Integr Plant Biol, 2014, 56(4): 388-399. DOI:10.1111/jipb.12149 |
| [20] |
WATANABE K A, HOMAYOUNI A, GU L K, et al. Transcriptomic analysis of rice aleurone cells identified a novel abscisic acid response element[J]. Plant Cell Environ, 2017, 40(9): 2004-2016. DOI:10.1111/pce.13006 |
| [21] |
URAO T, YAMAGUCHI-SHINOZAKI K, URAO S, et al. An Arabidopsis myb homolog is induced by dehydration stress and its gene product binds to the conserved MYB recognition sequence[J]. Plant Cell, 1993, 5(11): 1529-1539. DOI:10.1105/tpc.5.11.1529 |
| [22] |
ABE H, URAO T, ITO T, et al. Arabidopsis AtMYC2(bHLH) and AtMYB2(MYB) function as transcriptional activators in abscisic acid signaling[J]. Plant Cell, 2003, 15(1): 63-78. DOI:10.1105/tpc.06130 |
| [23] |
GERMAIN H, LACHANCE D, PELLETIER G, et al. The expression pattern of the Picea glauca Defensin 1 promoter is maintained in Arabidopsis thaliana, indicating the conservation of signalling pathways between angiosperms and gymnosperms[J]. J Exp Bot, 2012, 63(2): 785-795. DOI:10.1093/jxb/err303 |
| [24] |
FAUTEUX F, STRÖMVIK M V. Seed storage protein gene promoters contain conserved DNA motifs in Brassicaceae, Fabaceae and Poaceae[J]. BMC Plant Biol, 2009, 9: 126. DOI:10.1186/1471-2229-9-126 |
 2019, Vol. 27
2019, Vol. 27


