蔗糖合成酶(sucrose synthase, SuSy)是植物体内蔗糖代谢的重要调控酶,不仅参与调控蔗糖代谢,而且能够影响细胞分化和细胞壁的形成,为细胞壁的合成提供底物[1-3]。纤维素合成的直接底物是尿苷二磷酸-D-葡萄糖(UDPG),在多个酶和基因的调控下,SuSy催化蔗糖和UDP生成UDPG,所以SuSy影响纤维素合成[4-5]。有研究表明,SuSy参与植物韧皮部的代谢、木纤维细胞分化等[6-10]。SuSy基因对增加棉花(Gossypium spp.)、烟草(Nicotiana tabacum)和杨树(Populus sp.)中纤维素含量、纤维长度、提高纤维强度至关重要,抑制SuSy基因的表达, 会降低棉花纤维质量[11-13]。
SuSy基因广泛存在于高等植物中,Cardini等首次从小麦(Triticum aestivum)中克隆了SuSy基因[14], 此后从拟南芥(Arabidopsis thaliana)、棉花、马铃薯(Solanum tuberosum)、胡萝卜(Daucus carota)、玉米(Zea mays)、柑橘(Citrus reticulata)、水稻(Oryza sativa)、枣(Ziziphus jujuba)、甘蔗(Saccharum offici- narum)等植物中获得SuSy基因[15-21]。但关于巨桉SuSy基因方面的研究鲜见报道[22]。
桉树(Eucalyptus sp.)作为三大速生材树种之一,是重要的制浆造纸原料,具有成为新型生物质能源树种的潜力[23-24]。巨桉(E. grandis)原产于澳大利亚东部沿海地区,作为我国优质的速生用材林树种,现已大面积种植[25]。目前,巨桉基因组测序已经完成[26],为桉树分子生物学研究奠定了坚实的基础。本研究根据巨桉基因组测序结果,利用生物信息学方法系统研究了SuSy基因家族成员的序列信息,主要包括EgrSuSy基因家族成员的染色体分布、亚细胞定位、信号肽预测、基因和蛋白质结构、表达模式和进化分析等,为深入研究巨桉纤维素代谢通路的分子机制和EgrSuSy在巨桉木材形成中的作用提供提供科学依据。
1 材料和方法 1.1 SuSy家族基因序列的提取巨桉、毛果杨、拟南芥、玉米和水稻SuSy基因家族成员基因序列信息于2017年11月下载于Phytozome (http://www.phytozome.net)、美国国立生物技术信息中心数据库(NCBI) (http://www.ncbi.nlm.nih.gov/)。
1.2 EgrSuSy家族基本特征和亚细胞定位分析利用ExPASy中的ProtParam (http://web.expasy.org/protparam/)分析EgrSuSy的相对分子质量、理论等电点、不稳定系数、亲水性指数和脂溶性指数; 采用SignalP (http://www.cbs.dtu.dk/services/SignalP/)预测EgrSuSy家族蛋白的信号肽;采用WoLF PSORT (http://www.genscript.com/wolf-psort.html)预测EgrSuSy家族蛋白的亚细胞定位。
1.3 EgrSuSy家族基因结构和蛋白质结构分析利用GSDS2.0 (http://gsds.cbi.pku.edu.cn/)分析基因的CDS序列和全基因组序列;利用SOPMA (https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)分析蛋白序列二级结构; 利用SWISS-MODEL (https://swissmodel.expasy.org/interactive#sequence)分析蛋白序列3D结构。
1.4 EgrSuSy的进化学分析利用ClustalW软件比对分析EgrSuSy家族蛋白氨基酸序列同毛果杨、拟南芥、玉米以及水稻的SuSy家族蛋白氨基酸序列,参数为默认值;用MEGA 6.0中的邻接(Neighbor-Joining)算法绘制系统进化树,用重复1 000次的bootstrap进行检验。
1.5 EgrSuSy的表达分析EgrSuSy在未成熟木质部、成熟叶片、韧皮部、茎尖、木质部和幼叶的转录组数据下载于Phytozome (http://www.phytozome.net)。用EgrSuSy基因的FPKM值表示基因的表达丰度,以Log2的对数绘制基因表达热图。
2 结果和分析 2.1 EgrSuSy基因家族鉴定与理化特征分析从Phytozome与NCBI数据库中筛选出18条EgrSuSy基因序列,分别命名为EgrSuSy1~EgrSuSy18, 分布在7条染色体上,其中6个SuSy基因位于3号染色体上(表 1)。EgrSuSy基因编码405~2 351个氨基酸,编码氨基酸序列最长的是EgrSuSy15,相对分子量最大,为274.762 kD;EgrSuSy3的相对分子量最小,为46.931 kD。18个EgrSuSy蛋白的理论等电点为5.27~9.12,EgrSuSy10、EgrSuSy15为碱性蛋白,其余均为酸性蛋白。EgrSuSy11的不稳定系数最小,为27.39。根据亲水性系数可知, EgrSuSy基因家族编码的蛋白均为亲水蛋白, EgrSuSy的脂溶指数为81.88~96.38,EgrSuSy蛋白定位在细胞质膜上,且EgrSuSy1~EgrSuSy18均不存在信号肽。
| 表 1 EgrSuSy基因家族成员的基本信息 Table 1 Informations of EgrSuSy gene family |
EgrSuSy家族成员的基因结构中均含有内含子、外显子(图 1),但各成员仍有明显的差异,表现在外显子、内含子的数量和位置不同。EgrSuSy15的外显子数量最多(25个),EgrSuSy3的最少(8个)。
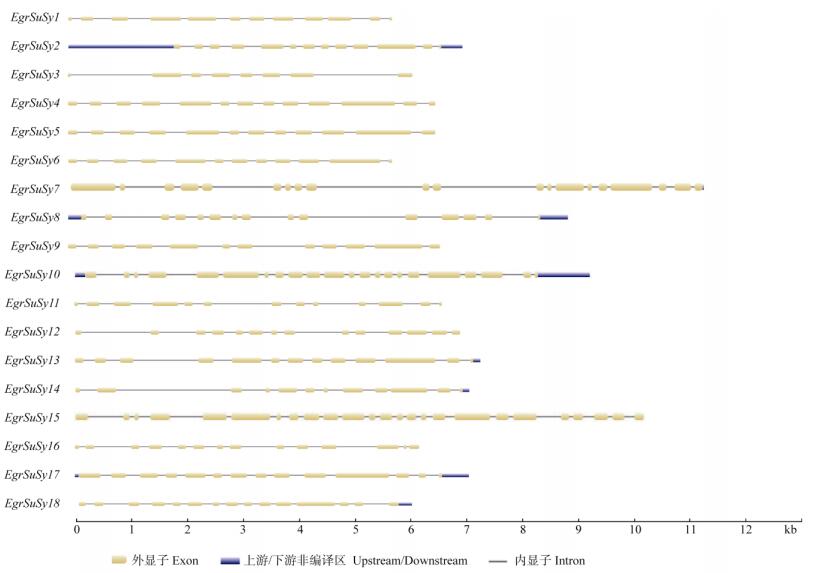
|
图 1 EgrSuSy的基因结构 Fig. 1 Structures of EgrSuSy genes |
对EgrSuSy基因家族18条蛋白的二级结构的预测结果表明,EgrSuSy蛋白由α-螺旋、延伸链、无规则卷曲、β-转角组成。EgrSuSy蛋白的α-螺旋比例最高,其次为无规则卷曲(表 2)。
| 表 2 EgrSuSy的二级结构分析 Table 2 Second structures of EgrSuSy proteins |
由图 2可知,EgrSuSy蛋白的3D结构由多个螺旋、延伸链和转角组成。SWISS-MODEL结果表明,EgrSuSy蛋白的3D结构与拟南芥的相似性较高,达85.69%,且EgrSuSy2、EgrSuSy4、EgrSuSy5、EgrSuSy6、EgrSuSy8、EgrSuSy9、EgrSuSy12、EgrSuSy16、EgrSuSy17和EgrSuSy18的3D结构相似,EgrSuSy1和EgrSuSy11相似,均为四聚体结构。
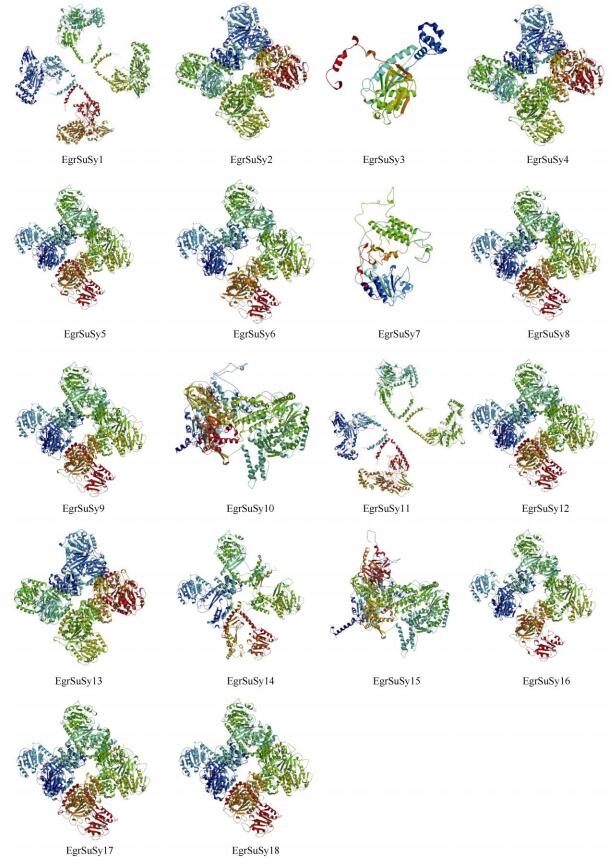
|
图 2 EgrSuSy蛋白的3D结构 Fig. 2 3D structures off EgrSuSy proteins |
构建基于SuSy蛋白的系统进化树(图 3),结果表明,EgrSuSy同毛果杨的进化关系最近。同时, EgrSuSy5、EgrSuSy6、EgrSuSy13、EgrSuSy9、EgrSuSy4、EgrSuSy14、EgrSuSy2、EgrSuSy1和EgrSuSy11的进化关系极为相近,EgrSuSy15和EgrSuSy10相近,EgrSuSy8和EgrSuSy12相近, EgrSuSy1、EgrSuSy2、EgrSuSy5、EgrSuSy6、EgrSuSy9和EgrSuSy14相近。
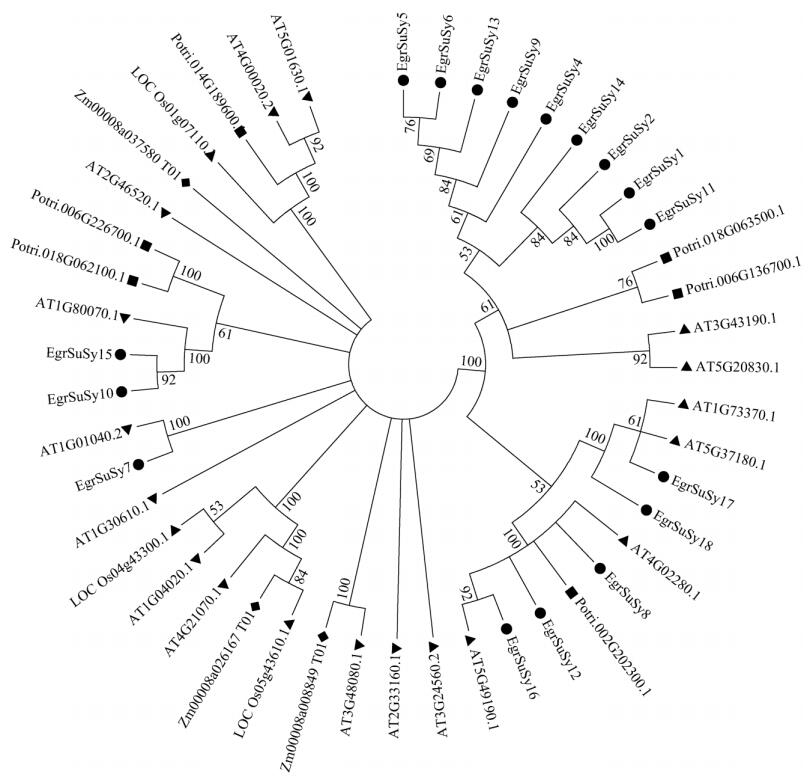
|
图 3 基于SuSy蛋白构建的系统进化树。Egr:巨桉; Potri:毛果杨; AT:拟南芥; Zm:玉米; LOC:水稻。 Fig. 3 Phylogenetic tree based on proteins of SuSy. Egr: Eucalyptus grandis; Potri: Populus trichocarpa; AT: Arabidopsis thaliana; Zm: Zea mays; LOC: Oryza sativa. |
从基因表达谱热图(图 4)可以看出,EgrSuSy基因在巨桉的未成熟木质部、成熟叶片、韧皮部、茎尖、木质部和幼叶中的表达不同。EgrSuSy2、EgrSuSy3、EgrSuSy4、EgrSuSy7、EgrSuSy8和EgrSuSy10在这些组织中的表达量相对较高, EgrSuSy2、EgrSuSy3、EgrSuSy4和EgrSuSy8在未成熟木质部、韧皮部、木质部的表达量相对较高, 可能与巨桉的纤维素代谢有关。EgrSuSy5、EgrSuSy6、EgrSuSy12、EgrSuSy13、EgrSuSy15和EgrSuSy16在这些组织中的表达均较低。EgrSuSy基因成员在不同组织中的表达差异,说明它们在巨桉不同组织、发育时期的功能存在差异。
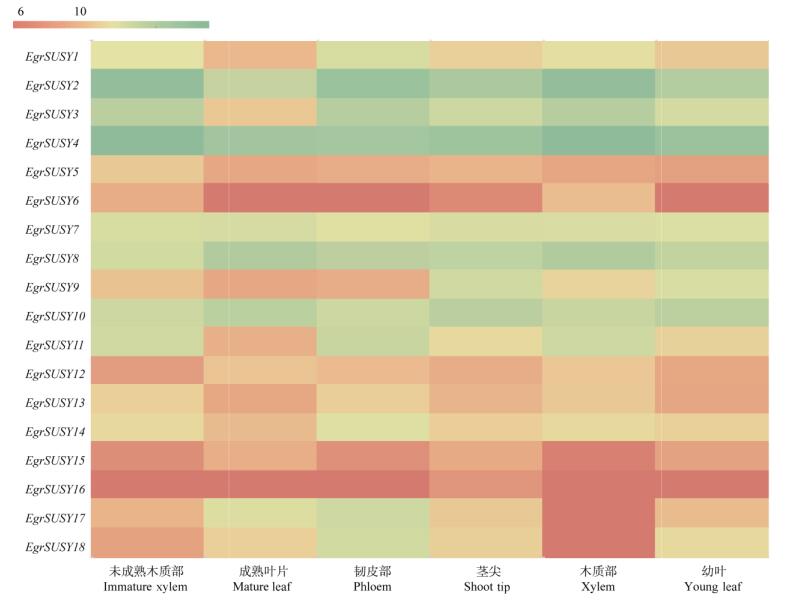
|
图 4 EgrSuSy基因在巨桉不同组织中的表达 Fig. 4 Expression of EgrSuSy in different tissues of Eucalyptus grandis |
SuSy是植物蔗糖代谢中的关键调节酶,在细胞生长分化的相关调控和结构组成中具有重要的功能[27],在淀粉和纤维素的合成等过程中发挥重要作用[23, 28]。SuSy的活性与纤维素合成有关,主要是参与调节细胞壁的构建以及细胞发育[9, 13]。
本研究分析了EgrSuSy家族成员的生物学基本信息,为深入研究EgrSuSy基因功能、揭示SuSy在巨桉中的生物学功能和了解桉树纤维素代谢奠定基础。拟南芥SuSy1是由2个对称的四聚体组成[29-30],本研究表明,EgrSuSy家族成员多为四聚体结构。在大麦、黄瓜中,SuSy定位在细胞质和质膜中,SuSy以可溶性形式存在于细胞质中,亦可结合在质膜上,因存在形式不同而行使不同的功能; 当其结合在质膜上时,为纤维素、胼胝质合成提供前体物质[31]。本研究表明,EgrSuSy定位在细胞质膜上,其具体功能还有待进一步研究。
本研究分析了巨桉与毛果杨、玉米、水稻和拟南芥SuSy基因家族间的进化关系,结果表明,巨桉和毛果杨的SuSy基因同源性较高,表明毛果杨和巨桉的SuSy家族在生物学功能、生长调控机制上存在相似性。结构分析与进化分析表明, EgrSuSy15、EgrSuSy10结构相似、进化关系相近,EgrSuSy8、EgrSuSy12亦结构相似、进化关系相近,故可推测其功能相似。
植物在不同的生长发育阶段和不同器官中,蔗糖合成酶基因表达具有时间和空间的差异。海岛棉(Gossypium barbadense)的SuSy基因在花和果柄中的表达较高,而在根和果实中的表达普遍偏低,海岛棉SuSy3A基因在纤维次生壁加厚初期起作用,而SuSy1A、SuSy1D基因作用于纤维次生壁加厚后期和末期[32]。本研究中,EgrSuSy基因家族在不同组织中的差异表达,说明它们在巨桉不同组织、发育时期的功能存在差异。EgrSuSy2、EgrSuSy3、EgrSuSy4、EgrSuSy8在未成熟的木质部、韧皮部、木质部的表达量较高,推测这些成员的功能与巨桉纤维素代谢相关。EgrSuSy2、EgrSuSy3、EgrSuSy4、EgrSuSy7、EgrSuSy8和EgrSuSy10在6个组织中的表达量相对较高,说明这些基因有可能参与了巨桉各组织的生长和代谢过程。
| [1] | Barrero-sicilia C, Hernando-amado S, González-melendi P, et al. Structure, expression profile and subcellular locali-sation of four different sucrose synthase genes from barley[J]. Planta, 2011, 234(2): 391-403. DOI:10.1007/s00425-011-1408-x |
| [2] | POOVAIAH C R, MAZAREI M, DECKER S R, et al. Transgenic switchgrass (Panicum virgatum L.) biomass is increased by over-expression of switchgrass sucrose synthase (PvSUS1)[J]. Biotechnol J, 2015, 10(4): 552-563. DOI:10.1002/biot.201400499 |
| [3] |
FANG J G, ZHU X D, JIA H F, et al. Research advances on physio-logical function of plant sucrose synthase[J].
J Nanjing Agric Univ, 2017, 40(5): 759-768. 房经贵, 朱旭东, 贾海锋, 等. 植物蔗糖合酶生理功能研究进展[J]. 南京农业大学学报, 2017, 40(5): 759-768. DOI:10.7685/jnau.201706003 |
| [4] | DELMER D P, HAIGLER C H. The regulation of metabolic flux to cellulose, a major sink for carbon in plants[J]. Metab Eng, 2002, 4(1): 22-28. DOI:10.1006/mben.2001.0206 |
| [5] | HAIGLER C H, IVANOVA-DATCHEVA M, HOGAN P S, et al. Carbon partitioning to cellulose synthesis[J]. Plant Mol Biol, 2001, 47(1/2): 29-51. DOI:10.1023/A:1010615027986 |
| [6] | MARTIN T, FROMMER W B, SALANOUBAT M, et al. Expression of an Arabidopsis sucrose synthase gene indicates a role in metabo-lization of sucrose both during phloem loading and in sink organs[J]. Plant J, 1993, 4(2): 367-377. DOI:10.1046/j.1365-313X.1993.04020367.x |
| [7] | COLEMAN H D, YAN J, MANSFIELD S D, et al. Sucrose synthase affects carbon partitioning to increase cellulose production and altered cell wall ultrastructure[J]. Proc Natl Acad Sci USA, 2009, 106(31): 13118-13123. DOI:10.1073/pnas.0900188106 |
| [8] | NOLTE K D, HENDRIX D L, RADIN J W, et al. Sucrose synthase localization during initiation of seed development and trichome differentiation in cotton ovules[J]. Plant Physiol, 1995, 109(4): 1285-1293. DOI:10.1104/pp.109.4.1285 |
| [9] | ALBRECHT G, MUSTROPH A. Localization of sucrose synthase in wheat roots:increased in situ activity of sucrose synthase correlates with cell wall thickening by cellulose deposition under hypoxia[J]. Planta, 2003, 217(2): 252-260. DOI:10.1007/s00425-003-0995-6 |
| [10] | FUJII S, HAYASHI T, MIZUNO K. Sucrose synthase is an integral component of the cellulose synthesis machinery[J]. Plant Cell Physiol, 2010, 51(2): 294-301. DOI:10.1093/pcp/pcp190 |
| [11] | JIANG Y J, GUO W Z, ZHU H Y, et al. Overexpression of GhSusA1 increases plant biomass and improves cotton fiber yield and quality[J]. Plant Biotechnol J, 2012, 10(3): 301-312. DOI:10.1111/j.1467-7652.2011.00662.x |
| [12] | WEI Z G, QU Z S, ZHANG L J, et al. Overexpression of poplar xylem sucrose synthase in tobacco leads to a thickened cell wall and increased height[J]. PLoS One, 2015, 10(3): e0120669 DOI:10.1371/journal.pone.0120669 |
| [13] | XU S M, BRILL E, LLEWELLYN D J, et al. Overexpression of a potato sucrose synthase gene in cotton accelerates leaf expansion, reduces seed abortion, and enhances fiber production[J]. Mol Plant, 2012, 5(2): 430-441. DOI:10.1093/mp/ssr090 |
| [14] | CARDINI C E, LELOIR L F, CHIRIBOGA J. The biosynthesis of sucrose[J]. J Biol Chem, 1955, 214(1): 149-156. |
| [15] |
LEI M H, YE B Y, WANG B M, et al. Cloning of sucrose synthase gene from sugarcane (Saccharum officenarum L.)[J].
Chin J Appl Environ Biol, 2008, 14(2): 177-179. 雷美华, 叶冰莹, 王冰梅, 等. 甘蔗蔗糖合成酶基因的克隆[J]. 应用与环境生物学报, 2008, 14(2): 177-179. DOI:10.3321/j.issn:1006-687X.2008.02.006 |
| [16] | BAUD S, VAULTIER M N, ROCHAT C. Structure and expression profile of the sucrose synthase multigene family in Arabidopsis[J]. J Exp Bot, 2004, 55(396): 397-409. DOI:10.1093/jxb/erh047 |
| [17] | CARLSON S J, CHOUREY P S, HELENTJARIS T, et al. Gene expression studies on developing kernels of maize sucrose synthase (SuSy) mutants show evidence for a third SuSy gene[J]. Plant Mol Biol, 2002, 49(1): 15-29. DOI:10.1023/A:1014457901992 |
| [18] | HIROSE T, SCOFIELD G N, TERAO T. An expression analysis profile for the entire sucrose synthase gene family in rice[J]. Plant Sci, 2008, 174(5): 534-543. DOI:10.1016/j.plantsci.2008.02.009 |
| [19] | CHEN A Q, HE S, LI F F, et al. Analyses of the sucrose synthase gene family in cotton:Structure, phylogeny and expression patterns[J]. BMC Plant Biol, 2012, 12(1): 85-102. DOI:10.1186/1471-2229-12-85 |
| [20] |
FENG Y Z, WEI Q Q, HE X, et al. Cloning and expression analysis of sucrose synthase gene (SS6) in Ziziphus jujube[J].
Nonwood For Res, 2017(4): 36-42. 冯延芝, 魏琦琦, 何潇, 等. 枣蔗糖合成酶基因SS6的克隆及表达分析[J]. 经济林研究, 2017(4): 36-42. DOI:10.14067/j.cnki.1003-8981.2017.04.006 |
| [21] |
JIA C P, GENG H W, ZHU Y F, et al. Cloning between bioinformatics and expression pattern analysis of sucrose synthase in island cotton (Gossypium barbadense L.)[J].
Mol Plant Breed, 2016, 14(2): 286-301. 贾春平, 耿洪伟, 朱亚夫, 等. 海岛棉蔗糖合成酶基因克隆及生物信息学和表达模式分析[J]. 分子植物育种, 2016, 14(2): 286-301. |
| [22] | SHINYA T, IWATA E, NAKAHAMA K, et al. Transcriptional profiles of hybrid Eucalyptus genotypes with contrasting lignin content reveal that monolignol biosynthesis-related genes regulate wood composition[J]. Front Plant Sci, 2016, 7: 443 DOI:10.3389/fpls.2016.00443 |
| [23] |
XIE Y J, TAN X F. Introduction of Eucalyptus urophylla and the evaluation of its biological invasion risk[J].
J CS Univ For Technol, 2007, 27(6): 169-173. 谢耀坚, 谭晓风. 尾叶桉遗传资源引进及其生物学入侵风险评估[J]. 中南林业科技大学学报, 2007, 27(6): 169-173. DOI:10.3969/j.issn.1673-923X.2007.06.022 |
| [24] |
ZHOU Q Y, CHEN S X, HAN F Y, et al. Studies on biomass and energy characteristics of short rotation energy plantations of Eucalyptus urophylla×E. grandis[J].
J Trop Subtrop Bot, 2013, 21(1): 45-51. 周群英, 陈少雄, 韩斐扬, 等. 短周期尾巨桉能源林生物量与能量特征研究[J]. 热带亚热带植物学报, 2013, 21(1): 45-51. DOI:10.3969/j.issn.1005-3395.2013.01.006 |
| [25] |
YI Z B, XIONG J W. Eucalyptus grandis introduction cutivation and management technology in Guang'an[J].
Ningxia J Agric For Sci Technol, 2011, 52(3): 80-81, 84. 易昭彬, 熊剑文. 巨桉引种栽培管理技术[J]. 宁夏农林科技, 2011, 52(3): 80-81, 84. DOI:10.3969/j.issn.1002-204X.2011.03.039 |
| [26] | YU X, GUO Y, ZHANG X, et al. Integration of EST-CAPS markers into genetic maps of Eucalyptus urophylla and E. tereticornis and their alignment with E. grandis genome sequence[J]. Silv Genet, 2012, 61(6): 247-255. |
| [27] | KLECZKOWSKI L A, KUNZ S, WILCZYNSKA M. Mechanisms of UDP-glucose synthesis in plants[J]. Crit Rev Plant Sci, 2010, 29(4): 191-203. DOI:10.1080/07352689.2010.483578 |
| [28] | BAI W Q, XIAO Y H, ZHAO J, et al. Gibberellin overproduction promotes sucrose synthase expression and secondary cell wall depo-sition in cotton fibers[J]. PLoS One, 2014, 9(5): e96537 DOI:10.1371/journal.pone.0096537 |
| [29] | SCHMÖLZER K, GUTMANN A, DIRICKS M, et al. Sucrose synthase:A unique glycosyltransferase for biocatalytic glycosylation process development[J]. Biotechnol Adv, 2016, 34(2): 88-111. DOI:10.1016/j.biotechadv.2015.11.003 |
| [30] | ZHENG Y, ANDERSON S, ZHANG Y F, et al. The structure of sucrose synthase-1 from Arabidopsis thaliana and its functional impli-cations[J]. J Biol Chem, 2011, 286(41): 36108-36118. DOI:10.1074/jbc.M111.275974 |
| [31] | RUAN Y L, LLEWELLYN D J, FURBANK R T. Suppression of sucrose synthase gene expression represses cotton fiber cell initiation, elongation, and seed development[J]. Plant Cell, 2003, 15(4): 952-964. DOI:10.1105/tpc.010108 |
| [32] |
JIA C P, GENG H W, NI Z Y, et al. Activity changes and expression profiles of Sus with relation to fiber strength in island cotton (Gossypium barbadense L.)[J].
Acta Bot Boreali-Occid Sin, 2016, 36(2): 249-256. 贾春平, 耿洪伟, 倪志勇, 等. 海岛棉Sus活性变化特征及时空表达模式与纤维比强度的关系[J]. 西北植物学报, 2016, 36(2): 249-256. DOI:10.7606/j.issn.1000-4025.2016.02.0249 |
 2018, Vol. 26
2018, Vol. 26


