UGE基因为脱氢酶/还原酶超基因家族成员(SDRs)[1],编码UDP-半乳糖/葡萄糖-4-差向异构酶, UGE是半乳糖代谢途径中的一个关键酶,与半乳糖激酶、1-磷酸半乳糖尿苷酰转移酶共同参与半乳糖代谢活动[2-3],催化UDP-葡萄糖与UDP-半乳糖间的相互转化[1]。高等植物中多种复杂碳水化合物在糖代谢中起着重要作用,一些糖类如蔗糖、果聚糖、棉子糖等可以作为渗透调节物质保护细胞抵御外界胁迫。UGE可以使植株的棉子糖含量增加,而棉子糖作为渗透保护物质,可以保护植物细胞免受非生物胁迫引起的活性氧伤害[4]。UDP-半乳糖/葡萄糖- 4-差向异构酶在生物体中广泛存在,植物中UDP-糖作为主要的代谢中间产物、存储物质和结构组分, 为糖蛋白和糖脂提供重要的前体物质,也是合成纤维素、木葡聚糖等细胞壁多糖的重要底物[5-6]。除合成代谢外,UGE也参与到半乳糖的分解代谢中,缺少UGE会加剧植物和酵母中半乳糖的毒性作用[7]。这表明UGE基因在植物生长发育过程中发挥重要的作用。目前已从许多植物中克隆出多个UGE基因, 拟南芥(Arabidopsis thaliana)中已成功鉴定了5个UGE基因成员[7];经数据库搜索和荧光定量PCR分析表明,大麦(Hordeum vulgare)中至少有2个UGE基因成员[8];在水稻(Oryza sativa)中至少有4个UGE基因成员[9];在毛竹(Phyllostachys edulis)中克隆了UGE基因,转化拟南芥后,认为其可能与次生根形成有关[10]。毛竹过表达UGE基因能提高非生物胁迫的抗性,这表明在毛竹生长发育过程中,PeUGE可能是细胞壁多糖合成中的一个重要基因。UGE在半乳糖代谢和碳水化合物合成方面有重要的作用,它能将过量的UDP-半乳糖转化成UDP-葡萄糖, 拟南芥UGE基因编码的5个UGE都能够提高半乳糖的耐受性[7],减少半乳糖对植株的毒性作用。UGE基因还可能会参与到胁迫应答过程中[11],转OsUGE植株在干旱、盐以及低温胁迫下耐受性均得到提高,而植株表型不发生改变[4]。UGE基因参与细胞壁多糖和重要糖类化合物的生物合成[12-13]、抵御胁迫环境等诸多生理生化过程,在植物的生长发育中起着至关重要的作用。
鹅掌楸(Liriodendron chinense)主要分布在我国中部及长江以南的山区,是我国重要的珍贵用材和园林观赏树种,鹅掌楸种群规模小、生境濒危,现已被我国列为濒危保护植物[14-15],因此,研究鹅掌楸的适应性对其推广栽培有重要的意义[16]。本研究以鹅掌楸为研究材料,利用RT-PCR和RACE技术克隆鹅掌楸UGE基因全长表达序列,并进行生物信息学和组织表达特异性分析,为研究鹅掌楸适应性和抗逆生理奠定基础。
1 材料和方法 1.1 材料2016年春季,在南京林业大学下蜀实习林场鹅掌楸属种源试验林地采集材料,并迅速放入液氮中冷冻,带回实验室置于-80℃超低温冰箱中保存备用。基因克隆材料为26年生鹅掌楸(种源为松阳)叶片和花瓣的混合样,qPCR材料为叶片、花瓣、雄蕊、雌蕊、花萼、花芽、叶芽和茎。
1.2 基因克隆及ORF区验证用植物总RNA提取试剂盒(TIANGEN)提取鹅掌楸总RNA,用Thermo NanoDrop 2000紫外分光光度计检测提取的RNA浓度和纯度,用1%的琼脂糖凝胶电泳检测RNA的完整性。以提取的总RNA为模板,按照PrimeScriptTM RT Master Mix反转录试剂盒(TaKaRa)说明书合成cDNA第一链,用3′-Full RACE Core Set with PrimeScriptTM RTase反转录试剂盒合成cDNA的3′末端序列,用SMARTer® RACE 5′/3′ Kit (TaKaRa)反转录试剂盒合成cDNA的5′末端序列。
在北美鹅掌楸转录组数据库(http://ancangio.uga.edu/content/liriodendron-tulipifera)中搜索UGE基因的EST序列,用Oligo 7软件设计中间片段的引物(表 1),以cDNA第一链为模板扩增中间片段。根据中间片段设计3′ RACE引物,以反转录后3′ RACE的cDNA为模板进行巢式PCR,克隆测序后将中间片段和3′ RACE进行拼接。根据拼接结果设计5′ RACE引物,以反转录后5′ RACE的cDNA为模板进行巢式PCR,得到5′端序列后拼接cDNA全长。
| 表 1 基因克隆所用引物序列 Table 1 Primer sequences for gene clone |
用ORF Finder在线软件(https://www.ncbi.nlm.nih.gov/orffinder)预测UGE基因的开放阅读框,并在ORF区的两端设计引物,反向引物不包括终止密码子,进行PCR扩增,将PCR产物切胶回收, 连接pEASY®-Blunt克隆载体后涂板测序,验证UGE基因的ORF区。
1.3 生物信息学分析用在线软件ExasyProtParam (http://web.expasy.org/protparam/)预测LcUGE蛋白质的理化性质,用在线软件TargetP 1.1 Server (http://www.cbs.dtu.dk/services/TargetP/)预测LcUGE蛋白质的亚细胞位置。利用在线软件SOPMA (https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)对LcUGE蛋白质进行二级结构预测。用InterProScan (http://www.ebi.ac.uk/InterProScan/index.html)工具进行结构域和功能位点的预测。在NCBI数据库的BLASTP中比对LcUGE蛋白质的同源序列,选取相似性较高的蛋白序列,用MEGA 6.0软件构建系统进化树, 预测LcUGE与其他树种UGE的亲缘关系[17]。
1.4 LcUGEs基因的gDNA克隆和结构分析内含子是分析物种间种群结构和重建近缘物种间关系较为适合的标记[18-19]。Lessa开创了内含子靶向PCR[20]。非编码序列,尤其是内含子序列的变异性是群体变异和分类的有价值标记,用其保守的外显子引物通过PCR方法来扩增分析,被称为EPIC- PCR技术(exon-primed-intron-crossing-polymerase chain reaction)[21-22],这种方法可以通过证明内含子长度多态性来识别内含子序列显著的变异性[23]。
用DNA secure Plant Kit新型植物基因组DNA提取试剂盒(TIANGEN DP320)从鹅掌楸的叶片中提取基因组DNA。将获得的LcUGE基因cDNA序列与拟南芥、水稻等数据库基因组比对,推测LcUGE基因外显子与内含子的断裂位点,利用在外显子区域设计引物(表 2)扩增内含子序列,逐段克隆出LcUGE1和LcUGE2基因内含子,最终利用Seqman 2软件完成gDNA的拼接与验证。
| 表 2 LcUGE1与LcUGE2基因组引物序列 Table 2 Primer sequences for LcUGE1 and LcUGE2 genome |
EPIC-PCR反应体积50 μL,包括PrimeSTAR Max DNA Polymerase (TaKaRa) 25 μL,正反向引物各2 μL,灭菌水20 μL;反应程序为:预变性98℃1 min; 变性98℃ 10 s,退火15 s,延伸5 s,30~35个循环。
1.5 组织表达分析分别提取鹅掌楸的叶、茎、花瓣、花萼、雄蕊、雌蕊、花芽、叶芽等组织的RNA,用PrimeScriptTM RT Master Mix (Perfect Real Time)反转录试剂盒(TaKaRa)合成cDNA后稀释10倍,将cDNA模板统一稀释到1 000 ng μL–1,使用SYBR® Premix Ex TaqTM (Tli RNaseH Plus)试剂盒进行实时定量RT- PCR (qRT-PCR),以Actin作为内参基因[24],引物Actin-F (5′-GATCTTGCTGGGCGTGACCTC-3′)和Actin- R (5′-CCATCGGGCATCTCGTAGC-3′),检测LcUGE1与LcUGE2在组织中的表达情况。
实时定量反应体积为20 μL,包括SYBR Premix Ex Taq (Tli RNaseH Plus)(2×) 10 μL,正反向引物(表 3)各0.4 μL,ROX Reference Dye (50×) 0.4 μL,cDNA模板2 μL,灭菌水6.8 μL;在ABI StepOnePlus实时定量仪器上的反应程序为:预变性95℃ 30 s; 95℃ 5 s,60℃ 30 s,40个循环。
| 表 3 qPCR引物 Table 3 Primer sequences for qPCR |
提取鹅掌楸总RNA后,用NanoDrop 2000分光光度计检测RNA的浓度和纯度,表明OD260/OD280均为1.8~2.2,OD260/OD230大于2。用琼脂糖凝胶电泳检测RNA的完整性,28S RNA和18S RNA清晰可见,且前者亮度约为后者的2倍(图 1)。
|
图 1 RNA提取。M: DL2 000 DNA Marker; 1:叶片; 2:花瓣; 3:茎; 4:花萼; 5:雄蕊; 6:雌蕊; 7:花芽; 8:叶芽。 Fig. 1 RNA extraction. M: DL2 000 DNA Marker; 1: Leaf; 2: petal; 3: Stem; 4: Petal; 5: Steman; 6: Pistil; 7: Flower bud; 8: Leaf bud. |
以LcUGE1基因cDNA第一链为模板扩增出长度为670 bp的中间片段,然后分别以3′ RACE cDNA和5′ RACE cDNA为模板,结合特异性引物进行巢式PCR,最后将序列拼接得到LcUGE1基因cDNA,长度为1 531 bp。以LcUGE2基因的cDNA第一链为模板扩增出长度为635 bp的中间片段,用同样的方法得到LcUGE2基因cDNA,长度为1 378 bp (图 2)。
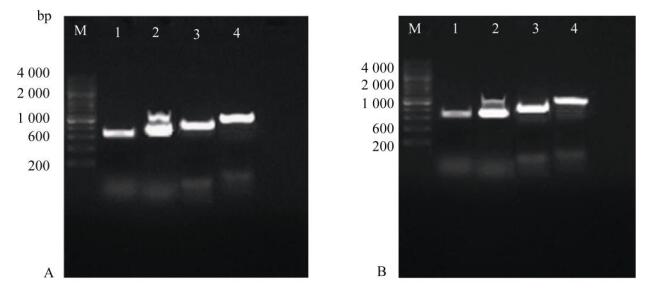
|
图 2 LcUGE基因的PCR扩增结果。A: UGE1; B: UGE2; 1:中间片段; 2: 3′ RACE; 3: 5′ RACE; 4: ORF; M: 2 000 bp DNA Ladder Marker。 Fig. 2 PCR of LcUGEs. A: UGE1; B: UGE2; 1: Fragment; 2: 3′ RACE; 3: 5′ RACE; 4: ORF; M: 2 000 bp DNA Ladder Marker. |
ORF Finder预测LcUGE1开放阅读框(ORF)长度为1 050 bp,LcUGE2基因的为1 056 bp,通过设计引物扩增ORF区,经测序得到的序列与预测的序列比对一致。
2.3 LcUGEs基因gDNA序列克隆和结构分析采用EPIC-PCR技术扩增LcUGEs内含子, 用Seqman 2软件对序列重叠区域拼接,获得基因组序列。结果表明,LcUGE1和LcUGE2基因的gDNA序列长度差异较大,LcUGE1长度为11 920 bp,而LcUGE2长度仅为6 544 bp,LcUGE1的cDNA和gDNA的G+C含量分别为45.4%和38.7%;LcUGE2分别为44.8%和39.1%。采用GeneStructure Display Server在线软件(http://gsds.cbi.pku.edu.cn/)分析Lc-UGE1和LcUGE2基因的gDNA和cDNA序列(图 3),可见两个基因均含有9个外显子和8个内含子,且每个外显子长度几乎一致,说明两个LcUGE基因的长度差异来自于内含子。

|
图 3 鹅掌楸LcUGE1和LcUGE2基因结构 Fig. 3 Structure of LcUGE1 and LcUGE2 in Liriodendron chinense |
采用Splign软件对LcUGE1和LcUGE2基因结构进行分析(表 4),可见内含子差异主要来源于内含子1、2、5、6,其中LcUGE1基因的内含子1、2、6长度远大于LcUGE2,仅第5个内含子长度比LcUGE2小。内含子剪切识别位点分析表明,除LcUGE1内含子2为AG/TG类型之外,其余剪切识别位点均遵循AG/GT剪切原则,这与拟南芥、水稻等UGE基因剪切识别位点一致,表明不同物种间共用一套剪切体。
| 表 4 LcUGEs基因结构 Table 4 Structure of LcUGE genes |
理化性质 LcUGE1基因编码349个氨基酸, LcUGE1分子式为C1714H2675N459O508S12,包含5 368个原子, 分子质量为38.2 kD,带正电荷(Asp+Glu)数目41,带负电荷(Arg+Lys)数目38,等电点为6.19, 属于酸性蛋白。LcUGE1所含的甘氨酸(Gly)最多, 达9.7%;其次是异亮氨酸(Leu, 8.3%);色氨酸(Trp)最少(1.4%);无吡咯赖氨酸(Pyl)和硒半胱氨酸(Sec)。不稳定系数是27.80,为稳定蛋白。疏水性平均值为-0.154,是亲水蛋白。LcUGE2基因编码351个氨基酸,LcUGE2分子式是C1735H2713N471O515S13, 包含5 447个原子,分子质量为38.8 kD,带正电荷(Asp+Glu)数目43,负电荷(Arg+Lys)数目39,等电点为6.02,属于酸性蛋白。LcUGE2的甘氨酸(Gly)最多,占9.1%,其次是异亮氨酸(Leu, 8.5%),最少是色氨酸(Trp, 1.4%)。无吡咯赖氨酸(Pyl)和硒半胱氨酸(Sec)。不稳定系数是27.81,为稳定蛋白。疏水性平均值为-0.253,是亲水蛋白。
亚细胞定位预测 用在线软件TargetP 1.1 Server (http://www.cbs.dtu.dk/services/TargetP/)分析蛋白质亚细胞定位,预测LcUGE1和LcUGE2的定位为除线粒体和叶绿体之外的其他位置,预测的可信度分别为4和2,较为可信,初步认为其蛋白质定位于除线粒体和叶绿体的其它位置。
系统发育分析 将LcUGE1和LcUGE2与其他物种进行BLASTP同源性比对,用UPGMA法构建系统进化树。从图 4可见,LcUGE1与罂粟科(Papaveraceae)博落回(Macleaya cordata)的McUGE亲缘关系较近,LcUGE2与豆科(Leguminosae)豌豆(Pisum sativum)的PsUGE1亲缘关系较近。
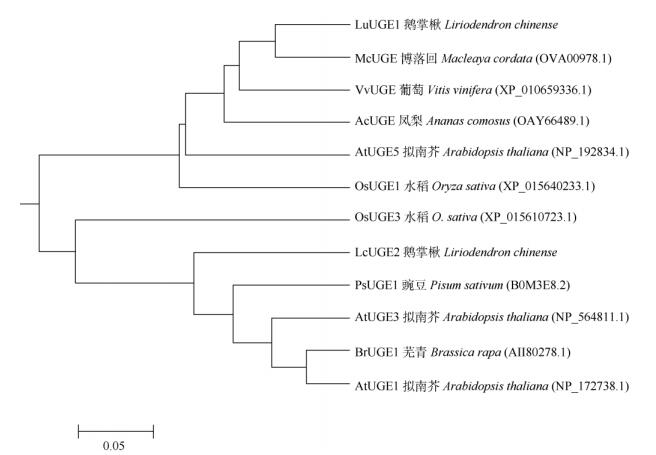
|
图 4 LcUGE1和LcUGE2与其他植物UGE的系统进化树 Fig. 4 Phylogenetic tree of LcUGE1 and LcUGE2 with UGE in other plants |
蛋白质二级结构预测 采用在线SOPMA软件预测,LcUGE1的二级结构由α螺旋(31.52%)、β转角(11.17%)、无规则卷曲(36.96%)和延伸链(20.34%)构成, α螺旋是该蛋白质二级结构的主要成分。LcUGE2的二级结构中α螺旋占40.46%,β转角占10.54%,无规则卷曲占33.05%,延伸链占15.95%。
蛋白质同源性分析 通过InterProScan在线工具预测蛋白质保守结构域,结果表明,在LcUGE1和LcUGE2的N端有UGE家族最典型的NADPH结合域(GXXGXXG),还有1个三联体催化保守域(YXXXK),C端结构不同。用NCBI搜索与LcUGE1和LcUGE2高度同源的蛋白质,并用DNAMAN软件对氨基酸序列进行多重比对,结果表明,LcUGE1和LcUGE2与博落回的McUGE、可可树(Theobroma cacao)的TcUGE、豌豆的PsUGE1、Anthurium amnicola的AaUGE1和瓜儿豆(Cyamopsis tetrago- noloba)的CtUGE均含有同样的保守结构域,且相似度为82.05%。
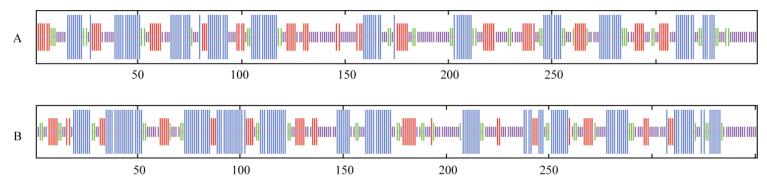
|
图 5 LcUGE的二级结构预测。A: LcUGE1; B: LcUGE2。 Fig. 5 Secondary structure prediction of LcUGEs. A: LcUGE1; B: LcUGE2. |

|
图 6 UGE的氨基酸序列比对。Lc:鹅掌楸; Mc:博落回; Tc:可可树; Ps:豌豆; Aa: Anthurium amnicola; Ct:瓜儿豆。 Fig. 6 Alignment of amino acid sequences of UGEs. Lc: Liriodendron chinense; Mc: Macleaya cordata; Tc: Theobroma cacao; Ps: Pisum sativum; Aa: Anthurium amnicola; Ct: Cyamopsis tetragonoloba. |
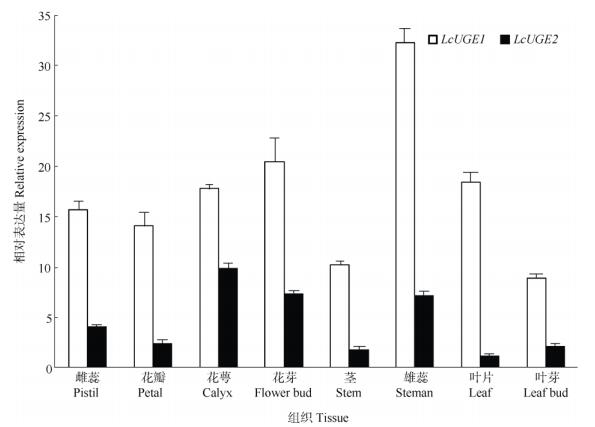
分别提取鹅掌楸茎、叶、花瓣、花萼、雄蕊、雌蕊、花芽和叶芽8个组织的RNA,经反转录后用qTR-PCR方法分析LcUGE的组织表达模式。结果表明,LcUGE1与LcUGE2在8个组织中均有表达,LcUGE1的相对表达量以雄蕊 > 花芽 > 叶 > 花萼> 雌蕊 > 花瓣 > 茎 > 叶芽,其中在雄蕊的相对表达量显著高于其他组织;LcUGE2的相对表达量以花萼> 花芽 > 雄蕊 > 雌蕊 > 花瓣 > 叶芽 > 茎 > 叶片,其中在花萼、雄蕊、花芽的相对表达量显著高于其他组织。这表明LcUGEs基因在生殖器官中的表达量显著高于营养器官,代谢旺盛器官高于代谢缓慢的器官,说明LcUGEs基因很可能为生长发育相关的组成型基因。
|
图 7 鹅掌楸LcUGE1与LcUGE2基因的组织表达 Fig. 7 Expression of LcUGE1and LcUGE2 in tissues of Liriodendron chinense |
本研究从鹅掌楸中克隆得到了2个LcUGE基因,并对其编码的蛋白质进行了生物信息学分析, 同源序列比对表明LcUGEs蛋白有UGE家族典型的结构域。系统进化分析表明LcUGE1与博落回的McUGE亲缘关系最近,LcUGE2与豌豆的PsUGE1亲缘关系最近;同时,LcUGE1与拟南芥的AtUGE5聚为一类,LcUGE2与拟南芥的AtUGE3聚为一类,这可能与UGE在不同种间的进化差异有关。
基因结构和内含子研究是进化生物学研究的热点,本研究采用EPIC-PCR技术从鹅掌楸基因组中扩增出LcUGEs的gDNA,与拟南芥等物种的UGE基因结构相似,均含有9个外显子和8个内含子。但LcUGE1基因内含子1与内含子2的长度显著大于LcUGE2基因的,LcUGE1的gDNA长度11.9 kb,约为LcUGE2的gDNA长度的2倍,而他们的外显子长度相近,长度差异主要集中在第1、2、5和6个内含子上,因此推测该家族基因在经历基因家族扩张后,内含子区域出现过大片段插入/缺失,而外显子区域由于受到强烈的纯化选择作用, 并在进化过程中固定下来,因此外显子序列长度和编码蛋白保持相对稳定。
本研究预测鹅掌楸LcUGEs蛋白定位于除线粒体和叶绿体以外的其他位置。曾有研究通过核磁共振观察到大多数细胞中的UDP-半乳糖和UDP-葡萄糖都定位在细胞质中[25],有报道UDP-半乳糖和UDP-葡萄糖可用来合成高尔基体中非纤维素的多糖和糖蛋白,并且由特定的转运蛋白将核糖从细胞质转移到高尔基体腔囊泡中[26]。从拟南芥中已经克隆了这种核糖转运基因,基因产物会将UDP-半乳糖和UDP-葡萄糖转运到高尔基体中[27]。对于鹅掌楸中是否也存在这种转运蛋白还不确定,LcUGE1与LcUGE2的亚细胞定位还需要进一步研究。组织特异性表达表明,LcUGE1和LcUGE2在8个组织中均有表达,LcUGE1在雄蕊中相对表达量最丰富,其次是花芽,在叶芽的表达量最少,而LcUGE2在花萼、雄蕊和花瓣中相对表达量均显著高于其他组织,这与拟南芥AtUGE5和芜菁(Brassica rapa) Br- UGE5在花器官中的表达类似,可能与其在花器官中的功能有关[28]。鹅掌楸LcUGEs在生殖器官中的表达量最高,生殖器官是植物代谢最旺盛的器官之一,推测LcUGE1基因为与代谢密切相关的组成型基因,除参与细胞壁的合成过程外,可能还与生殖器官发育有关。
有研究表明,拟南芥的5个UGE都能够提高半乳糖耐受性,AtUGE2和AtUGE4基因会影响植株的生长发育和细胞壁糖类物质的合成,AtUGE1和AtUGE5基因可能会参与胁迫应答机制中[11]。这表明UGE基因不仅在半乳糖代谢和碳水化合物合成方面有着重要的作用,还可能增加植株忍受非生物胁迫的能力。本研究获得鹅掌楸LcUGE1和Lc- UGE2基因的全长序列,并对基因断裂结构进行了初步分析,为后续LcUGE基因的调控表达元件和功能研究提供参考。
| [1] | REITER W D, VANZIN G F. Molecular genetics of nucleotide sugar interconversion pathways in plants[J]. Plant Mol Biol, 2001, 47(1/2): 95-113. DOI:10.1023/A:1010671129803 |
| [2] | Ross K L, Davis C N, Fridovich-Keil J L. Differential roles of the Leloir pathway enzymes and metabolites in defining galactose sensitivity in yeast[J]. Mol Genet Met, 2004, 83(1/2): 103-116. DOI:10.1016/j.ymgme.2004.07.005 |
| [3] | HOLDEN H M, RAYMENT I, THODEN J B. Structure and function of enzymes of the Leloir pathway for galactose metabolism[J]. J Biol Chem, 2003, 278(45): 43885-43888. DOI:10.1074/jbc.R300025200 |
| [4] | LIU H L, DAI X Y, XU Y Y, et al. Over-expression of OsUGE-1 altered raffinose level and tolerance to abiotic stress but not morphology in Arabidopsis[J]. J Plant Physiol, 2007, 164(10): 1384-1390. DOI:10.1016/j.jplph.2007.03.005 |
| [5] | KLECZKOWSKI L A, KUNZ S, WILCZYNSKA M. Mechanisms of UDP-glucose synthesis in plants[J]. Crit Rev Plant Sci, 2010, 29(4): 191-203. DOI:10.1080/07352689.2010.483578 |
| [6] | SEIFERT G J, BARBER C, WELLS B, et al. Growth regulators and the control of nucleotide sugar flux[J]. Plant Cell, 2004, 16(3): 723-730. DOI:10.1105/tpc.019661 |
| [7] | BARBER C, RÖSTI J, RAWAT A, et al. Distinct properties of the five UDP-d-glucose/UDP-d-galactose 4-epimerase isoforms of Arabidopsis thaliana[J]. J Biol Chem, 2006, 281(25): 17276-17285. DOI:10.1074/jbc.M512727200 |
| [8] | ZHANG Q S, HRMOVA M, SHIRLEY N J, et al. Gene expression patterns and catalytic properties of UDP-d-glucose 4-epimerases from barley (Hordeum vulgare L.)[J]. Biochem J, 2006, 394(Pt 1): 115-124. DOI:10.1042/BJ20051329 |
| [9] | KIM S K, KIM D H, KIM B G, et al. Cloning and characterization of the UDP glucose/galactose epimerases of Oryza sativa[J]. J Korean Soc Appl Biol Chem, 2009, 52(4): 315-320. DOI:10.3839/jksabc.2009.056 |
| [10] | SUN H Y, LI L C, LOU Y F, et al. Cloning and preliminary functional analysis of PeUGE gene from moso bamboo (Phyllostachys edulis)[J]. DNA Cell Biol, 2016, 35(11): 706-714. DOI:10.1089/dna.2016.3389 |
| [11] | RÖSTI J, BARTON C J, ALBRECHT S, et al. UDP-glucose 4-epimerase isoforms UGE2 and UGE4 cooperate in providing UDP-galactose for cell wall biosynthesis and growth of Arabidopsis thaliana[J]. Plant Cell, 2007, 19(5): 1565-1579. DOI:10.1105/tpc.106.049619 |
| [12] | SOLDO B, SCOTTI C, KARAMATA D, et al. The Bacillus subtilis Gne (GneA, GalE) protein can catalyse UDP-glucose as well as UDP-N-acetylglucosamine 4-epimerisation[J]. Gene, 2003, 319: 65-69. DOI:10.1016/S0378-1119(03)00793-5 |
| [13] | GUEVARA D R, EL-KEREAMY A, YAISH M W, et al. Functional characterization of the rice UDP-glucose 4-epimerase 1, OsUGE1:A potential role in cell wall carbohydrate partitioning during limiting nitrogen conditions[J]. PLoS One, 2014, 9(5): e96158 DOI:10.1371/journal.pone.0096158 |
| [14] |
WANG Z R.
Utilization and Species Hybridization in Liriodendron[M]. Beijing: China Forestry Publishing House, 2005: 15-27.
王章荣. 鹅掌楸属树种杂交育种与利用[M]. 北京: 中国林业出版社, 2005: 15-27. |
| [15] |
LI H G, CHEN L, LIANG C Y, et al. A case study on provenance testing of tulip tree (Liriodendron spp.)[J].
China For Sci Technol, 2005, 19(5): 13-16. 李火根, 陈龙, 梁呈元, 等. 鹅掌楸属树种种源试验研究[J]. 林业科技开发, 2005, 19(5): 13-16. DOI:10.3969/j.issn.1000-8101.2005.05.005 |
| [16] |
CHENG Y L, ZHONG W P, HAO Z Y, et al. Cloning and expression analysis of CCD1 gene in Liriodendron tulipifera[J].
Mol Plant Breed, 2017, 15(6): 2139-2146. 成彦丽, 仲维平, 郝自远, 等. 北美鹅掌楸CCD1基因的克隆与表达分析[J]. 分子植物育种, 2017, 15(6): 2139-2146. DOI:10.13271/j.mpb.015.002139 |
| [17] | TAMURA K, STECHER G, PETERSON D, et al. MEGA6:Molecular evolutionary genetics analysis, Version 6.0[J]. Mol Biol Evol, 2013, 30(12): 2725-2729. DOI:10.1093/molbev/mst197 |
| [18] | BERREBI P, RETIF X, FANG F, et al. Population structure and syste-matics of Opsariichthys bidens (Osteichthyes:Cyprinidae) in south-east China using a new nuclear marker:The introns (EPIC-PCR)[J]. Biol J Linn Soc Lond, 2006, 87(1): 155-166. DOI:10.1111/j.1095-8312.2006.00563.x |
| [19] | TAY W T, BEHERE G T, HECKEL D G, et al. Exon-primed intron-crossing (EPIC) PCR markers of Helicoverpa armigera (Lepidoptera:Noctuidae)[J]. Bull Entomol Res, 2008, 98(5): 509-518. DOI:10.1017/S000748530800583X |
| [20] | LESSA E P. Rapid surveying of DNA sequence variation in natural populations[J]. Mol Biol Evol, 1992, 9(2): 323-330. DOI:10.1093/oxfordjournals.molbev.a040723 |
| [21] | PALUMBI S R, BAKER C S. Contrasting population structure from nuclear intron sequences and mtDNA of humpback whales[J]. Mol Biol Evol, 1994, 11(3): 426-435. DOI:10.1093/oxfordjournals.molbev.a040115 |
| [22] | SLADE R W, MORITZ C, HEIDEMAN A, et al. Rapid assessment of single-copy nuclear DNA variation in diverse species[J]. Mol Ecol, 1993, 2(6): 359-373. DOI:10.1111/j.1365-294X.1993.tb00029.x |
| [23] | LARDEUX F, ALIAGA C, TEJERINA R, et al. Development of exon-primed intron-crossing (EPIC) PCR primers for the malaria vector Anopheles pseudopunctipennis (Diptera:Culicidae)[J]. C R Biol, 2012, 335(6): 398-405. DOI:10.1016/j.crvi.2012.05.002 |
| [24] |
YANG Y, LI H G. Cloning of NAC gene from Liriodendron tulipifera and its expression analysis[J].
J Plant Res Environ, 2015, 24(3): 1-9. 杨颖, 李火根. 北美鹅掌楸NAC基因的克隆与表达分析[J]. 植物资源与环境学报, 2015, 24(3): 1-9. DOI:10.3969/j.issn.1674-7895.2015.03.01 |
| [25] | BLIGNY R, GARDESTROM P, ROBY C, et al. 31P NMR studies of spinach leaves and their chloroplasts[J]. J Biol Chem, 1990, 265(3): 1319-1326. |
| [26] | MOORE P J, SWORDS K M, LYNCH M A, et al. Spatial organization of the assembly pathways of glycoproteins and complex polysaccha-rides in the golgi apparatus of plants[J]. J Cell Biol, 1991, 112(4): 589-602. DOI:10.1083/jcb.112.4.589 |
| [27] | NORAMBUENA L, MARCHANT L, BERNINSONE P, et al. Transport of UDP-galactose in plants:Identification and functional characterization of AtUTr1, an Arabidopsis thaliana UDP-galactos/UDP-glucose transporter[J]. J Biol Chem, 2002, 277(36): 32923-32929. DOI:10.1074/jbc.M204081200 |
| [28] | JUNG Y J, KYOUNG J H, NOU I S, et al. Molecular characterization of the UDP-glucose 4-epimerase (BrUGE) gene family in response to biotic and abiotic stress in Chinese cabbage (Brassica rapa)[J]. Plant Biotechnol Rep, 2015, 9(6): 339-350. DOI:10.1007/s11816-015-0370-7 |
 2018, Vol. 26
2018, Vol. 26



