2. 福建农林大学园艺植物生物工程研究所, 福州 350002
2. Institute of Horticultural Biotechnology, Fujian Agriculture and Forestry University, Fuzhou 350002, China
橄榄[Canarium album (Lour.) Raeusch.]又称青果,为橄榄科(Burseraceae)橄榄属常绿乔木果树, 是我国热带亚热带地区特色名贵经济作物,在福建、广东、广西、云南、海南和台湾等南部地区均有种植。橄榄果实营养价值高,富含多酚、多糖、氨基酸、膳食纤维和矿质元素等多种药理活性物质,是广受欢迎的药食同源作物。然而,橄榄抗寒能力差,气温低于-2℃时叶片开始受冻,低于-2.7℃时末一级枝条受冻,而低于-3℃时末二级枝条受冻,因此通常认为-3℃是橄榄所能承受的极限临界低温[1-2]。在我国南部橄榄种植区,尤其在福建省闽侯、闽清、尤溪、连江和上杭等地区均发生过橄榄大规模冻害,导致产量和种植面积锐减,成为制约橄榄产业稳定发展的重要限制因素[3]。
CBF信号转导途径是植物感知和响应外界低温环境的关键信号通路, 当植物遭遇低温胁迫时, ICE1转录因子能够结合在CBF3启动子特定区域, 诱导CBF3大量表达,进而激活下游的抗寒关键基因COR上调表达以提高植物抗寒能力,在植物冷驯化和低温胁迫响应过程中发挥重要作用[4-6]。此外,ICE1也是HOS1 (high expression of osmotically responsive gene 1)[7]、SIZ1 (SAP and Miz 1)[8]、OST1 (open stomata 1)[9-10]、MAPK (mitogen-activated protein kinase)[11-12]和精氨酸脱羧酶(arginine decarboxylase)[13]等参与调节植物抗寒能力的桥梁,是植物抗寒调控网络的关键转录因子之一。目前,ICE1转录因子在白菜(Brassica campestris)[14]、秋子梨(Pyrus ussuri- ensis)[15]、番茄(Lycopersicon esculentum)[16]和葡萄(Vitis amurensis)[17]等作物抗寒过程中的调控功能和作用机理已有大量研究,而在橄榄上未见报道。本文以‘福榄1号’橄榄为材料,对其ICE1转录因子进行基因克隆和生物信息学分析,同时分析了CaICE1在组织器官和低温胁迫下的表达模式,为橄榄抗寒分子育种提供科学依据。
1 材料和方法 1.1 材料以福建省农业科学院果树研究所选育的橄榄鲜食品种‘福榄1号’[Canarium album (Lour.) Raeusch. ‘Fulan-1’]为材料。于2017年3月播种,同年7月选取长势健壮一致且无病虫害的幼苗分别置于-3℃、4℃和25℃下处理24 h,采集幼苗第3和4叶,置于液氮中速冻,保存于-80℃超低温冰箱中备用,每个处理重复3次。于2016年11月采集15年生‘福榄1号’橄榄成熟果肉和种子,于2017年5月上旬采集同一植株健康无损伤的根、茎、叶和花,液氮速冻后超低温保存备用。
1.2 CaICE1克隆采用植物多糖多酚试剂盒E.Z.N.A.TM Plant RNA Kit (OMEGA bio-tek)分别提取橄榄根、茎、叶、花、果实和种子的总RNA,经琼脂糖凝胶电泳和紫外分光光度计检测质量和浓度。将不同组织器官的总RNA等量混合,用Thermo Scientific RevertAid First Strand cDNA Synthesis Kit逆转录cDNA用于PCR扩增。以橄榄转录组数据为参考,通过序列预测和NCBI BLAST序列比对,选取CaICE1转录因子可能的cDNA序列,采用DNAMAN设计引物, 上游引物CaICE1-F:5′-CTCTCTCTTTTCCCAACAA- ACT-3′,下游引物CaICE1-R:5′-TCTACATCATGC- CATGGAAG-3′。PCR扩增体系包含2×TransTaq High Fidelity PCR SuperMix 12.5 μL,cDNA模板50 ng μL–1,上下游引物各0.4 μmol L–1,补足无菌ddH2O至终体积25 μL。PCR反应程序为:预变性95℃ 4 min,变性95℃ 30 s,退火55℃ 30 s,延伸72℃ 75 s,共35个循环,延伸72℃ 10 min。扩增结束后,利用1.5%琼脂糖凝胶电泳进行目的条带检测,采用EasyPure® Quick Gel Extraction Kit进行胶回收,并将目的条带连接pEASY® -T5 Zero Cloning Kit转化到Trans1-T1感受态细胞中,置于37℃恒温培养箱中培养12 h后挑取单克隆子进行PCR检测,选取阳性克隆子送北京六合华大基因科技有限公司进行测序。
1.3 CaICE1生物信息学分析采用生物信息学软件(表 1)对CaICE1转录因子的基本理化性质、结构、亲缘关系、密码子偏好性和调控miRNA等进行预测和分析。
| 表 1 CaICE1转录因子生物信息学分析 Table 1 Bioinformatic analysis of CaICE1 transcription factor |
分别提取不同温度处理后的橄榄的总RNA,检测合格后经TransScript All-in-One First-Strand cDNA Synthesis SuperMix for qPCR (One-Step gDNA Removel; TransGen Biotech, Beijing)逆转录cDNA后用于qRT-PCR试验。以橄榄ACTB7和TUB5为内参基因[18],采用TransStart® Top Green qPCR Super- Mix试剂盒对CaICE1的表达模式进行检测。将橄榄不同处理的cDNA样品进行等量混合,随后进行×10、×40、×160和×640梯度稀释用于绘制标准曲线,根据扩增效率筛选合适的退火温度。采用Eppendorf Realplex4荧光定量PCR仪检测样品中CaICE1的相对表达量。qRT-PCR扩增程序为:94℃预变性30 s;94℃变性10 s,退火15 s,72℃延伸10 s,循环40次;94℃保持15 s,60℃保持15 s, 再以0.11 ℃ s–1速度升温至94℃保持15 s,绘制融解曲线。qRT-PCR反应体系为:2×TransStart® Top Green qPCR SuperMix 10 μL,Passive Reference Dye (50×) 0.4 μL,上下游引物各0.2 μmol L–1,模板0.5 μmol L–1, 引物序列为CaICE1-qF: 5′-GGATGGAAGACAAA- GACGATG-3′和CaICE1-qR: 5′-TGTTGCTGCTGT- TGATGATG-3′。采用Excel 2003和SPSS 19.0进行数据统计和差异显著性分析。
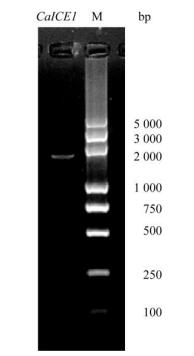
2 结果和分析 2.1 CaICE1的克隆根据橄榄转录组数据库筛选橄榄ICE1 cDNA序列并设计引物,使用RT-PCR法克隆CaICE1序列并进行测序验证。获得的cDNA序列为1 743 bp,其中开放阅读框(open reading frame,ORF)长度为1 650 bp,编码549个氨基酸,起始密码子和终止密码子分别为ATG和TAG (图 1, 2)。通过NCBI比对,CaICE1与枳(Citrus trifoliata)的ICE1氨基酸序列相似度达71%,表明所获得的序列为橄榄CaICE1基因,GenBank登录号为MG459422。
|
图 1 橄榄CaICE1的扩增。M: 5 000 bp Plus DNA Marker。 Fig. 1 Amplification of CaICE1. M: 5 000 bp Plus DNA Marker. |

|
图 2 CaICE1的cDNA序列和推测的氨基酸序列。阴影和下划线分别表示HLH结构域和ACT_UUR-ACR-like。 Fig. 2 Sequences of CaICE1 and encoding amino acids. The shade and underline indicate HLH domain and ACT_UUR-ACR-like, respectively. |
生物信息学分析结果表明,CaICE1有549个氨基酸,分子式为C2588H4115N755O850S27,原子总数和分子量分别为8.335和60.272 kD,pI等电点为5.29,不稳定系数、脂肪系数和亲水性系数分别为48.62、73.15和-0.581,说明CaICE1为不稳定亲水酸性蛋白质,其中亮氨酸(Leu)和丝氨酸(Ser)含量最多,均占总数的10.4%。进一步分析表明,CaICE1可能定位于细胞核,不含信号肽,含有56个磷酸化位点(酪氨酸、苏氨酸和丝氨酸磷酸化位点分别为3、14和39个),此外含有2个由内向外和1个由外向内的跨膜螺旋,推测为跨膜蛋白。保守结构域预测表明,CaICE1包含有ICE1蛋白家族保守的螺旋-环-螺旋结构域(helix loop helix domain, HLH)和ACT保守域,能够特异结合具有调控酶活性的氨基酸或小配体(图 2)。此外,CaICE1存在α-螺旋、β-折叠和无规则卷曲3种二级结构,比重分别占12.39%、6.01%和81.60%。SWISS-MODEL预测表明CaICE1三维结构与PDB5gnj.1.B相似度达到48.44% (图 3)。采用MEGA 5.2.2基于邻近归并法对物种间的ICE1进行聚类分析,除番木瓜(Carica papaya)和核桃(Juglans regia)外,总体上物种间的ICE1可分为4个大类(图 4), 其中橄榄的CaICE1与枳(Citrus trifo- liata)的ICE1亲缘关系最近,这说明物种间的ICE1存在多样性。

|
图 3 CaICE1的三维结构 Fig. 3 Three-dimensional structure of CaICE1 |

|
图 4 ICE1的系统发育树 Fig. 4 Phylogenetic tree of ICE1 |
采用psRNATarget预测可能靶定CaICE1的miRNA,结果表明CaICE1可能与20个miRNA存在靶向关系,其中miR2938、miR6464、miR5537和miR437b的数学期望值为3.0,UPE均低于20, 是最可能调控CaICE1的miRNA (表 2)。从作用方式上看,这些miRNA对CaICE1的调控方式可能主要包括裂解和翻译抑制2类(均为10个)。
| 表 2 预测获得的靶向CaICE1的miRNA Table 2 Predicted miRNAs targeting CaICE1 |
密码子偏好性分析表明,CaICE1有效密码子数(ENc)和密码子适应指数(CAI)分别为51.54和0.203, 表明其密码子偏好性较弱。密码子第三位GC含量和总GC含量分别为0.440和0.445,说明其偏好使用含有A和T并以A或T结尾的密码子。同义密码子相对使用度分析表明,CaICE1偏好性较强的密码子(RSCU≥2.0)仅有4个,分别为AGA、AGG、TTG和CCA,可能作为其最优密码子群,也进一步验证CaICE1密码子偏好性较弱(表 3)。
| 表 3 CaICE1同义密码子相对使用度 Table 3 Relative synonymous codon usage of CaICE1 |
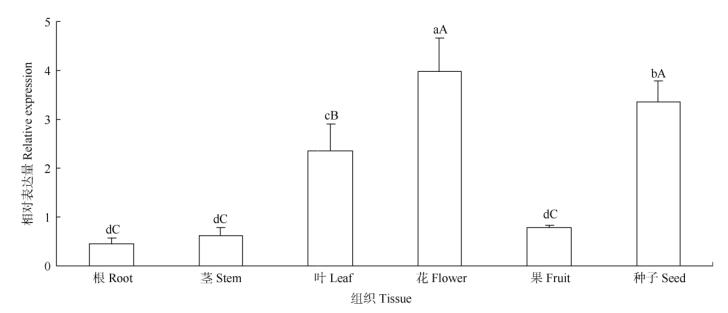
CaICE1在橄榄各组织器官中均有表达,以花中的表达量最高,其次是种子和叶,而在根、茎和果实中的表达量相对较低(图 5)。此外,差异显著性分析表明,CaICE1的表达存在组织器官特异性表达,暗示其可能在不同组织器官抗寒过程中发挥调控作用。
|
图 5 CaICE1在组织中的表达。柱上不同大写和小写字母分别表示差异极显著(P < 0.01)和显著(P < 0.05)。下同。 Fig. 5 Relative expression of CaICE1 in tissues. Different capital and small letters upon column indicate significant differences at 0.01 and 0.05 levels, respectively. The same is following Figure. |
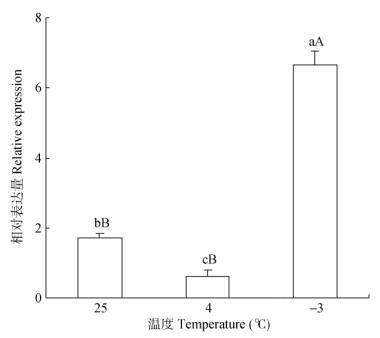
qRT-PCR结果表明,与25℃对照相比,4℃处理的CaICE1表达出现稍微下调,说明4℃低温处理时CaICE1并未被激活,这可能与植株并未受到显著寒害有关。而-3℃处理24 h后,CaICE1的表达水平急剧上升,为对照的3.8倍,说明-3℃低温冻害后橄榄CaICE1基因被诱导显著表达,从而迅速激活其介导的抗寒系统以维持机体正常生长代谢。
3 讨论 3.1 CaICE1参与橄榄低温胁迫响应过程植物ICE1是一类含有bHLH保守结构域的MYC组成型表达基因,通常在细胞中处于无活性状态,低温活化后能够与CBF启动子特定区域结合并以‘主效开关’的身份激活CBF信号途径[19]。在拟南芥(Arabidopsis thaliana)中,约40%的抗寒关键基因和46%的冷响应基因均受ICE1调控,说明ICE1在植物抗寒过程中发挥至关重要的作用[8, 20]。本研究结果表明,CaICE1含有植物典型的HLH保守结构域,且与其他物种的ICE1均保持较近的亲缘关系,说明其为橄榄的抗寒调控转录因子。生物信息学分析表明,CaICE1可能受一系列miRNA转录后水平或翻译水平抑制调控,其中已有报道miR825[21]、miR477[22]、miR5658[23]、miR1436[24]和miR394[25]等均为植物逆境胁迫响应miRNA,尤其miR394直接参与了拟南芥冷胁迫应答,可见CaICE1可能与橄榄逆境胁迫密切相关。进一步的qRT-PCR分析表明,-3℃处理后CaICE1的表达量急剧上升,说明冻害后CaICE1介导的抗寒调控网络迅速启动以维持橄榄正常生理代谢。此外,橄榄CaICE1在不同组织器官中均维持一定的表达水平以随时应对外界低温胁迫。由此看来,CaICE1可能在橄榄低温胁迫过程中发挥了重要的调控作用。
|
图 6 CaICE1在低温胁迫下的表达 Fig. 6 Expression of CaICE1 under low temperature stress |
物种或基因在长期进化过程中往往形成特定的密码子偏好性规律,密码子偏好性分析可为揭示基因的分子功能和进化规律提供科学依据[26-27]。本研究中,橄榄CaICE1的密码子偏好性较弱,但偏好使用含有A和T并以A或T结尾的密码子,这与对茶树(Camellia sinensis) ICE1[28]的研究结果一致,且符合双子叶植物[29]基本特征,但橄榄CaICE1密码子的具体进化过程、进化方式和关键作用力等仍需进一步研究。
| [1] |
LAI Z X, CAI Y Q, XU W D, et al.
Quality Cultivating and Integrated Utilization of Chinese Olive[M]. Beijing: Chinese Sanxia Press, 2007: 14-17.
赖钟雄, 蔡英卿, 许伟东, 等. 橄榄优质栽培与综合利用[M]. 北京: 中国三峡出版社, 2007: 14-17. |
| [2] |
CAI W H, CHEN H, LI W, et al. Investigation of low air temperature and preliminary study of indexes of olive freeze harm in the winter of 2004/2005 in Lianjiang County[J].
Chin J Agrometeorol, 2006, 27(3): 200-203. 蔡文华, 陈惠, 李文, 等. 2004/2005年冬季连江县低温考察和橄榄树冻害指标初探[J]. 中国农业气象, 2006, 27(3): 200-203. DOI:10.3969/j.issn.1000-6362.2006.03.010 |
| [3] |
LAI R L, CHEN J, FENG X, et al. Advances on germplasm resource evaluation and cold resistance research in Chinese olive of China[J].
Chin J Trop Crop, 2017, 38(11): 2188-2194. 赖瑞联, 陈瑾, 冯新, 等. 中国橄榄种质资源评价与抗寒性研究进展[J]. 热带作物学报, 2017, 38(11): 2188-2194. DOI:10.3969/j.issn.1000-2561.2017.11.031 |
| [4] | THOMASHOW M F. Plant cold acclimation:Freezing tolerance genes and regulatory mechanisms[J]. Annu Rev Plant Physiol Plant Mol Biol, 1999, 50: 571-599. DOI:10.1146/annurev.arplant.50.1.571 |
| [5] | KIM Y S, LEE M, LEE J H, et al. The unified ICE-CBF pathway provides a transcriptional feedback control of freezing tolerance during cold acclimation in Arabidopsis[J]. Plant Mol Biol, 2015, 89(1/2): 187-201. DOI:10.1007/s11103-015-0365-3 |
| [6] | WANG D Z, JIN Y N, DING X H, et al. Gene regulation and signal transduction in the ICE-CBF-COR signaling pathway during cold stress in plants[J]. Biochemistry, 2017, 82(10): 1103-1117. DOI:10.1134/S0006297917100030 |
| [7] | DONG C H, AGARWAL M, ZHANG Y Y, et al. The negative regulator of plant cold responses, HOS1, is a RING E3 ligase that mediates the ubiquitination and degradation of ICE1[J]. Proc Natl Acad Sci USA, 2006, 103(21): 8281-8286. DOI:10.1073/pnas.0602874103 |
| [8] | MIURA K, JIN J B, LEE J, et al. SIZ1-mediated sumoylation of ICE1 controls CBF3/DREB1A expression and freezing tolerance in Arabidopsis[J]. Plant Cell, 2007, 19(4): 1403-1414. DOI:10.1105/tpc.106.048397 |
| [9] | DING Y L, LI H, ZHANG X Y, et al. OST1 kinase modulates freezing tolerance by enhancing ICE1 stability in Arabidopsis[J]. Dev Cell, 2015, 32(3): 278-289. DOI:10.1016/j.devcel.2014.12.023 |
| [10] | LANG Z B, ZHU J K. OST1 phosphorylates ICE1 to enhance plant cold tolerance[J]. Sci China Life Sci, 2015, 58(3): 317-318. DOI:10.1007/s11427-015-4822-7 |
| [11] | ZHAO C Z, WANG P C, SI T, et al. MAP kinase cascades regulate the cold response by modulating ICE1 protein stability[J]. Dev Cell, 2017, 43(5): 618-629. DOI:10.1016/j.devcel.2017.09.024 |
| [12] | LIU Y K, ZHOU J. MAPping kinase regulation of ICE1 in freezing tolerance[J]. Trends Plant Sci, 2018, 23(2): 91-93. DOI:10.1016/j.tplants.2017.12.002 |
| [13] | HUANG X S, ZHANG Q, ZHU D, et al. ICE1 of Poncirus trifoliata functions in cold tolerance by modulating polyamine levels through interacting with arginine decarboxylase[J]. J Exp Bot, 2015, 66(11): 3259-3274. DOI:10.1093/jxb/erv138 |
| [14] | XIANG D J, CHAI Y S, MAN L L, et al. Overexpression of a heading Chinese cabbage ICE1 gene confers freezing tolerance in transgenic rice[J]. Plant Cell Tiss Org Cult, 2017, 128(1): 43-54. DOI:10.1007/s11240-016-1080-8 |
| [15] | HUANG X S, LI K Q, JIN C, et al. ICE1 of Pyrus ussuriensis functions in cold tolerance by enhancing PuDREB a transcriptional levels through interacting with PuHHP1[J]. Sci Rep, 2015, 5: 17620 DOI:10.1038/srep17620 |
| [16] | YU X H, JUAN J X, GAO Z L, et al. Cloning and transformation of inducer of CBF expression1(ICE1) in tomato[J]. Genet Mol Res, 2015, 14(4): 13131-13143. DOI:10.4238/2015 |
| [17] | DONG C, ZHANG Z, REN J P, et al. Stress-responsive gene ICE1 from Vitis amurensis increases cold tolerance in tobacco[J]. Plant Physiol Bioch, 2013, 71: 212-217. DOI:10.1016/j.plaphy.2013.07.012 |
| [18] |
HUANG M J, WEN Z F, CHI Y B, et al. Molecular cloning and expression analysis of flavonoids 3'-hydroxylase (CaF3'H) in Canarium album[J].
Mol Plant Breed, 2017, 15(3): 839-847. 黄敏杰, 文志丰, 池毓斌, 等. 橄榄类黄酮3'-羟化酶CaF3'H基因的克隆及其表达特性分析[J]. 分子植物育种, 2017, 15(3): 839-847. DOI:10.13271/j.mpb.015.000839 |
| [19] |
WEI J Y, ZHAO J, ZHAO S Q, et al. Activation and regulation on the cold response pathway of ICE1-CBF in plants[J].
Biotechnol Bull, 2015, 31(6): 8-12. 魏俊燕, 赵佳, 赵仕琪, 等. 植物ICE1-CBF冷反应通路的激活与调控研究进展[J]. 生物技术通报, 2015, 31(6): 8-12. DOI:10.13560/j.cnki.biotech.bull.1985.2015.06.016 |
| [20] | HAAKE V, COOK D, RIECHMANN J L, et al. Transcription factor CBF4 is a regulator of drought adaptation in Arabidopsis[J]. Plant Physiol, 2002, 130(2): 639-648. DOI:10.1104/pp.006478 |
| [21] | NIU D D, XIA J, JIANG C H, et al. Bacillus cereus AR156 primes induced systemic resistance by suppressing miR825/825* and acti-vating defense-related genes in Arabidopsis[J]. J Integr Plant Biol, 2016, 58(4): 426-439. DOI:10.1111/jipb.12446 |
| [22] |
ZHOU J. Expression profile of miRNA of Populus cathayana Rehd and Salix matsudana Koidz under salt stress[D]. Beijing: Chinese Academy of Forestry, 2010: 38-51.
周婧.盐胁迫条件下青杨和旱柳miRNA表达变化研究[D].北京: 中国林业科学研究院, 2010: 38-51. http://cdmd.cnki.com.cn/Article/CDMD-82201-2010264700.htm |
| [23] | SINGH N, SRIVASTAVA S, SHARMA A. Identification and analysis of miRNAs and their targets in ginger using bioinformatics approach[J]. Gene, 2016, 575(2 Pt 2): 570-576. DOI:10.1016/j.gene.2015.09.036 |
| [24] | MANGRAUTHIA S K, BHOGIREDDY S, AGARWAL S, et al. Genome-wide changes in microRNA expression during short and prolonged heat stress and recovery in contrasting rice cultivars[J]. J Exp Bot, 2017, 68(9): 2399-2412. DOI:10.1093/jxb/erx111 |
| [25] | SONG J B, GAO S, WANG Y, et al. miR394 and its target gene LCR are involved in cold stress response in Arabidopsis[J]. Plant Gene, 2016, 5: 56-64. DOI:10.1016/j.plgene.2015.12.001 |
| [26] |
LAI R L, LIN Y L, ZHONG C S, et al. Analysis of codon bias of auxin receptor gene TIR1 in Dimocarpus longan[J].
Acta Hort Sin, 2016, 43(4): 771-780. 赖瑞联, 林玉玲, 钟春水, 等. 龙眼生长素受体基因TIR1密码子偏好性分析[J]. 园艺学报, 2016, 43(4): 771-780. DOI:10.16420/j.issn.0513-353x.2015-0662 |
| [27] |
LAI R L, FENG X, CHEN J, et al. Codon usage pattern of chalcone isomerase gene (CHI) in Canarium album (Lour.) Raeusch.[J].
Chin J Appl Environ Biol, 2017, 23(5): 945-951. 赖瑞联, 冯新, 陈瑾, 等. 橄榄查尔酮异构酶基因CHI的密码子偏好模式[J]. 应用与环境生物学报, 2017, 23(5): 945-951. DOI:10.3724/SP.J.1145.2016.11037 |
| [28] |
SHI H, WANG Y, YANG L C, et al. Analysis of codon bias of the cold regulated transcription factor ICE1 in tea plant[J].
Acta Hort Sin, 2012, 39(7): 1341-1352. 时慧, 王玉, 杨路成, 等. 茶树抗寒调控转录因子ICE1密码子偏性分析[J]. 园艺学报, 2012, 39(7): 1341-1352. DOI:10.16420/j.issn.0513-353x.2012.07.002 |
| [29] | MURRAY E E, LOTZER J, EBERLE M. Codon usage in plant genes[J]. Nucl Acids Res, 1989, 17(2): 477-498. DOI:10.1093/nar/17.2.477 |
 2018, Vol. 26
2018, Vol. 26



