2. 阜阳职业技术学院生化工程系, 安徽 阜阳 236031
2. Department of Biochemical Engineering, Fuyang Institute of Technology, Fuyang 236031, Anhui, China
随着全球气候变暖,干旱已经成为植物面临的主要非生物胁迫之一。在漫长的进化过程中,植物形成了一套复杂的抗旱机制来应对干旱,包括形态适应、生理生化响应及基因表达调控等[1]。杜鹃(Rhododendron sp.)是中国的十大名花之一,素有“花中西施”的美誉,但由于其对水分比较敏感,再加上城市土壤水分缺失,在园林绿化中经常受到干旱胁迫的影响,进而限制了在园林中的进一步推广, 因此,对于杜鹃花干旱响应机理的研究已显得尤为迫切。目前,对杜鹃花干旱胁迫的研究主要集中在种子萌发[2]和植株生理生化水平的适应性调节[3-5], 而相关分子水平的研究鲜有报道。
植物体内的转录因子(transcription factor, TF)是通过与靶基因启动子区的特定DNA序列(顺式作用元件)结合,从而提高或抑制靶基因转录水平的调控基因[6]。在植物基因组中,大约7%的编码序列被注释为TFs[7],而且常常是以基因家族的形式出现。这些转录因子家族,如MYB[8]、bHLH[9]、NAC[10]、WRKY[11]、bZIP[12]和ERF[13]等对于构建植物应对外界生物或非生物胁迫刺激的转录调控网络至关重要。因此,基因工程中可以运用转录因子来提高植物的抗逆性[14],而挖掘抗逆转录因子则成为了基因工程的基础工作之一。
本研究在杜鹃花转录组测序研究的基础上,深入挖掘杜鹃花应答不同水分条件的转录因子,为阐明杜鹃花抗旱的分子机理及培育抗旱新品种奠定基础。
1 材料和方法 1.1 材料和处理以长势良好的2年生杜鹃花‘白凤4号’ (Rhodo- dendron pulchurum ‘Baifeng 4’)盆栽扦插苗为试验材料,在人工气候室内进行处理,以正常浇灌为对照(CK),土壤含水量为(70±5)%; 停止浇水2 d为中等干旱(middle drought,MD),土壤含水量为(60± 5)%; 停止浇水3 d为严重干旱(severe drought, SD), 土壤含水量为(45±5)%; 然后恢复正常浇水3 d设为复水处理(rewatering, RW),土壤含水量为(70±5)%, 每个处理30株。人工气候室的光/暗为12 h/12 h, 白天温度25℃,晚上温度15℃,空气相对湿度为65%。每个处理摘取从上往下第2~4片成熟功能叶进行RNA提取,用于转录组测序。
1.2 转录组测序杜鹃花叶片的RNA提取和转录组序列测定及分析参考WU等[1]的方法,获得Clean reads后通过De novo组装得到基因序列,然后,对差异表达的基因进行分析。本研究中,基因的表达量用RPKM值来统计,且界定Log2Ratio绝对值≥1、FDR≤0.01的基因为差异表达基因。在此基础上,通过BLAST与Nr库进行比对,筛选出差异表达的转录因子。
1.3 差异表达转录因子Real-time PCR验证转录组测序数据的有效性和准确性用实时荧光定量(RT-qPCR)法来验证。随机挑选6个差异表达的转录因子,用Primer Premier 5.0设计荧光定量PCR引物(表 1),以杜鹃叶片的cDNA为模板,以Actin基因为内参,并参考WU等[1]的方法分析基因的相对表达量。RPKM值用于统计基因的表达量, 是表示106个reads中来自于某基因1 kb长度的reads。每个样品4次重复。
| 表 1 6个转录因子的RT-qPCR引物 Table 1 qRT-PCR primers of 6 TFs |
以杜鹃花‘白凤4号’在4种处理下的植株叶片混合样本转录组数据构建参照文库,分析叶片的转录组表达谱信息,不同处理间共得到了962~12 558个差异表达基因(differential expression genes, DEGs) (表 2),其中对照与中等干旱处理间的DEGs最少,而严重干旱和复水处理间的DEGs最多。
| 表 2 差异表达基因的统计 Table 2 Statistic of differential expression genes |
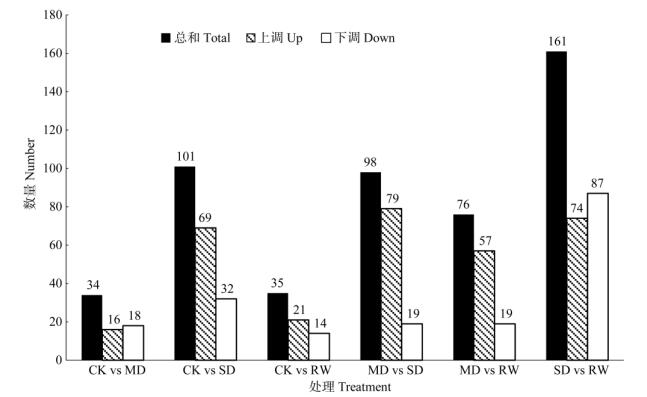
将这些差异表达基因通过BLAST与Nr库进行比对,总共有505个转录因子(图 1),其中含有转录因子最多的是严重干旱与复水处理间(161个,占本组DEGs总数的1.3%),其次是对照和严重干旱处理间(101个,占2.6%),最少的是对照与中等干旱处理间(34个,占3.7%)。进一步分析表明,与对照相比,随着干旱程度加深直至复水,转录因子的数量呈先上升后下降的趋势,且转录因子都以上调表达为主。与中等干旱相比,从严重干旱到复水,转录因子数量下降,但仍以上调表达为主。特别值得关注的是,复水与严重干旱相比,转录因子数量骤然增加,且以下调表达为主。
|
图 1 干旱处理的转录因子数量。CK:对照; MD:中等干旱; SD:严重干旱; RW:复水。下同 Fig. 1 Number of transcription factors under drought stress. CK: Control; MD: Middle drought; SD: Severe drought; RW: Rewatering. The same is following Figure |
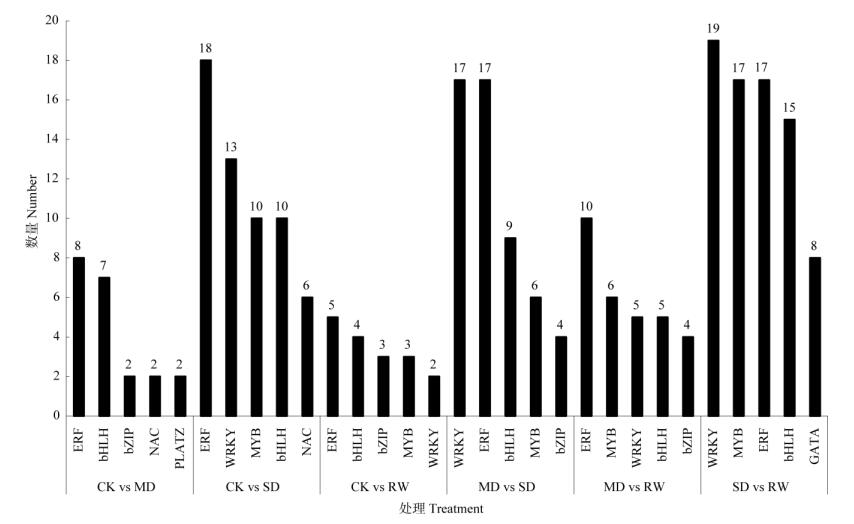
在获得转录因子数量基本信息后,进一步统计分析转录因子家族及其成员的数量关系。差异表达基因数量排前5位的家族共有254个转录因子,占转录因子总数(505个)的一半以上(图 2),其中ERF家族(75个)、WRKY家族(56个)、bHLH家族(50个)、MYB家族(42个)和bZIP家族(13个)占92.9%,且干旱处理的差异表达转录因子都比复水处理的多, 而NAC和PLATZ家族转录因子只在干旱处理中差异表达,GATA家族转录因子则只在复水处理中差异表达。
|
图 2 差异表达基因数量居前5位的转录因子家族统计 Fig. 2 Statistic of top five transcription factor families with DEGs |
由表 3可知,ERF、bHLH和MYB等3个家族在干旱和复水处理都有上调和下调表达的基因, WRKY和PLATZ家族基因在干旱胁迫中只上调表达,且PLATZ家族基因只在中等干旱时差异表达, bZIP家族基因在干旱和复水处理都只上调表达, GATA则通过基因的上调和下调响应旱后复水的刺激。
| 表 3 差异表达基因数量居前5位的家族基因转录因子数量 Table 3 Number of transcription factor in top 5 families with DEGs |
从表 4可见,差异表达基因在干旱处理和复水处理中的表达量(|log2(Fold change)|≥4)分别为-4.707~13.216、-4.545~10.749和-11.911~4.357, 且在干旱处理中以上调表达为主,在复水处理中以下调表达为主。其中,上调表达最显著的是NF-Y家族基因;其次是ERF家族基因。下调表达最显著的是ERF类转录因子,在复水处理中下调最为显著。此外,在干旱处理中bZIP家族基因的上调表达及热应激转录因子HsF家族基因的差异表达也比较显著。
| 表 4 高表达的转录因子 Table 4 Highly expressed genes in three comparison groups |
随机选取2个上调表达和4个下调表达的转录因子,用Real-time PCR方法验证测序数据的有效性(表 5)。结果表明,6个转录因子在不同处理中的表达趋势与转录组结果一致,从而验证了转录组测序数据的有效性。
| 表 5 差异表达转录因子的RT-qPCR验证 Table 5 RT-qPCR validation of differential expression transcription factors |
随着转录组测序技术的飞速发展和多个生物信息平台的加入,即使在没有基因组数据的前提下,转录组测序结果也可以为明确植物特定生理功能相关的代谢途径、挖掘未知基因及明晰基因调控机制提供大量信息。利用该技术,前人已挖掘了陆地棉(Gossypium hirsutum)[15]、枇杷(Eriobotrya japo- nica)[16]和梭梭(Haloxylon ammodendron)[17]等作物中的抗逆基因,并分析了相关基因的调控机制。
本研究中,杜鹃花协同调动了ERF、bHLH、MYB基因的差异表达来响应干旱胁迫和水分刺激,而在面临干旱胁迫时,增加NAC的差异表达和WRKY、bZIP、PLATZ上调表达来协同响应;在水分刺激下, 则偏向于增加GATA的差异表达来协同调节。同时, 在不同水分条件下,NF-Y家族基因的差异表达尤其明显,这说明这些转录因子在杜鹃花在应对干旱胁迫和水分刺激中具有重要调节作用。
PLATZ和GATA同属锌指蛋白,前者含有2个锌指结构域:C-X2-H-X11-C-X2-C-X(4-5)-C-X2-CX(3-7)- H-X2-H和C-X2-C-X(10-11)-C-X3-C[18],而后者在大多数植物中只含有1个锌指结构域C-X2-C-X18-C-X2- C[19]。这两类基因在植物抗逆中的作用研究相对较少,但其调控作用已得到验证。有研究表明,大豆(Glycine max)中GmPLATZ1可以被干旱、盐和ABA诱导表达[20],水稻(Oryza sativa)中的GATA响应冷处理[21]并能被ABA诱导表达[22],茉莉酸甲酯处理的穇(Eleusine coracana)中GATA基因可以通过与JAZ互作调控茉莉酸信号的转导[23]。
NF-Y类转录因子,含有3个亚基(NF-YA、NF- YB和NF-YC),这3个亚基形成异源三聚体后特异地结合真核生物启动子元件CCAAT盒,从而调控基因的转录,行使复杂的调控作用[24]。NF-Y三聚体及其亚基参与抗旱调节已在拟南芥(Arabidopsis)、大豆、大白菜(Brassica pekinensis)和小麦(Triticum aestivum)等植物中得到了验证。而且这种调控作用可能是通过参与ABA的合成和信号转导[25]、调控植物叶片的气孔开度[26]、促进脯氨酸的积累[27]、增加叶片叶绿素和可溶性蛋白含量[28]以及提高植物抗氧化能力[29]来实现的。同时,在植物抗旱中,NF-Y转录因子的转录还受到MicroRNAs的调节[25]和bZIP转录因子的协调作用[30]。
由此可见,ERF、bHLH、MYB、WRKY、NAC及bZIP等转录因子在杜鹃花干旱胁迫和水分刺激应答中可能起着主导作用,同时这些转录因子与PLATZ、GATA以及NF-Y等其他家族转录因子之间可能存在共表达性或协同作用,从而形成复杂且精细的调控网络来响应不同水分条件的刺激。
综上所述,本研究借助转录组测序技术,分析了干旱和复水处理中杜鹃‘白凤4号’叶的转录因子种类和表达规律,将为下一步筛选杜鹃花抗旱关键基因、研究杜鹃花干旱胁迫应答调控网络和分子育种等提供理论依据。
| [1] | WU J, WANG L F, LI L, et al. De novo assembly of the common bean transcriptome using short reads for the discovery of drought-responsive genes[J]. PLoS One, 2015, 10(3): e0119369 DOI:10.1371/journal.pone.0119369 |
| [2] |
LI C, SU J L, LIU X Q, et al. Effects of drought stress on seed germination and seedling physiological characteristics of Rhododendron latoucheae[J].
Acta Bot Boreali-Occid Sin, 2015, 35(7): 1421-1427. 李畅, 苏家乐, 刘晓青, 等. 干旱胁迫对鹿角杜鹃种子萌发和幼苗生理特性的影响[J]. 西北植物学报, 2015, 35(7): 1421-1427. DOI:10.7606/j.issn.1000-4025.2015.07.1421 |
| [3] |
GAO X N, ZHAO B, LIU X M, et al. Physiological response to drought stress and drought resistance evaluation of four Rhododendron cultivars[J].
J Zhejiang Agric For Univ, 2017, 34(4): 597-607. 高晓宁, 赵冰, 刘旭梅, 等. 4个杜鹃花品种对干旱胁迫的生理响应及抗旱性评价[J]. 浙江农林大学学报, 2017, 34(4): 597-607. DOI:10.11833/j.issn.2095-0756.2017.04.005 |
| [4] |
TANG F Y, JIN H X, TANG Y L. Effects of different drought stress on chlorophyll fluorescence of four Rhododendron cultivars[J].
J NW For Univ, 2017, 32(5): 64-68, 108. 汤飞洋, 金荷仙, 唐宇力. 不同程度干旱胁迫对4个杜鹃品种叶绿素荧光参数的影响[J]. 西北林学院学报, 2017, 32(5): 64-68, 108. DOI:10.3969/j.issn.1001-7461.2017.05.12 |
| [5] | FERNÀNDEZ-MARTÍNEZ J, FRANSI M A, FLECK I. Ecophysio-logical responses of Betula pendula, Pinus uncinata and Rhododendron ferrugineum in the catalan pyrenees to low summer rainfall[J]. Tree Physiol, 2016, 36(12): 1520-1535. DOI:10.1093/treephys/tpw104 |
| [6] | KIDOKORO S, WATANABE K, OHORI T, et al. Soybean DREB1/CBF-type transcription factors function in heat and drought as well as cold stress-responsive gene expression[J]. Plant J, 2015, 81(3): 505-518. DOI:10.1111/tpj.12746 |
| [7] | AMBAWAT S, SHARMA P, YADAV N R, et al. MYB transcription factor genes as regulators for plant responses:An overview[J]. Physiol Mol Biol Plants, 2013, 19(3): 307-321. DOI:10.1007/s12298-013-0179-1 |
| [8] |
FENG B, ZHU T F, ZHANG C, et al. Expression characteristics of poplar MYB gene in response to abiotic stresses[J].
J NE For Univ, 2017, 45(4): 19-22. 冯波, 朱腾飞, 张弛, 等. 杨树MYB基因应答非生物胁迫的表达特性[J]. 东北林业大学学报, 2017, 45(4): 19-22. DOI:10.13759/j.cnki.dlxb.2017.04.004 |
| [9] | MENG C M, ZHANG T Z, WANG Z G. Molecular cloning and characterization of a novel Gossypium hirsutum L. bHLH gene in response to ABA and drought stresses[J]. Plant Mol Biol Rep, 2009, 27(3): 381-387. DOI:10.1007/s11105-009-0112-5 |
| [10] |
ZHANG L, ZHANG T, TAN D F, et al. Isolation and drought induced expression characterization of ZmNAC99 gene from maize[J].
Acta Bot Boreali-Occid Sin, 2017, 37(4): 629-635. 张丽, 张庭, 谭登峰, 等. 玉米ZmNAC99基因的克隆及干旱诱导表达分析[J]. 西北植物学报, 2017, 37(4): 629-635. DOI:10.7606/j.issn.1000-4025.2017.04.0629 |
| [11] | SHI W N, HAO L L, LI J, et al. The Gossypium hirsutum WRKY gene GhWRKY39-1 promotes pathogen infection defense responses and mediates salt stress tolerance in transgenic Nicotiana benthamiana[J]. Plant Cell Rep, 2014, 33(3): 483-498. DOI:10.1007/S00299-013-1548-5 |
| [12] | HUANG W, SHANG Y F, CHEN P L, et al. Basic leucine zipper (bZIP) domain transcription factor MBZ1 regulates cell wall integrity, spore adherence, and virulence in Metarhizium robertsii[J]. J Biol Chem, 2015, 290(13): 8218-8231. DOI:10.1074/jbc.M114.630939 |
| [13] | DOSSA K, WEI X, LI D H, et al. Insight into the AP2/ERF trans-cription factor superfamily in sesame and expression profiling of DREB subfamily under drought stress[J]. BMC Plant Biol, 2016, 16: 171 DOI:10.1186/s12870-016-0859-4 |
| [14] | GOLLDACK D, LÜKING I, YANG O. Plant tolerance to drought and salinity:Stress regulating transcription factors and their functional significance in the cellular transcriptional network[J]. Plant Cell Rep, 2011, 30(8): 1383-1391. DOI:10.1007/s00299-011-1068-0 |
| [15] |
PENG Z, HE S P, GONG W F, et al. Transcriptome analysis of trans-cription factors in upland cotton seedlings under NaCl stress[J].
Acta Agron Sin, 2017, 43(3): 354-370. 彭振, 何守朴, 龚文芳, 等. 陆地棉幼苗NaCl胁迫下转录因子的转录组学分析[J]. 作物学报, 2017, 43(3): 354-370. DOI:10.3724/SP.J.1006.2017.00354 |
| [16] |
YANG W, GONG R G, SHI J J, et al. De novo assembly and functional annotation of the loquat young fruit transcriptome under chilling stress[J].
J NW Agric For Univ (Nat Sci), 2014, 42(8): 138-146. 杨伟, 龚荣高, 石佳佳, 等. 低温胁迫下枇杷幼果转录组的De novo组装和功能注释[J]. 西北农林科技大学学报(自然科学版), 2014, 42(8): 138-146. DOI:10.13207/j.cnki.jnwafu.2014.08.013 |
| [17] |
HAN C, XIE W H, LI J G, et al. Gene expression profile of haloxylon branch under the stresses of drought and heat in summer season[J].
Genom Appl Biol, 2015, 34(3): 607-614. 韩超, 谢文华, 李建贵, 等. 夏季高温干旱逆境下梭梭枝条中转录组表达谱分析[J]. 基因组学与应用生物学, 2015, 34(3): 607-614. DOI:10.13417/j.gab.034.000607 |
| [18] | NAGANO Y, FURUHASHI H, INABA T, et al. A novel class of plant-specific zinc-dependent DNA-binding protein that binds to A/T-rich DNA sequences[J]. Nucl Acid Res, 2001, 29(20): 4097-4105. DOI:10.1093/nar/29.20.4097 |
| [19] | REYES J C, MURO-PASTOR M I, FLORENCIO F J. The GATA family of transcription factors in Arabidopsis and rice[J]. Plant Physiol, 2004, 134(4): 1718-1732. DOI:10.1104/pp.103.037788 |
| [20] | SO H A, CHOI S J, CHUNG E, et al. Molecular characterization of stress-inducible PLATZ gene from soybean (Glycine max L.)[J]. Plant Omics J, 2015, 8(6): 479-484. |
| [21] | ZHANG T, HUANG L Y, WANG Y X, et al. Differential transcriptome profiling of chilling stress response between shoots and rhizomes of Oryza longistaminata using RNA sequencing[J]. PLoS One, 2017, 12(11): e0188625 DOI:10.1371/journal.pone.0188625 |
| [22] | GUPTA P, NUTAN K K, SINGLA-PAREEK S L, et al. Abiotic stresses cause differential regulation of alternative splice forms of GATA trans-cription factor in rice[J]. Front Plant Sci, 2017, 8: 1944 DOI:10.3389/fpls.2017.01944 |
| [23] | SEN S, KUNDU S, DUTTA S K. Proteomic analysis of JAZ inter-acting proteins under methyl jasmonate treatment in finger millet[J]. Plant Physiol Biochem, 2016, 108: 79-89. DOI:10.1016/j.plaphy.2016.05.033 |
| [24] | DOLFINI D, ZAMBELLI F, PAVESI G, et al. A perspective of promoter architecture from the CCAAT box[J]. Cell Cycle, 2009, 8(24): 4127-4137. DOI:10.4161/cc.8.24.10240 |
| [25] | NI Z Y, HU Z, JIANG Q Y, et al. GmNFYA3, a target gene of miR169, is a positive regulator of plant tolerance to drought stress[J]. Plant Mol Biol, 2013, 82(1/2): 113-129. DOI:10.1007/s11103-013-0040-5 |
| [26] | MU J Y, TAN H L, HONG S L, et al. Arabidopsis transcription factor genes NF-YA1, 5, 6 and 9 play redundant roles in male gametogenesis, embryogenesis, and seed development[J]. Mol Plant, 2013, 6(1): 188-201. DOI:10.1093/mp/sss061 |
| [27] |
HUANG R H, WANG G X, LIU F. Cloning and functional analysis of a drought-responsive gene BpNFYA5 from Chinese cabbage[J].
Acta Hort Sin, 2012, 39(8): 1501-1510. 黄人卉, 王桂香, 刘凡. 大白菜耐旱相关基因BpNFYA5的克隆及功能初步分析[J]. 园艺学报, 2012, 39(8): 1501-1510. DOI:10.16420/j.issn.0513-353x.2012.08.010 |
| [28] | YANG J, ZHU J H, YANG Y X. Genome-wide identification and expression analysis of NF-Y transcription factor families in watermelon (Citrullus lanatus)[J]. J Plant Growth Regul, 2017, 36(3): 590-607. DOI:10.1007/s00344-017-9670-1 |
| [29] | KUMIMOTO R W, SIRIWARDANA C L, GAYLER K K, et al. Nuclear factor Y transcription factors have both opposing and additive roles in ABA-mediated seed germination[J]. PLoS One, 2013, 8(3): e59481 DOI:10.1371/journal.pone.0059481 |
| [30] | LIU J X, HOWELL S H. bZIP28 and NF-Y transcription factors are activated by ER stress and assemble into a transcriptional complex to regulate stress response genes in Arabidopsis[J]. Plant Cell, 2010, 22(3): 782-796. DOI:10.1105/tpc.109.072173 |
 2018, Vol. 26
2018, Vol. 26



