巴西橡胶树(Hevea brasiliensis)原产于亚马逊流域[1],是大戟科(Euphorbiaceae)重要的热带经济作物之一,也是目前唯一大规模产业化的天然橡胶来源[2]。因天然橡胶具有合成橡胶无法替代的独特物理性能[3],使其在工业和国防等领域有广泛需求, 以天然橡胶为原料的产品超过4 000余种[4]。我国天然橡胶自给率较低,绝大多数天然橡胶原料依然需要进口。
由于巴西橡胶树属于典型的热带植物,国内宜植地区极其有限,只能通过选育优良高产品种来实现增产。但巴西橡胶树传统育种周期较长,基于基因测序的分子筛选和定向改良则为提高其育种效率提供了一条新的思路。近年来对巴西橡胶树的核基因组进行了测序和组装[5-8],重要细胞器叶绿体[9]和线粒体[10]的基因组也陆续发表,数万个与关键性状相关联的基因得以发掘和定位。随着生物技术手段的进步和测序成本的持续降低,针对大多数栽培品种和特异种质资源的重测序也即将开展,而基因组C值则是开展基因组测序的重要参考。
基因组C值(C-value)是指特定物种单倍体细胞核或配子中DNA的含量[11],也称之为基因组大小,通常以质量皮克(pg)或核苷酸碱基对数量(Gb)来表述,二者之间的换算关系为1 pg=0.978 Gb=0.978× 109 bp。对于同一植物而言,基因组C值不仅与物种的进化位置有关[12-13],而且在一定程度上与其种质资源的遗传多样性紧密相关,因此,基因组C值变异也是评价种质资源遗传基础宽窄的一种手段[14-15]。此外,对于同一种植物而言,细胞核中DNA含量与细胞核大小和染色体倍性呈线性相关,也可通过测定细胞核大小来进行倍性鉴定[16-17]。流式细胞术作为一种基于核酸荧光染色原理的基因组C值测定技术,因其操作简单,效率较高,目前已有许多作物种质资源的基因组C值被测定和分析[18-19],但对巴西橡胶树的流式细胞术基因组C值测定和变异分析的研究尚未见报道。
本研究以53份在云南植胶区综合性状表现较好的巴西橡胶树栽培种质为材料,采用流式细胞术对其细胞核DNA含量进行了测定,并对其基因组C值变异和染色体倍性进行了分析,提出了一种简洁有效的巴西橡胶树基因组C值测定方法,报道了一批巴西橡胶树栽培种质的基因组C值和染色体倍性,为巴西橡胶树基因组重测序和优良品种选育提供理论依据。
1 材料和方法 1.1 材料53份巴西橡胶树(Hevea brasiliensis)栽培种质是从云南省热带作物科学研究所农业部景洪巴西橡胶树种质资源圃中保存的600余份栽培种质中筛选出来的,这些种质的综合性状表现较好。内标材料玉米(Zea mays ‘CE-777’)和豌豆(Pisum sativum)的种子由捷克共和国实验植物学研究所(奥洛摩茨) Jaroslav Dolezel惠赠,繁殖出苗后采集嫩叶。
1.2 方法根据巴西橡胶树叶片组织特征和老嫩程度,在柳觐等[20-21]的基础上对流式细胞术测定方法进行了改进。首先选择CRTG-9、CRTG-191和CRTG-314等3份种质的古铜色嫩叶、浅绿色嫩叶和深绿色成熟叶(图 1)分别制样并测试(图 2)。然后选择供试种质的浅绿色嫩叶,以基因组大小已知的玉米或豌豆嫩叶作为内标进行混合制样并测试,每份种质重复测定3组,每组测定6~10个样品,细胞核荧光强度取平均值。运用CFlow Plus 1.0.264.15软件和Microsoft Office Excel 2016软件进行数据收集、作图和分析。内标玉米和豌豆的2n细胞核DNA含量分别为5.43和9.09 pg[22],待测样品基因组C值=内标样品基因组C值×(待测样品荧光强度/内标样品荧光强度)。

|
图 1 巴西橡胶树叶片。A:古铜色嫩叶; B:浅绿色嫩叶; C:深绿色成熟叶。图 2同 Fig. 1 Leaves of rubber tree. A: Bronze young leaves; B: Light green young leaf; C: Dark green mature leaf. The same is Figure 2 |
|
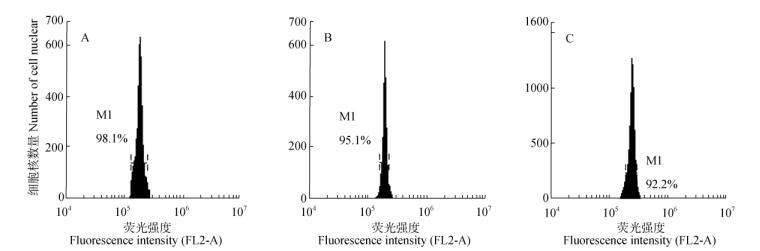
图 2 不同叶龄巴西橡胶树叶片的流式细胞术 Fig. 2 Flow cytometry diagram of rubber tree leaf at different age |
从图 2可以看出,古铜色嫩叶和深绿色成熟叶的流式细胞术样本峰(M1)较宽,表示样本中各个细胞核荧光强度值变异较大,说明测定精确度较差; 浅绿色嫩叶的流式细胞术样本峰窄,荧光强度值变异小,说明测定精确度更高。因此,在巴西橡胶树栽培种质的基因组大小测定中,均选择浅绿色嫩叶进行制样和测定。
2.2 基因组C值与变异分析对巴西橡胶树、玉米和豌豆的嫩叶进行了单独制样和流式细胞术检测,预估了基因组C值,表明基因组C值为巴西橡胶树 < 玉米 < 豌豆。因流式细胞术检测图中样本峰的横轴代表样本细胞核的荧光强度,而细胞核荧光强度直接反映样本的基因组C值大小,因此,在后续的检测中(图 3),无论是以玉米还是以豌豆作为内标,流式细胞术检测图中峰由左向右分别是巴西橡胶树体细胞细胞核的峰、内标材料体细胞细胞核的峰、内标材料体细胞有丝分裂前期染色质复制加倍后细胞核的峰(内标材料有丝分裂旺盛时有此峰,内标材料有丝分裂不旺盛时无此峰)。
|
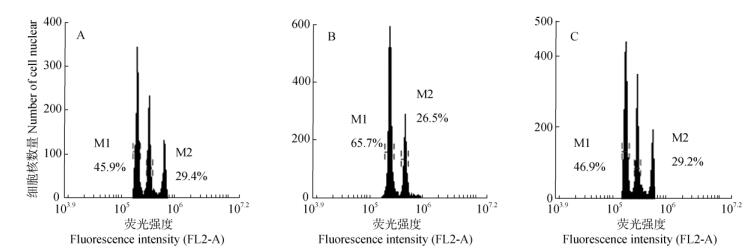
图 3 巴西橡胶树的流式细胞术测定图。A: CRTG-9种质, 玉米为内标; B: CRTG-186种质, 玉米为内标; C: CRTG-186种质, 豌豆为内标 Fig. 3 Flow cytometry diagram of rubber trees. A: CRTG-9 germplasm with Zea mays as interior label; B: CRTG-186 germplasm with Z. mays as interior label; C: CRTG-186 germplasm with Pisum sativum as interior label |
相比而言,玉米的基因组C值与巴西橡胶树更为接近。为减少误差,首先采用玉米作为内标,结果表明,其中47份种质的细胞核DNA含量可被准确测定(图 3: A),说明其基因组C值也可被准确测定和换算(表 1)。而CRTG-186、CRTG-187、CRTG- 188、CRTG-255、CRTG-272和CRTG-314等6份种质的流式细胞术检测图中仅有玉米的峰,缺少巴西橡胶树的峰(图 3: B),但将二者单独制样检测时却都可以得到很好的检测峰图,因此推测这6份种质的细胞核DNA含量与其他47份种质差异较大, 但与内标玉米的接近,故在流式细胞术检测图中二者的峰是重叠的。因此,再次采用豌豆作为内标进行测定(图 3: C),结果这6份种质的细胞核DNA含量也可被准确测定(表 1)。对53份种质的细胞核DNA含量比较发现,这6份种质的细胞核DNA含量为其他47份种质的1.5倍左右,因普通巴西橡胶树种质为二倍体[5-8],因此判定这6份种质为三倍体。
| 表 1 53份巴西橡胶树栽培种质的细胞核DNA含量和基因组C值 Table 1 Nuclear DNA content and genome C value of 53 cultivation rubber tree germplasms |
从表 1可见,53份巴西橡胶树栽培种质的细胞核DNA含量和基因组C值存在一定差异,细胞核DNA含量最小的是CRTG-172种质(3.007 966 pg), 最大的是CRTG-187种质(4.533 920 pg);基因组C值最小的是CRTG-272种质(1.465 908×109 bp), 最大的是CRTG-83种质(1.600 381×109 bp)。53份巴西橡胶树栽培种质的基因组C值平均值为1.531 696× 109 bp,与前人的研究结果[6-8]一致。变异分析表明,53份巴西橡胶树栽培种质的基因组C值变异系数较小(CV=0.035 5),说明其遗传基础相对狭窄。
2.3 巴西橡胶树和大戟科物种的基因组C值对比通常认为,物种的基因组C值是代表某个物种特征的常数,与其遗传和进化位置相关[12-13]。在植物基因组C值数据库[23]中检索到40种大戟科植物,隶属于14属(表 2)。基因组C值最小的是佛肚树(Jatropha podagrica),为0.293 4×109 bp,最大的是宝轮玉(Euphorbia polygona),为14.034 3×109 bp, 基因组C值变异系数(CV)高达1.248 6。不仅大戟科不同物种间的基因组C值变异较大,且隶属于同一属的不同物种的基因组C值变异也较大,大戟属19种植物中,基因组C值最小仅为0.337 41×109 bp,最大的为14.034 3×109 bp,变异系数(CV)也高达0.934 5。
| 表 2 大戟科植物的基因组C值 Table 2 Genome C value of Euphorbiaceae species |
样品选择是流式细胞术制样和检测的关键,过于幼嫩的叶片样品在采集后容易萎蔫,造成部分细胞核结构破坏;而过于成熟的叶片含有较多的次生代谢物,检测中易出现较多的杂质峰。本研究比较了不同幼嫩程度巴西橡胶树叶片的流式细胞术检测效果,首次建立了一套快速、准确测定巴西橡胶树细胞核DNA含量和基因组C值的方法,并对云南植胶区综合性状表现较好的主要栽培种质的基因组C值进行了测定和变异分析,为巴西橡胶树种质资源鉴定评价、基因组重测序和良种选育提供了重要理论依据。
在植物基因组C值数据库中,Bennett等[23]测定巴西橡胶树的基因组C值为2.102 7×109 bp,而本研究测定的巴西橡胶树基因组C值为1.531 696× 109 bp。通过比较[6-8],我们认为本研究的测定值更为准确,推测可能是Bennett等的测定时间为1997年,受研究技术、标准品选择等条件限制,导致估值出现了偏差。
本研究结果表明,53份云南植胶区综合性状表现较好的巴西橡胶树栽培种质中有11.32% (6份)为三倍体,远高于自然状态下植物多倍化的几率。这说明巴西橡胶树三倍体种质在云南高海拔植胶区本身就具有较好的综合性状表现优势,才使得其在多年的观测评价中被筛选出来。当前云南植胶区主推品种‘云研77-2’和‘云研77-4’等均为三倍体,在高海拔植胶区抗寒性强、产量表现好,提示我们可以在巴西橡胶树品种选育中重点关注三倍体种质, 以便提高云南植胶区高产抗寒品种的选育效率。
大戟科是被子植物中的大科,不仅是一个具有重要经济价值的植物学类群,也是热带植物区系中的重要成分[24]。本研究结果表明,大戟科不同物种的基因组C值大小与其种属分类没有必然联系,意味着其基因组C值与进化位置间不存在严格的对应关系,这与前人“C值悖论”的观点较为一致[11],原因尚需进一步研究。
| [1] | Zou Z, Liu J T, Yang L F, et al. Survey of the rubber tree genome reveals a high number of cysteine protease-encoding genes homo-logous to Arabidopsis SAG12[J]. PLoS One, 2017, 12(2): e0171725 DOI:10.1371/journal.pone.0171725 |
| [2] | ZOU Z, GONG J, AN F, et al. Genome-wide identification of rubber tree (Hevea brasiliensis Muell. Arg.) aquaporin genes and their response to ethephon stimulation in the laticifer, a rubber-producing tissue[J/OL]. BMC Genom, 2015, 16: 1001. doi: 10.1186/s12864-015-2152-6. |
| [3] | LERTPANYASAMPATHA M, GAO L, KONGSAWADWORAKUL P, et al. Genome-wide analysis of microRNAs in rubber tree (Hevea brasiliensis L.) using high-throughput sequencing[J]. Planta, 2012, 236(2): 437-445. DOI:10.1007/s00425-012-1622-1 |
| [4] | LIU J P, XIA Z Q, TIAN X Y, et al. Transcriptome sequencing and analysis of rubber tree (Hevea brasiliensis Muell.) to discover putative genes associated with tapping panel dryness (TPD)[J/OL]. BMC Genom, 2015, 16: 398. doi: 10.1186/s12864-015-1562-9. |
| [5] | RAHMAN A Y A, USHARRAJ A O, MISRA B B, et al. Draft genome sequence of the rubber tree Hevea brasiliensis[J/OL]. BMC Genom, 2013, 14: 75. doi: 10.1186/1471-2164-14-75. |
| [6] | TANG C R, YANG M, FANG Y J, et al. The rubber tree genome reveals new insights into rubber production and species adaptation[J/OL]. Nat Plant, 2016, 2: 16073. doi: 10.1038/nplants.2016.73. |
| [7] | LAU N S, MAKITA Y, KAWASHIMA M, et al. The rubber tree genome shows expansion of gene family associated with rubber biosynthesis[J/OL]. Sci Rep, 2016, 6: 28594. doi: 10.1038/srep28594. |
| [8] | POOTAKHAM W, SONTHIROD C, NAKTANG C, et al. De novo hybrid assembly of the rubber tree genome reveals evidence of paleote-traploidy in Hevea species[J/OL]. Sci Rep, 2017, 7: 41457. doi: 10.1038/srep41457. |
| [9] | TANGPHATSORNRUANG S, UTHAIPAISANWONG P, SANGSRA-KRU D, et al. Characterization of the complete chloroplast genome of Hevea brasiliensis reveals genome rearrangement, RNA editing sites and phylogenetic relationships[J]. Gene, 2011, 475(2): 104-112. DOI:10.1016/j.gene.2011.01.002.Epub2011Jan15 |
| [10] | SHEARMAN J R, SANGSRAKRU D, RUANG-AREERATE P, et al. Assembly and analysis of a male sterile rubber tree mitochondrial genome reveals DNA rearrangement events and a novel transcript[J/OL]. BMC Plant Biol, 2014, 14: 45. doi: 10.1186/1471-2229-14-45. |
| [11] | GREILHUBER J, DOLEŽEL J, LYSÁK MA, et al. The origin, evolu-tion and proposed stabilization of the terms 'genome size' and 'C-value' to describe nuclear DNA contents[J]. Ann Bot, 2005, 95(1): 255-260. DOI:10.1093/aob/mci019 |
| [12] | BIÉMONT C. Genome size evolution:Within-species variation in genome size[J]. Heredity, 2008, 101(4): 297-298. DOI:10.1038/hdy.2008.80 |
| [13] | PETROV D A. Evolution of genome size:New approaches to an old problem[J]. Trends Genet, 2001, 17(1): 23-28. DOI:10.1016/S0168-9525(00)02157-0 |
| [14] | KORBAN S S, WANNARAT W, RAYBURN C M, et al. Genome size and nucleotypic variation in Malus germplasm[J]. Genome, 2009, 52(2): 148-155. DOI:10.1139/G08-109 |
| [15] | YAN H H, MARTIN S L, BEKELE W A, et al. Genome size variation in the genus Avena[J]. Genome, 2016, 59(3): 209-220. DOI:10.1139/gen-2015-0132 |
| [16] | SAMOLUK S S, CHALUP L, ROBLEDO G, et al. Genome sizes in diploid and allopolyploid Arachis L. species (Section Arachis)[J]. Genet Resour Crop Evol, 2015, 62(5): 747-763. DOI:10.1007/s10722-014-0193-3 |
| [17] | CASTRO S, LOUREIRO J, RODRIGUEZ E, et al. Evaluation of poly-somaty and estimation of genome size in Polygala vayredae and P. calcarea using flow cytometry[J]. Plant Sci, 2007, 172(6): 1131-1137. DOI:10.1016/j.plantsci.2007.03.002 |
| [18] |
LIU J, KONG G H, NI S B, et al. Estimation of genomic C value of Macadamia integrifolia spp. by flow cytometry[J].
Chin Agric Sci Bull, 2013, 29(34): 96-101. 柳觐, 孔广红, 倪书邦, 等. 基于流式细胞术的澳洲坚果基因组C值测定[J]. 中国农学通报, 2013, 29(34): 96-101. DOI:10.11924/j.issn.1000-6850.2013-1305 |
| [19] |
LIU J, LI K X, KONG G H, et al. Genome size and variation analysis of mango (Mangifera indica L.) germplasms in Yunnan by flow cyto-metry[J].
J Trop Subtrop Bot, 2015, 23(4): 386-390. 柳觐, 李开雄, 孔广红, 等. 云南芒果种质基因组大小测定与变异分析[J]. 热带亚热带植物学报, 2015, 23(4): 386-390. DOI:10.11926/j.issn.1005-3395.2015.04.004 |
| [20] |
LIU J, KONG G H, LI K X, et al. Estimation of genomic C value of Mangifera indica L. by flow cytometry[J].
Chin J Trop Crop, 2015, 36(9): 1626-1630. 柳觐, 孔广红, 李开雄, 等. 基于流式细胞术的芒果基因组C值测定[J]. 热带作物学报, 2015, 36(9): 1626-1630. DOI:10.3969/j.issn.1000-2561.2015.09.013 |
| [21] | OTTO F. DAPI staining of fixed cells for high-resolution flow cyto-metry of nuclear DNA[J]. Methods Cell Biol, 1990, 33: 105-110. DOI:10.1016/S0091-679X(08)60516-6 |
| [22] | DOLEZEL J, GREILHUBER J, SUDA J. Estimation of nuclear DNA content in plants using flow cytometry[J]. Nat Protoc, 2007, 2(9): 2233-2244. DOI:10.1038/nprot.2007.310 |
| [23] | Royal Botanic Gardens, Kew. Plant DNA C-values database[EB/OL]. (2012-12-01)[2017-07-20]. http://data.kew.org/cvalues. |
| [24] |
XU Z L, YU B Y, XU L S. Numerical analysis of the euphorbiaceae[J].
J Trop Subtrop Bot, 2004, 12(5): 399-404. 徐增莱, 余伯阳, 徐珞珊. 大戟科植物分类的数值分析[J]. 热带亚热带植物学报, 2004, 12(5): 399-404. DOI:10.3969/j.issn.1005-3395.2004.05.002 |
 2018, Vol. 26
2018, Vol. 26


