2. 广东药科大学, 广州 510006
2. Guangdong Pharmaceutical University, Guangzhou 510006, China
加勒比松(Pinus caribaea Morelet)分布于中美洲,有3个变种,即古巴加勒比松(P. caribaea var.caribaea)、洪都拉斯加勒比松[P. caribaea var. hondurensis (Sénéclauze) W. H. Barrett & Golfari]和巴哈马加勒比松[P. caribaea var. bahmaensis (Grise-bach) W. H. Barrett & Golfari],是世界上重要的用材、纸浆材和采脂树种,具有生长快,适应性强等特点。3个加勒比松变种于20世纪60年代后期先后引入我国,在我国两广南部及海南岛种植。在我国加勒比松作为南方重要工业原料树种湿加松(P.elliottii×P.caribaea)的父本得到进一步改良, 如何有效利用加勒比松种质资源已成为树种改良及培育良种的重要因素之一。
种质资源收集与保存是实现树种改良的前提, 种质资源间亲缘关系、遗传多样性是加快改良进程、制定遗传改良策略的重要参考。表型、分子标记、测序等技术是种质资源亲缘关系和多样性评价的常用手段。通过表型性状研究加勒比松3个变种的系统发育,发现变种间的亲缘关系与其地理分布是相对一致[1]。同工酶研究表明,古巴加勒比松和巴哈马加勒比松自然群体具有高的基因多样性水平(He=0.26)[2]。SSR标记研究表明,巴哈马加勒比松先与北部洪都拉斯加勒比松聚为一组,东南部洪都拉斯加勒比松聚为一组,再与古巴加勒比松聚为一组[3]。RAPD标记聚类分析表明,巴哈马加勒比松与湿地松存在较近的亲缘关系,然后是古巴加勒比松、洪都拉斯加勒比松[4]。质体DNA测序表明,洪都拉斯加勒比松与巴哈马加勒比松遗传关系较近,与古巴加勒比松的亲缘关系相对较远[5]。目前加勒比松变种亲缘关系和多样性研究取得一些进展,但从表观遗传方面开展加勒比松DNA甲基化多样性的研究尚未涉及。
DNA甲基化属于表观遗传学范畴,是真核细胞基因组在复制和转录后最为常见的一种修饰方式, 对于生物生命活动起重要作用[6]。目前普遍采用甲基化敏感扩增多态性(methylation sensitive amplifi- cation polymorphism, MSAP)技术检测DNA甲基化, 进而开展多样性研究,该方法利用同裂酶HpaⅡ/MspⅠ对识别序列CCGG的甲基化敏感性的不同, 能够识别的序列CCGG产生的甲基化状态就会导致两者发生酶切和扩增,从而产生多态性,已在玉米(Zea may)[7]、菊花脑(Chrysanthemum nankin- gense)[8]、陆地棉(Gossypium hirsutum)[9]、樱桃(Prunus avium)[10]、苏铁(Cycas)[11]等植物遗传多样性、亲缘关系的研究上广泛应用。
本研究以17个加勒比松种源为研究对象,采用MSAP技术对基因组DNA甲基化多样性进行分析,了解加勒比松种源DNA甲基化多样性,为进一步利用加勒比松种源奠定基础。
1 材料和方法 1.1 试验材料供试17个加勒比松种源材料见表 1,1~10号洪都拉斯加勒比松种源引自洪都拉斯,12号洪都拉斯加勒比松种源引自澳大利亚昆士兰种子园,18号洪都拉斯加勒比松种源引自巴西Lençóis Paulista种子园;11号、17号、41号古巴加勒比松种源分别引自古巴Pinar del Río天然林、巴西Lençóis Paulista种子园、墨西哥,13号古巴加勒比松种源来自湛江遂溪种子园;16号巴哈马加勒比松种源引自巴西Lençóis Paulista种子园;上述各类种子园中,不同变种间没有混合种植,其种子园中子代种子不存在变种间杂交。2015年6月种子播种于广东省林业科学研究院中心苗圃,2016年4月当苗龄10个月时取成熟针叶用于试验。
| 表 1 加勒比松种源 Table 1 Provenances of Piuns caribaea |
基因组DNA提取 采用DNeasy Plant Mini Kit试剂盒(QIAGEN),DNA的完整性用1%的琼脂糖凝胶电泳进行检测,紫外吸收法检测基因组DNA的浓度及纯度。每个种源采集15株针叶提取DNA,等量DNA混合进行分析,-20℃保存备用。
DNA甲基化的MSAP分析 采用优化的MSAP反应体系。分别用EcoRI+HpaⅡ (Promega, 美国)和EcoRI+MspⅠ (Promega,美国)对杂交子代及亲本基因组DNA进行酶切。随后用T4 DNA ligase (TaKaRa,日本)进行连接,接头引物、预扩增引物、选择性扩增引物序列见表 2。1 μL连接产物用于预扩增引物,程序为:94℃30 s,65℃1 min,72℃1 min,25个循环。2 μL稀释10倍的预扩增产物用于降落PCR扩增,反应程序参考Neha等[7]的方法。选择性扩增产物经变性、分离后,8%聚丙烯酰胺凝胶电泳2.5 h (30 W),硝酸银染色干燥后读带。
| 表 2 MSAP引物序列 Table 2 Sequences of primers used for MSAP analysis |
将所有扩增得到的MSAP条带转为二维矩阵, “1”、“0”分别代表条带的有、无。多态位点百分率(PPL)、Nei’s基因多态性指数(H)、Shannon信息指数(Ⅰ)等遗传多样性参数采用Popgene V. 1.32软件计算。利用2.10e版NTSYSpc软件(Applied Biostatistics, 美国)进行聚类,并对MSAP分析进行Mantel检测。
2 结果和分析 2.1 加勒比松DNA甲基化差异利用56对引物组合共扩增出425条谱带,平均每对引物组合扩增7.6条谱带,E2-H6引物扩增条带最少,只有3条,E5-H6、E5-H8、E7-H1引物扩增的最多,有13条。多态性谱带422条,多态性谱带百分率99.25%,说明各种源间DNA甲基化多态性位点非常丰富。
石奔[12]将甲基化模式分为4类,Ⅰ型为5′-CC- GG-3′胞嘧啶未甲基化和内侧胞嘧啶未甲基化(1, 1);Ⅱ型为5′-CCGG-3′外侧胞嘧啶半甲基化(1, 0); Ⅲ型为5′-CCGG-3′内侧胞嘧啶全甲基化(0,1);Ⅳ型为5′-CCGG-3′内外侧胞嘧啶全甲基化(0, 0)(可能)。由于Ⅳ型为不确定类型,不作计算。从表 3可见,12个洪都拉斯加勒比松种源中Ⅱ型甲基化率(11.74%)高于Ⅲ型(10.65%),除1、2、8号种源的Ⅱ型甲基化率低于Ⅲ型的外,其余种源均高于Ⅲ型;3号种源的Ⅱ型甲基化率(13.88%)较高,而2号种源的Ⅲ型甲基化率(13.18%)最高。4个古巴加勒比松种源中11、13号种源的Ⅱ型甲基化率低于Ⅲ型,17、41号种源的Ⅱ型甲基化率高于Ⅲ型;13号种源的Ⅱ型(13.41%)、Ⅲ型甲基化率(18.59%)均最高。巴哈马加勒比松16号种源的Ⅱ型甲基化率为11.76%,Ⅲ型甲基化率为10.59%,Ⅱ型甲基化率高于Ⅲ型。DNA全甲基化率(Ⅲ型)最小的是洪都拉斯加勒比松10号种源(7.28%),最大的是古巴加勒比松13号种源(18.59%);DNA半甲基化率(Ⅱ型)为6.59%~15.29%。17个种源间不同模式的甲基化率存在一定差异。
| 表 3 加勒比松种源CCGG位点的胞嘧啶甲基化水平 Table 3 Cytosine methylation levels of CCGG locus among provenances of Piuns caribaea |
从变种来看,洪都拉斯加勒比松种源、巴哈马加勒比松种源的Ⅱ型甲基化率(分别为11.74%和11.76%)高于Ⅲ型的(分别为10.65%和10.59%), 古巴加勒比松种源的相反,Ⅲ型的甲基化率(11.82%)高于Ⅱ型的(10.47%)。洪都拉斯加勒比松种源与巴哈马加勒比松种源的Ⅱ型和Ⅲ型甲基化率相近, 他们的Ⅱ型甲基化率均高于古巴加勒比松种源的,而Ⅲ型甲基化率则低于古巴加勒比松种源。Ⅲ型甲基化率以古巴加勒比松种源 > 洪都拉斯加勒比松种源 > 巴哈马加勒比松种源,Ⅱ型甲基化率以巴哈马加勒比松种源 > 洪都拉斯加勒比松种源 > 古巴加勒比松种源。加勒比松的DNA总甲基化水平相对较低,其中洪都拉斯加勒比松种源为18.78%~27.53%, 古巴加勒比松种源为17.41%~32%,巴哈马加勒比松种源种源为22.35%。
2.2 MSAP遗传多样性分析为探讨不同加勒比松种源基因组中发生甲基化的多样程度,依据Cervera等[13]的方法产生了17个种源的2个二维矩阵,即由DNA序列变异引起的甲基化不敏感扩增多态性(methylation-insensitive polymorphism, MISP)和由甲基化变异引起的甲基化敏感扩增多态性(methylation-sensitive polymer- phism, MSP)。以MISP为基础分析17个种源的遗传多样性水平,加勒比松种源多态性位点数为425,多态性位点百分率为100.00%。Nei’s基因多态性指数(H)和Shannon信息信息指数(Ⅰ)分别为0.4376、0.6272。以MSP为基础分析17个种源的DNA甲基化多样性,加勒比松种源的多态性位点数为421,多态性位点百分率为99.06%。Nei’s基因多态性指数(H)和Shannon信息信息指数(Ⅰ)分别为0.3274和0.5026。这表明17个加勒比松种源在物种水平上具有较高的遗传与表观遗传多样性,DNA序列遗传多样性高于表观遗传多样性。将MISP矩阵与MSP矩阵进行Mantel检验,基因组遗传变异与表观遗传变异不存在相关性(r=-0.171,P=0.16)。
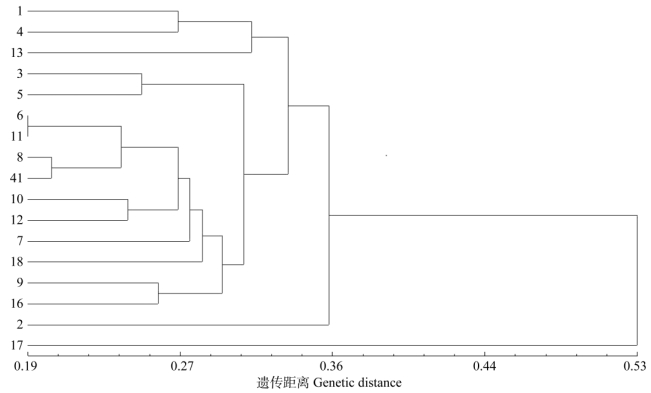
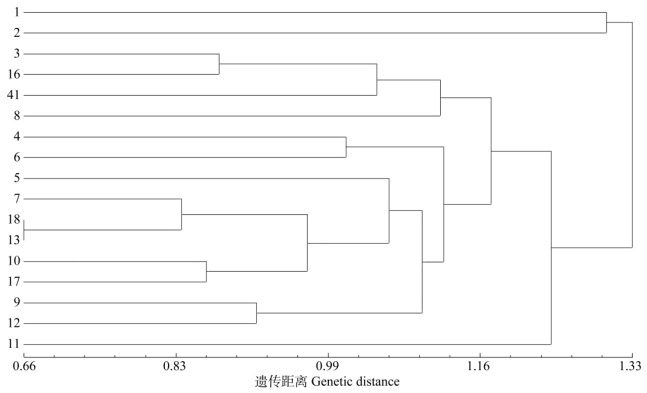
2.3 聚类分析利用NTSYSpc软件进行Mantel检测,将已经获得的MISP和MSP两种转化数据,通过UPGMA法,得到MISP和MSP的聚类分析结果(图 1, 2), Mantel检测的r值分别为0.9004和0.6342,说明聚类结果相对较好。
|
图 1 加勒比松17个种源的MISP聚类。1~17见表 1。下图同。 Fig. 1 UPGMA dendrogram of 17 provenances of Piuns caribaea based MISP. 1-17 see Table 1. The same is following Figure. |
|
图 2 加勒比松17个种源的MSP聚类 Fig. 2 UPGMA dendrogram of 17 provenances of Piuns caribaea based MSP |
以MISP为基础,17个种源的遗传距离为0.186~0.608,6号种源与11号种源的遗传距离最小,为0.186,4号种源与17号种源的遗传距离最大,为0.608。从图 1可见,17个种源在遗传距离0.53处分为4大类,第一类为1、4和13号种源, 第二类为3、5、6、11、8、41、10、12、7、18、9和16号种源,第三类为2号种源,第四类为17号种源。
以MSP为基础,17个种源间的表观遗传距离为0.663~1.848,13号与18号种源的遗传距离最近(0.663),2号与11号种源的遗传距离最远(1.848), 平均表观遗传距离为1.133。从图 2可见,17个种源在遗传距离1.33处分为3大类,第一类为1和2号种源,第二类为3、16、41、8、4、6、5、7、18、13、10、17、9和12号种源,第三类为11号种源。
表观聚类与遗传聚类间存在较大差异,两种聚类方法均未将3个加勒比松变种分开,但MSP聚类结果可以将古巴加勒比松11号种源与其他种源分开,表明加勒比松变种间的表观遗传变异较为丰富,不同变种间可能存在一定的基因渗透。
3 讨论 3.1 加勒比松种源苗期的DNA甲基化模式本研究结果表明,加勒比松种源苗期的DNA甲基化水平相对较低,17个种源的DNA甲基化率为17.41%~32.00%,其中洪都拉斯加勒比松种源为18.78%~27.53%,古巴加勒比松种源为17.41%~ 32.00%,巴哈马加勒比松种源为22.35%,低于意大利石松(Pinus pinea, 42.73%~47.90%)[14]、菊花脑(54.6%~62.6%)[9]和白菜(Brassica rapa ssp. pekinensis, 23.9%~55.8%)[15],而与毛白杨(Populus tomentosa, 26.75%~29.39%)[16]、欧美杨(P. euramericana, 16.93%~25.11%)[17]、杉木(Cunninghamia lanceolata, 20.66%~25.40%)[18]、落叶松(Larix gmelinii, 19.92%~ 23.14%)[19]和棉花(Gossypium hirsutum, 19.18%~ 28.69%)[20]等物种相近。有研究表明,半甲基化是在DNA复制过程中产生的,是DNA复制过程中的一个中间状态[13]。如芦苇(Phragmites austrilis)发育完全的幼嫩叶片全甲基化率高于半甲基化率[12],对模式植物拟南芥(Arabidopsis thaliana)的甲基化研究得到同样结论[21];芥蓝(Brassica oleracea var.alboglabra)的DNA甲基化模式以半甲基化为主[22],一年生辣椒(Capsicum annuum)与中华辣椒(C. chinense)种质的内甲基化率大于外甲基化率[23]。本研究中1、2、8、11和13号种源的全甲基化率高于半甲基化率外,其他种源的全甲基化率低于半甲基化率,加勒比松种源苗期的甲基化模式以半甲基化为主,这是否与参试种源苗期幼态性、发育时期、种源数量、不同种源分布区环境以及不同种源类型等因素有关,还有待进一步研究。
3.2 加勒比松种源苗期的表观遗传多样性水稻(Oryza sativa)的DNA甲基化率(52.37%)明显高于DNA序列多样性位点的比例(17.00%)[24], 西瓜(Citrullus lanatus)的基因组甲基化率(43%)也高于遗传多样性(19.8%)[25],说明基因组DNA甲基化的可塑性强于DNA序列的可塑性。本研究中加勒比松种源的遗传多样性高于表观遗传多样性,表明种源间的遗传变异更加丰富。另外MISP遗传多样性比SSR/SRAP加勒比松群体大树的平均遗传多样性(H=0.1743、I=0.2665)高[26],这可能与试验材料及材料的不同发育时期有关。
栽培人参(Panax ginseng)与野生人参间的基因遗传距离(0.0343)显著大于二者的表观遗传距离(0.0164),说明驯化造成栽培人参和野生人参间的遗传分化要显著大于二者间的表观遗传分化[27]。本研究中,加勒比松的表观遗传距离明显大于基因遗传距离,这与对人参等植物的研究结果相反,初步表明在加勒比松进化过程中表观遗传分化作用明显大于基因遗传分化。中国杂交籼稻ISSR标记与MSAP标记的遗传距离呈正相关(r=0.55, P=0.04), 二者的相关系数达到0.89,表明在遗传多样性评价上两种技术大致等效[24]。本研究中3个加勒比松变种间的MISP遗传距离变异范围与SSR/SRAP[26]遗传距离变异范围相比,总体差异不大,说明可以用MSAP技术开展加勒比松遗传多样性研究。
有研究表明,表观等位基因可以引起明显的表型差异,由此可以推断,甲基化多样性是群体分化的一个重要影响因素[28]。对一年生野生大豆(Glycine soja)和栽培大豆(G. max)甲基化遗传多样性进行聚类分析,结果表明,有2~4个栽培大豆被分到野生大豆群体里,但没有野生大豆分在栽培大豆群体里,这有可能是野生大豆和栽培大豆间的渐渗产生的[29]。表观遗传聚类结果同样表明,栽培人参中有2个个体被分到野生人参的分支中[27]。丹参(Salvia miltiorrhiza)的MSP聚类表明,不同居群间的单株有交叉现象[30]。同一辣椒栽培种未归为同一表观遗传聚类中,表明辣椒表观遗传变异较基因组遗传变异更为丰富[23]。另有研究表明,与DNA序列变异相比,DNA甲基化变异更加普遍,如不同生态环境下(河畔和盐碱地)拉关木(Laguncularia racemosa)的DNA甲基化存在大量变异,而基因组变异较小[31];不同生态型拟南芥甲基化调控机制不同,导致甲基化多态性不相关,最终亲缘关系较近个体未归为同一类[13]。本研究的MISP聚类结果表明,古巴加勒比松11号种源与洪都拉斯加勒比松6号种源关系最近,与Dvorak等[4]用RAPD和Lev[3]用SSR标记获得的聚类结果不同。其他古巴加勒比松、巴哈马加勒比松也不同程度地与洪都拉斯加勒比松聚在一起,没有被分开,表明加勒比松表观遗传变异极为丰富。但值得注意的是利用MSP聚类,能将原生种源11号与其他大部分种源分开。本研究中参试材料有11个原生种源来自天然林,6个次生种源来自不同改良程度的种子园,可能这些原生种源和次生种源在适应不同环境过程中甲基化发生不同程度变化,环境不同导致不同种源间具有不同的甲基化调控机制。
3.3 加勒比松种源苗期的DNA甲基化与遗传变异对西瓜[25]、水稻[32]、芦苇[12]等的研究表明, 基因组遗传多样性与表观遗传多样性间存在相关性或聚类结果相似,说明DNA甲基化遗传变异与基因遗传变异相伴发生,前者依赖于后者调控,这也表明DNA甲基化在调节植物基本适应能力方面是一个可塑的机制[12]。对高度自花授粉植物拟南芥10个生态型(30~50株混合为1个DNA样品)的DNA甲基化研究表明,甲基化敏感多态性与甲基化不敏感多态性二维矩阵间没有相关性,说明甲基化可能具有独立的控制系统[13]。而对高度异交植物短芒野大麦(Hordeum brevisubulatum)的研究表明,基于MSP与MISP、AFLP和S-SAP的Jaccard相似性二维矩阵具有一定的相关,不能判别甲基化状态控制系统是否为独立的系统[33]。水稻的基因遗传与表观遗传变异间的相关系数为62%, 表明一些DNA甲基化与DNA序列间存在相关性,也存在一定独立性,独立性位点的选择对水稻品种改良具有独特贡献[24]。本研究中加勒比松甲基化多样性与遗传多样性没有相关性,这与拟南芥[13, 34-36]、水稻[36-37]、大豆[29]、甜樱桃[10]的研究结果一致,表明DNA甲基化与基因遗传变异的产生存在不同的机制。另外加勒比松种源表观和遗传的聚类结果差异较大,同样说明表观遗传学与基因组遗传变异可能存在不同的调控机制,这种调控可能是独立的。
| [1] | ADAMS D C, JACKSON J F. A phylogenetic analysis of the southern pines (Pinus subsect. Australes Loudon):Biogeographical and ecolo-gical implications[J]. Proc Biol Soc Washington, 1997, 110(4): 681-692. |
| [2] | ZHENG Y Q, ENNOS R A. Genetic variability and structure of natural and domesticated populations of Caribbean pine (Pinus caribaea Morelet)[J]. Theor Appl Genet, 1999, 98(5): 765-771. DOI:10.1007/s001220051133 |
| [3] | JARD N-BARBOLLA L, DELGADO-VALERIO P, GEADA-L PEZ G, et al. Phylogeography of Pinus subsection Australes in the Cari-bbean Basin[J]. Ann Bot, 2011, 107(2): 229-241. DOI:10.1093/aob/mcq232 |
| [4] | DVORAK W S, JORDON A P, HODGE G P, et al. Assessing evolu-tionary relationships of pines in the Oocarpae and Australes subsec-tions using RAPD markers[J]. New For, 2000, 20(2): 163-192. DOI:10.1023/A:1006763120982 |
| [5] | LÓPEZ G G, KAMIYA K, HARADA K. Phylogenetic relationships of Diploxylon pines (subgenus Pinus) based on plastid sequence data[J]. Int J Plant Sci, 2002, 163(5): 737-747. DOI:10.1086/342213 |
| [6] | IBARRA C A, FENG X Q, SCHOFT V K, et al. Active DNA demethylation in plant companion cells reinforces transposon methy-lation in gametes[J]. Science, 2012, 337(6100): 1360-1364. DOI:10.1126/science.1224839 |
| [7] | ROY N, CHOI J Y, LIM M J, et al. Genetic and epigenetic diversity among dent, waxy, and sweet corns[J]. Genes Genom, 2015, 37(10): 865-874. DOI:10.1007/s13258-015-0329-7 |
| [8] | QI X Y, WANG H B, NING Y X, et al. Genetic diversity and methy-lation polymorphism analysis of Chrysanthemum nankingense[J]. Biochem Syst Ecol, 2017, 72: 1-7. DOI:10.1016/j.bse.2017.03.006 |
| [9] | KEYTE A L, PERCIFIELD R, LIU B, et al. Infraspecific DNA methy-lation polymorphism in cotton (Gossypium hirsutum L.)[J]. J Hered, 2006, 97(5): 444-450. DOI:10.1093/jhered/esl023 |
| [10] | AVRAMIDOU E V, GANOPOULOS I V, DOULIS A G, et al. Beyond population genetics:Natural epigenetic variation in wild cherry (Prunus avium)[J]. Tree Genet Genom, 2015, 11(5): 95 DOI:10.1007/s11295-015-0921-7 |
| [11] | SAE-EUNG C, KANCHANAKETU T, SANGDUEN N, et al. DNA methylation and genetic diversity analysis of genus Cycas in Thailand[J]. Afr J Biotechnol, 2012, 11(4): 743-751. DOI:10.5897/AJB11.2835 |
| [12] |
SHI B. Epigenetic diversity of 4 wild reed[Phragmites austrilis (Cav. ) Trirn. ex Steud] natural populations endemic to Nenjiang Plain, China[D]. Changchun: Northeast Normal University, 2012: 15-28.
石奔. 中国嫩江平原芦苇[Phragmites austrilis (Cav. ) Trirn. ex Steud]四个天然种群的表观遗传多样性研究[D]. 长春: 东北师范大学, 2012: 15-28. |
| [13] | CERVERA M T, RUIZ-GARCĺA L, MARTĺNEZ-ZAPATER J. Analysis of DNA methylation in Arabidopsis thaliana based on methy-lation-sensitive AFLP markers[J]. Mol Genet Genom, 2002, 268(4): 543-552. DOI:10.1007/s00438-002-0772-4 |
| [14] | SĺEZ-LAGUNA E, GUEVARA M , DĺAZ L M, et al. Epigenetic variability in the genetically uniform forest tree species Pinus pinea L.[J]. PLoS One, 2014, 9(8): e103145 DOI:10.1371/journal.pone.0103145 |
| [15] | TAO L, WANG X L, GUO M H, et al. Analysis of genomic DNA methylation and gene expression in Chinese cabbage (Brassica rapa L. ssp. pekinensis) after continuous seedling breeding[J]. Russ J Genet, 2015, 51(8): 774-782. DOI:10.1134/S1022795415080116 |
| [16] |
MA K F, ZHANG Z Y, WANG S Q, et al. Optimization of MSAP system and preliminary analysis of DNA methylation in Populus tomentosa[J].
J NE For Univ, 2012, 40(12): 1-7. 马开峰, 张志毅, 王斯琪, 等. 毛白杨MSAP体系优化及DNA甲基化的初步分析[J]. 东北林业大学学报, 2012, 40(12): 1-7. DOI:10.3969/j.issn.1000-5382.2012.12.001 |
| [17] | HU B Q, HAN C L, WANG B, et al. Cytosine methylation at CG and CNG sites is differential during the development of triploid black poplar[J]. J Plant Biochem Biotechnol, 2013, 22(4): 414-424. DOI:10.1007/s13562-012-0170-x |
| [18] |
HONG Z, SHI J S, ZHENG R H, et al. Epigenetic inheritance and variation of DNA methylation in Chinese Fir (Cunninghamia lanceo-lata) intraspecific hybrids[J].
Mol Plant Breed, 2009, 7(3): 591-598. 洪舟, 施季森, 郑仁华, 等. 杉木亲本自交系及其杂交种DNA甲基化和表观遗传变异[J]. 分子植物育种, 2009, 7(3): 591-598. DOI:10.3969/mpb.007.000591 |
| [19] |
LI A, LIU C, HAN C L, et al. Variation in cytosine methylation patterns between reciprocal hybrids and their parental line in Larix[J].
Acta Sci Nat Univ Nankai, 2012, 45(5): 65-71. 李爱, 刘超, 韩春乐, 等. 落叶松优势杂交子代与亲本间基因组DNA甲基化变异研究[J]. 南开大学学报(自然科学版), 2012, 45(5): 65-71. |
| [20] | WEI J, FAN H H, LI T C, et al. The effects of reciprocal cross on inheritance of DNA methylation in cotton (Gossypium hirsutum)[J]. Afr J Biotechnol, 2012, 11(23): 6383-6386. DOI:10.5897/AJB11.4212 |
| [21] | KAEPPLER S M, PHILLIPS R L. Tissue culture-induced DNA methylation variation in maize[J]. Proc Natl Acad Sci USA, 1993, 90(19): 8773-8776. DOI:10.1073/pnas.90.19.8773 |
| [22] |
SHI W D, HUANG R K, CHEN Z D, et al. The epigenetic genetic diversity of 18 Chinese kales analyzed by MSAP when initiated flowering[J].
Genom Appl Biol, 2012, 31(5): 505-512. 史卫东, 黄如葵, 陈振东, 等. 利用MSAP分析18个芥蓝齐口期的表观遗传多样性[J]. 基因组学与应用生物学, 2012, 31(5): 505-512. DOI:10.3969/gab.031.000505 |
| [23] |
LI T, XU X W, LI Y, et al. Surveying DNA methylation diversity in the Capsicum annuum L. and Capsicum chinense Jacquin[J].
Mol Plant Breed, 2014, 12(2): 306-315. 李涛, 徐小万, 李颖, 等. 一年生辣椒(Capsicum annuum L.)与中华辣椒(Capsicum chinense Jacquin) DNA甲基化多样性分析[J]. 分子植物育种, 2014, 12(2): 306-315. DOI:10.13271/j.mpb.012.000306 |
| [24] |
PENG H, JIANG G H, ZHANG J, et al. DNA methylation poly-morphism and stability in Chinese indica hybrid rice[J].
Sci China Life Sci, 2014, 44(1): 45-53. 彭海, 江光怀, 张静, 等. 中国杂交籼稻DNA甲基化多样性与遗传稳定性[J]. 中国科学:生命科学, 2014, 44(1): 45-53. DOI:10.1007/s11427-013-4576-z |
| [25] | NIMMAKAYALA P, VAJJA G, GIST R A, et al. Effect of DNA methylation on molecular diversity of watermelon heirlooms and stabi-lity of methylation specific polymorphisms across the genealogies[J]. Euphytica, 2011, 177(1): 79-89. DOI:10.1007/s10681-010-0259-z |
| [26] |
LI Y L, ZHAO F C, LI X Z, et al. Analysis of genetic diversity of Pinus elliottii and P. caribaea germplasm resources[J].
Guangdong For Sci Technol, 2014, 30(6): 9-14. 李义良, 赵奋成, 李宪政, 等. 湿地松、加勒比松种质资源遗传多样性分析[J]. 广东林业科技, 2014, 30(6): 9-14. DOI:10.3969/j.issn.1006-4427.2014.06.002 |
| [27] |
ZHOU Y X. The study on epigenetic structure of Panax ginseng[D]. Changchun: Northeast Normal University, 2015: 25-36.
周玉新. 人参的表观遗传结构研究[D]. 长春: 东北师范大学, 2015: 25-36. |
| [28] | KALISZ S, PURUGGANAN M D. Epialleles via DNA methylation:Consequences for plant evolution[J]. Trends Ecol Evol, 2004, 19(6): 309-314. DOI:10.1016/j.tree.2004.03.034 |
| [29] |
ZHONG X F. The study of DNA methylation polymorphism and genetic diversity and copia-like retrotransposon reverse transcriptase of annual wild soybean (Glycine soja) and cultivated soybean (G. max)[D]. Changchun: Northeast Normal University, 2009: 30.
仲晓芳. 一年生野生大豆(Glycine soja)和栽培大豆(G. max)的DNA甲基化多样性和遗传多样性研究及copia-like反转录转座子反转录酶分析[D]. 长春: 东北师范大学, 2009: 30. |
| [30] |
ZHANG Y, CHU H J, ZHANG J J. Comparative research on genetic and epigenetic diversity of salvia miltiorrhiza populations in Qinba Mountain Area[J].
Acta Agric Boreali-Occid Sin, 2012, 21(10): 142-148, 206. 张逸, 褚会娟, 张今今. 秦巴山区丹参居群遗传与表观遗传多样性比较[J]. 西北农业学报, 2012, 21(10): 142-148, 206. |
| [31] | LIRA-MEDEIROS C F, PARISOD C, FERNANDES R A, et al. Epige-netic variation in mangrove plants occurring in contrasting natural environment[J]. PLoS One, 2010, 5(4): e10326 DOI:10.1371/journal.pone.0010326 |
| [32] | TAKATA M, KISHIMA Y, SANO Y. DNA methylation polymorphisms in rice and wild rice strains:Detection of epigenetic markers[J]. Breed Sci, 2005, 55(1): 57-63. DOI:10.1270/jsbbs.55.57 |
| [33] |
LI Y D. Molecular genetic and epigenetic diversity and genetic structure of wild barley[Hordeum brevisubulatum (Trin. ) Link] arti-ficial populations endemic to Songnen Prairie, China[D]. Changchun: Northeast Normal University, 2007: 33-34.
李毅丹. 中国松嫩草原短芒野大麦[Hordeum brevisubulatum (Trin. ) Link]人工种群的分子遗传与表观遗传多样性及其种群遗传结构的研究[D]. 长春: 东北师范大学, 2007: 33-34. |
| [34] | VAUGHN M V, TANURDŽIĆ M, LIPPMAN Z, et al. Epigenetic natural variation in Arabidopsis thaliana[J]. PLoS Biol, 2007, 5(7): e174 DOI:10.1371/journal.pbio.0050174 |
| [35] | RIDDLE N C, RICHARDS E J. Genetic variation in epigenetic inheritance of ribosomal RNA gene methylation in Arabidopsis[J]. Plant J, 2005, 41(4): 524-532. DOI:10.1111/j.1365-313X.2004.02317.x |
| [36] | Ashikawa I. Surveying CpG methylation at 5'-CCGG in the genomes of rice cultivars[J]. Plant Mol Biol, 2001, 45(1): 31-39. DOI:10.1023/A:1006457321781 |
| [37] | WANG Y M, LIN X Y, DONG B, et al. DNA methylation poly-morphism in a set of elite rice cultivars and its possible contribution to inter-cultivar differential gene expression[J]. Cell Mol Biol Lett, 2004, 9(3): 543-556. |
 2018, Vol. 26
2018, Vol. 26



