油菜素内酯(brassinolide, BL)是一种与植物生长发育相关的植物激素,具有参与调节细胞的伸长和分裂[1]、促进根的伸长和种子的萌发[2]、加速叶片细胞的衰老[3]、影响高等植物的开花周期[4]、调节植物的光形态建成[5]、增强光合作用强度[6]、维管束分化[7]、植物的育性、内源激素平衡[6]等作用。对BL作用机制的研究表明,细胞受体通过接受BL激素信号刺激,将信号整合发生级联反应,促进下游靶基因的表达,进而参与调控植物生理生化过程[8]。油菜素内酯受体激酶(brassinosteroid insensitive 1, BRI1)是直接与BL结合的唯一识别受体,在信号转导通路中起BL信号接收作用。
BRI1定位于细胞膜上,由胞外受体区(extra- cellular domain)、跨膜区(transmembrane domain)和胞内激酶区(kinase domain)共3个部分组成[9-10]。植物对BL的响应程度取决于BRI1受体激酶的数量[11]。BL通过与BRI1的胞外区结合,激活其胞内激酶区活性[9, 11-13]。BAK1 (BRI1-associated receptor kinase)与BRI1的胞内激酶区结合,通过BRI1/BAK1激酶复合体的转磷酸作用激活BL信号[14-17],同时抑制因子(BRI1 kinase inhibitor l, BKI1)与BRI1/BAK1复合体解离[18-20],进而BSK激酶(brassinosteroid- signalling kinase)和BSU1磷酸酶(BRI1-suppressor 1)发生磷酸化,使BIN2激酶(brassinosteroid insensitive 2)去磷酸化失活[14, 21],并在细胞核中积累大量具有活性的BZR转录因子,促进BL靶基因表达,最终促进细胞伸长[22]。
竹子具有速生的特点,能够在2~3个月内完成竹株的增高生长,在此过程中赤霉素、吲哚乙酸、油菜素内酯、玉米素和脱落酸等植物生长调节剂含量变化明显,他们之间的平衡对其快速生长发挥着重要调节作用[23-26]。然而,关于竹子植物生长调节剂的信号转导的研究却鲜有报道,尤其是BL的信号转导途径尚未见报道。毛竹(Phyllostachys edulis)是我国种植范围最广,最具经济价值的竹种之一。毛竹基因组草图及其基因组数据库的完成[27-28],为研究竹笋快速生长过程中的BL信号转导提供了便利。本研究以毛竹为材料,对参与毛竹BL信号转导的BL受体激酶同源基因进行全基因组分析与鉴定,包括基因结构特征分析、蛋白质理化性质和亚细胞定位预测、保守结构域分析、进化关系和组织特异性表达分析等。另外,为验证基因在竹笋中的表达情况,采用实时定量PCR方法分析了BL受体激酶基因在不同发育阶段毛竹竹笋中的表达模式, 以期为研究BL受体激酶基因在BL信号转导中的作用奠定基础,为全面揭示毛竹竹笋快速生长的调节机制提供参考依据。
1 材料和方法 1.1 材料以生长良好的毛竹(Phyllostachys edulis)为对象,选取3年生毛竹实生苗,分别取根(当年新发的幼嫩的根)、茎(当年生的幼茎)、未完全展开叶、完全展开叶、叶鞘和笋(高度为2~10 cm),用于基因组织表达特异性分析。从江西南昌野外生长的毛竹,分别取0.2、1.0、3.0和6.7 m高竹笋的基部, 用于检测目的基因在竹笋不同发育阶段的表达情况。样品经液氮处理后,存储于-80℃冰箱中,用于提取RNA。
1.2 RNA提取与cDNA合成利用RNA提取试剂盒(TaKaRa, 日本)提取各样本的总RNA,使用Recombinant DNase I去除残余DNA,并使用反转录试剂盒(Promega, 美国)合成cDNA第一条链,操作参照相关试剂盒说明书进行。
1.3 BL受体激酶同源基因的获取与生物信息学分析以模式植物拟南芥(Arabidopsis thaliana)和水稻(Oryza sativa)中BL受体激酶基因AtBRI1(NC_ 003075.7)、OsBRI1(XP_015621030)、OsBRL1(XP_ 015612606.1)、OsBRL2(XP_015615048.1)和OsBRL3(XP_015650921.1)作为种子序列,利用已有毛竹基因组数据库BambooGDB (http://www.bamboogdb.org/)[28]进行BLAST获取毛竹中BL受体激酶的同源基因序列。使用GSDS (http://gsds.cbi.pku.edu.cn/)分析获得目的基因的结构,用在线软件ProtParam (http://web.expasy.org/protparam/)分析其编码蛋白的理化性质,用WoLF PSORT (http://www.genscript.com/wolf-psort.html)预测目的基因表达蛋白的亚细胞定位。用ClustalW[29-30]比对同源序列并分析保守结构域,使用Neighbor-Joining法构建不同物种基于BL受体激酶的系统发育进化树。利用在线软件TMHMM Server V. 2.0 (http://www.cbs.dtu.dk/services/TMHMM/)预测目的基因编码蛋白的跨膜结构域。
1.4 实时定量PCR根据获取的BL受体激酶基因同源序列,使用Primer Premier 5.0软件筛选设计适宜的定量引物(表 1), 引物由上海生工生物工程技术服务有限公司合成。
| 表 1 实时定量PCR所用引物 Table 1 Primers used in quantitative real-time PCR |
利用qTOWER2.2 (Analytik Jena, 德国) PCR仪进行目的基因表达的定量分析,以毛竹PeNTB基因作为内参[31],每个反应3次重复。反应体系体积为10 μL,包括LightCycler® 480 SYBR Green I Master Mix (Roche, 美国) 5.0 μL,正、反向引物各0.2 μL,cDNA 0.8 μL,ddH2O 3.8 μL。反应程序:95℃6 min;95℃10 s,63℃12 s,共45个循环。利用2–ΔΔCT法[32]分析基因的表达变化情况,采用Excel对数据进行分析并绘图。
2 结果和分析 2.1 基因序列的获得和结构分析通过比对分析,从基因组数据库BambooGDB中共检索得到BL受体激酶基因同源序列12条,其中的PH01000636G0280不具有胞内激酶区的ATP结合保守结构位点,且亚细胞定位预测位点为细胞壁,与BL受体激酶为膜受体蛋白的结构功能不符;PH01001222G0530缺少多肽底物结合位点; PH010- 02609G0090和PH01002795G0230均不具有胞内激酶区的3个保守结构域。因此,以上4条基因序列不再进行分析。
根据编码蛋白的保守结构域,以及与模式植物水稻同源基因相似度和命名分类,将毛竹中具有完整保守结构域的8条BL受体激酶同源基因分为4类(BRL1、BRL2、BRL3和BRL4),分别命名为PeBRL1-1、PeBRL1-2、PeBRL2-1、PeBRL3-1、PeBRL4-1、PeBRL4-2、PeBRL4-3和PeBRL4-4。8个PeBRLs的编码区序列长度为2 577~3 675 bp,对应的基因组序列长度为2 770~3 675 bp。基因结构分析表明,PeBRL1-1、PeBRL2-1、PeBRL4-2和PeBRL4-4不含内含子,PeBRL1-2、PeBRL3-1、PeBRL4-1和PeBRL4-3含有1个内含子,长度分别为193、239、67和103 bp,转录过程中内含子剪接属于GT-AG类型[33](图 1)。
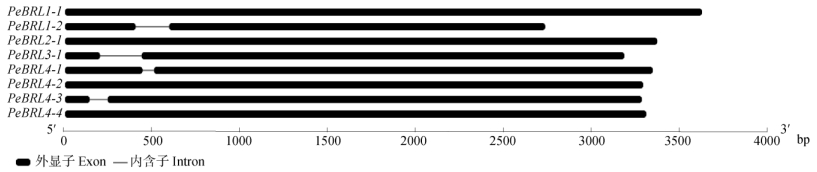
|
图 1 PeBRLs基因结构 Fig. 1 Structure of PeBRLs genes |
PeBRLs编码的肽链长度为858~1 224个氨基酸,分子量为92~130 kDa;编码蛋白的不稳定系数为29.12~34.04,均较稳定;脂肪系数为95.86~ 109.65;平均亲水系数(GRAVY)为-0.067~0.305;理论等电点为5.41~6.42,所编码的蛋白质均呈弱酸性。经过亚细胞定位预测,8个PeBRLs均被定位在细胞膜上,属于典型的膜嵌合蛋白(表 2)。
| 表 2 PeBRLs编码蛋白的理化性质和亚细胞定位预测 Table 2 Putative basic physical and chemical characteristics, and subcellular localization of proteins encoding by PeBRLs |
PeBRL1-1具有2个跨膜结构,跨膜区长度均为23个氨基酸;其余PeBRLs均具有1个跨膜结构, 其中PeBRL3-1的跨膜区长度为20个氨基酸; PeBRL1-2、PeBRL2-1、PeBRL4-1、PeBRL4-2、PeBRL4-3和PeBRL4-4的均为23个氨基酸。
2.3 PeBRLs编码蛋白保守性和进化分析利用ClustalW对PeBRLs编码的氨基酸和模式植物水稻、拟南芥的BRL/BRI氨基酸序列进行比对分析,结果表明,与水稻、拟南芥相类似,推测PeBRLs编码的蛋白在胞外受体区、跨膜区和胞内激酶区高度保守,且具有富亮氨酸重复结构(LRRs); 胞内激酶区保守结构域特殊,是决定BRI1激酶活性的关键区,包括3个特有保守结构域,分别是ATP结合位点(ATP binding site)、多肽底物结合位点(Polypeptide substrate binding site)和活性环结构(Activation loop)(图 2)。其推测编码的蛋白结构域的保守性表明PeBRLs可能与水稻、拟南芥中的同源蛋白具有相似的功能。

|
图 2 毛竹(Pe)、水稻(Os)和拟南芥(At)中BL受体激酶同源蛋白的胞内激酶区保守结构域分析。—: ATP结合位点; =:多肽底物结合位点; ۔۔۔۔۔۔:活性环结构。 Fig. 2 Analysis of kinase domain in homologous proteins of BL receptor kinasefrom Phyllostachys edulis(Pe), Oryza sativa (Os) and Arabidopsis thaliana(At).—: ATP binding site; =: Polypeptide substrate binding site; ۔۔۔۔۔۔: Activation loop. |
以毛竹、水稻和拟南芥中BRI1/BRL1同源蛋白氨基酸序列构建的系统进化树表明,毛竹8个PeBRLs分别被聚类到4个分支。PeBRL1-1、Pe- BRL1-2与OsBRL1, PeBRL2-1与OsBRL2, PeBRL3-1与OsBRL3,PeBRL4-1、PeBRL4-2、PeBRL4-3和PeBRL4-4与OsBRI1和AtBRI1分别聚类到一起, 其中PeBRL4-4与OsBRI1距离最近,而与AtBRI1则距离较远(图 3)。
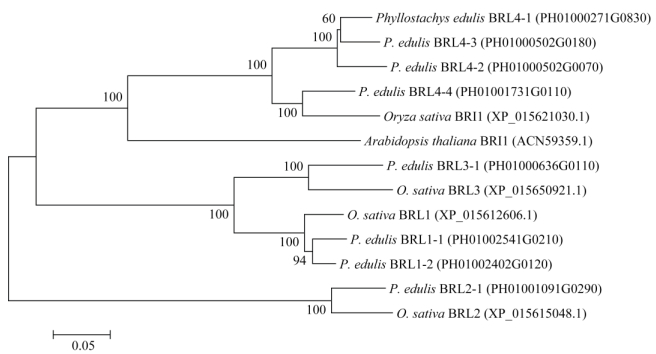
|
图 3 基于BL受体激酶蛋白氨基酸序列的系统发育进化树 Fig. 3 Phylogenetic tree based on amino acid sequences of BL receptor kinase proteins |
组织表达结果表明,PeBRL1-1、PeBRL1-2和PeBRL2-1在根中的表达量较高,约为其他组织的5倍。PeBRL3-1在根、茎、未完全展开叶中的表达量较高,在完全展开叶、鞘和笋中的表达量均较低, 其在未完全展开叶中的表达量高于完全展开叶,推测其可能参与了维管束发育的过程[7]。PeBRL4-1、PeBRL4-2、PeBRL4-3和PeBRL4-4的序列相似度最高,表达模式也相似,表达量均在未完全展开叶和叶鞘中较低,在其他组织中较高,且在完全展开叶的表达量明显高于未完全展开叶(图 4),推测他们可能主要在成熟组织中发挥功能。基因的表达差异, 表明PeBRLs功能的不同,具体情况尚需深入研究。
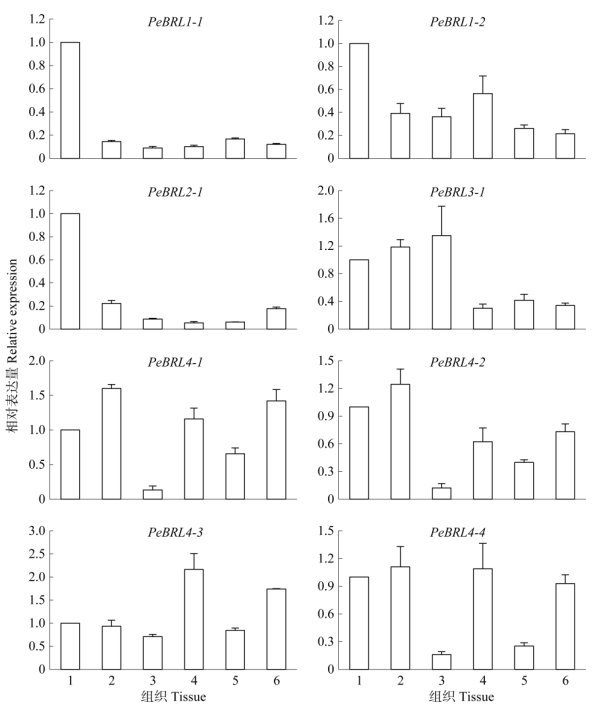
|
图 4 PeBRLs在不同组织中的表达。1:根; 2:茎; 3:未完全展开叶; 4:完全展开叶; 5:叶鞘; 6:笋。 Fig. 4 Expression of PeBRLs in different tissues. 1: Root; 2: Stem; 3: Not fully expanded leaf blade; 4: Fully expanded leaf blade; 5: Leaf sheath; 6: Shoot. |
实时定量PCR结果表明,PeBRLs在笋中的表达模式表现为4种趋势(图 5)。① PeBRL1-1、PeBRL1-2、PeBRL2-1和PeBRL4-4随笋的发育呈先上升后下降再上升的变化趋势,4个基因均以在1.0 m笋中的表达量最高,PeBRL1-1在1.0 m笋中的表达量约为对照(0.2 m)的140倍,而在3.0 m笋中下降至约为对照26倍,在6.7 m笋中又略上升约为对照的33倍,推测PeBRL1-1可能是参与竹笋BL信号传导的关键基因;PeBRL1-2在1.0 m笋中的表达量比0.2 m笋中略有上升,而在3.0和6.7 m笋中均下降,在3.0 m笋中的表达量最低,仅约为对照的1/10,虽然PeBRL1-1和PeBRL1-2属于同一个亚家族且序列相似度较高,但是基因表达差异明显,这可能是由于基因的顺式作用元件差异引起的,5′ UTR Py-rich stretch对基因具有高转录活性[34], 在PeBRL1-1的启动子区含有10个5′ UTR Py-rich stretch,而PeBRL1-2的启动子区仅含有1个; PeBRL2-1和PeBRL4-4表达量变化趋势与PeBRL1-1的相近。② PeBRL3-1随笋的发育呈先上升后下降的变化趋势,PeBRL3-1在1.0 m笋中的表达量达到最高,约为对照的47倍,随后持续下降,但在6.7 m笋中仍约为对照的15倍。③ PeBRL4-1随笋的发育呈先下降后上升的变化趋势,PeBRL4-1在1.0 m和3.0 m笋中的表达量均低于对照(0.2 m笋),以在3.0 m笋中的最低,约为对照的1/2,在6.7 m笋中的略高于对照(0.2 m笋)。④ PeBRL4-2和PeBRL4-3随笋的发育呈持续下降的变化趋势, PeBRL4-2在6.7 m笋中的表达量最低,约为对照的1/10; Pe- BRL4-3在3.0和6.7 m笋中的表达量相近,约为对照的1/5。

|
图 5 PeBRLs在不同发育阶段笋中的表达 Fig. 5 Expression of PeBRLs in bamboo shoots at different development stages |
竹子可再生性强、材性好,被广泛使用于建材、工艺品加工等多个方面,因此研究竹子生长发育特点及其速生机制等对于更好的利用竹材具有重要的意义。研究表明,赤霉素和油菜素甾醇在决定植物高度方面是最重要的调控因子[8, 35-37],油菜素内酯受体激酶在BL信号转导过程中发挥了极其重要的作用[38]。利用模式植物拟南芥、水稻BL受体激酶基因[39-40]同源比对,在毛竹中获得了12条同源基因,通过进一步分析其保守结构域,确定了编码蛋白具有完整BL受体激酶结构的基因8条,分别是水稻、拟南芥的2倍和8倍,这可能是与毛竹在进化过程中经历了染色体加倍事件有关[27]。
基因结构分析表明,毛竹8个PeBRLs基因长度差异较大(最大相差1 098 bp),其中4个基因不含内含子,4个基因含有1个内含子,分别属于4个亚家族。PeBRLs呈弱酸性,性质稳定;均被定位在细胞膜上,作为膜受体发挥其生物功能[21]。由此表明,虽然8个PeBRLs基因结构、理化性质存在差异,它们可能在细胞中发挥着不同的作用,但作为同一家族的不同成员,其基本的生物学功能是相似的。组织特异性表达结果表明,8个PeBRLs为组成型表达模式,在毛竹实生苗的根、茎和笋中的表达均高于在未完全展开叶、完全展开叶和叶鞘中的表达,这与毛竹不同组织的RNA-Seq表达谱相一致[27],表明PeBRLs在毛竹生长过程中发挥着重要作用。
BL作为一种微量高效的植物激素,具有参与调节细胞的伸长和分裂的作用[1],BL受体激酶是与BL直接结合的膜受体蛋白,在响应BL调节过程中发挥唯一识别受体的作用。转基因拟南芥过表达BRI1-GFP结合蛋白可以通过增加BL结合位点,增加对BL的响应[11]。竹笋中8个PeBRLs的表达随发育进程呈现4种变化趋势,如PeBRL1-1、PeBRL3-1的表达量均呈现先上升后下降的变化趋势,且在1.0 m笋中达到最高,分别为对照的140倍和47倍,而其他基因的表达量变化范围均在对照的7倍以内。这可能是由于1.0 m高的笋处于生长的指数增长阶段,在响应发育的调控下BL受体激酶基因的表达量升高,增加膜受体激酶数量和BL底物结合位点[11],进一步通过提高BL的结合效率,在BL作用下促进笋的快速伸长,以实现毛竹笋期快速生长。另外,也有BL受体激酶基因(PeBRL4-2和PeBRL4-3)的表达随着竹笋高度的增加而呈现下降的趋势,其原因有待于深入研究来揭示。
| [1] | CLOUSE S D, ZUREK D. Molecular analysis of brassinolide action in plant growth and development[M]//CUTLER H G, YOKOTA T, ADAM G. Brassinosteroids: Chemistry, Bioactivity, and Applications. Washington DC: American Chemical Society, 1991: 122-140. doi: 10.1021/bk-1991-0474.ch011. |
| [2] | DOMAGALSKA M A, SCHOMBURG F M, AMASINO R M, et al. Attenuation of brassinosteroid signaling enhances FLC expression and delays flowering[J]. Development, 2007, 134(15): 2841-2850. DOI:10.1242/dev.02866 |
| [3] | CHOE S, CHUNG Y. The regulation of brassinosteroid biosynthesis in Arabidopsis[J]. Crit Rev Plant Sci, 2013, 32(6): 396-410. DOI:10.1080/07352689.2013.797856 |
| [4] | HOU X L, HU W W, SHEN L S, et al. Global identification of DELLA target genes during Arabidopsis flower development[J]. Plant Physiol, 2008, 147(3): 1126-1142. DOI:10.1104/pp.108.121301 |
| [5] | NAKAMOTO D, IKEURA A, ASAMI T, et al. Inhibition of brassi-nosteroid biosynthesis by either a dwarf4 mutation or a brassinosteroid biosynthesis inhibitor rescues defects in tropic responses of hypocotyls in the Arabidopsis mutant nonphototropic hypocotyl 4[J]. Plant Physiol, 2006, 141(2): 456-464. DOI:10.1104/pp.105.076273 |
| [6] | SASSE J M. Physiological actions of brassinosteroids:An update[J]. J Plant Growth Regul, 2003, 22(4): 276-288. DOI:10.1007/s00344-003-0062-3 |
| [7] | NOMURA T, SATO T, BISHOP J, et al. Accumulation of 6 deoxo-cathasterone and 6 deoxocastasterone in Arabidopsis, pea and tomato is suggestive of common rate-limiting steps in brassinosteroid biosyn-thesis[J]. Phytochemistry, 2001, 57(2): 171-178. DOI:10.1016/S0031-9422(00)00440-4 |
| [8] | MANDAVA N B. Plant growth-promoting brassinosteroids[J]. Annu Rev Plant Physiol Plant Mol Biol, 1988, 39(1): 23-52. DOI:10.1146/annurev.pp.39.060188.000323 |
| [9] | LI J M, CHORY J. A putative leucine-rich repeat receptor kinase involved in brassinosteroid signal transduction[J]. Cell, 1997, 90(5): 929-938. DOI:10.1016/S0092-8674(00)80357-8 |
| [10] | FRIEDRICHSEN D M, JOAZEIRO C A P, LI J M, et al. Brassino-steroid-insensitive-1 is a ubiquitously expressed leucine-rich repeat receptor serine/threonine kinase[J]. Plant Physiol, 2000, 123(4): 1247-1255. DOI:10.1104/pp.123.4.1247 |
| [11] | WANG Z Y, SETO H, FUJIOKA S, et al. BRI1 is a critical component of a plasma-membrane receptor for plant steroids[J]. Nature, 2001, 410(6826): 380-383. DOI:10.1038/35066597 |
| [12] | SHE J, HAN Z F, KIM T W, et al. Structural insight into brassi-nosteroid perception by BRI1[J]. Nature, 2011, 474(7352): 472-476. DOI:10.1038/nature10178 |
| [13] | HOTHORN M, BELKHADIR Y, DREUX M, et al. Structural basis of steroid hormone perception by the receptor kinase BRI1[J]. Nature, 2011, 474(7352): 467-471. DOI:10.1038/nature10153 |
| [14] | LI J M, NAM K H. Regulation of brassinosteroid signaling by a GSK3/SHAGGY-like kinase[J]. Science, 2002, 295(5558): 1299-1301. DOI:10.1126/science.1065769 |
| [15] | WANG X F, KOTA U, HE K, et al. Sequential transphosphorylation of the BRI1/BAK1 receptor kinase complex impacts early events in brassinosteroid signaling[J]. Dev Cell, 2008, 15(2): 220-235. DOI:10.1016/j.devcel.2008.06.011 |
| [16] | CLOUSE S D. Brassinosteroid signal transduction:From receptor kinase activation to transcriptional networks regulating plant develop-ment[J]. Plant Cell, 2011, 23(4): 1219-1230. DOI:10.1105/tpc.111.084475 |
| [17] | GOU X P, YIN H J, HE K, et al. Genetic evidence for an indispensable role of somatic embryogenesis receptor kinases in brassinosteroid signaling[J]. PLoS Genet, 2012, 8(1): e1002452 DOI:10.1371/journal.pgen.1002452 |
| [18] | JAILLAIS Y, HOTHORN M, BELKHADIR Y, et al. Tyrosine phosphorylation controls brassinosteroid receptor activation by trig-gering membrane release of its kinase inhibitor[J]. Genes Dev, 2011, 25(3): 232-237. DOI:10.1101/gad.2001911 |
| [19] | WANG X L, CHORY J. Brassinosteroids regulate dissociation of BKI1, a negative regulator of BRI1 signaling, from the plasma membrane[J]. Science, 2006, 313(5790): 1118-1122. DOI:10.1126/science.1127593 |
| [20] | WANG S H, KUREPA J, HASHIMOTO T, et al. Salt stress-induced disassembly of Arabidopsis cortical microtubule arrays involves 26S proteasome-dependent degradation of SPIRAL1[J]. Plant Cell, 2011, 23(9): 3412-3427. DOI:10.1105/tpc.111.089920 |
| [21] | KIM T W, WANG Z Y. Brassinosteroid signal transduction from receptor kinases to transcription factors[J]. Annu Rev Plant Biol, 2010, 61(1): 681-704. DOI:10.1146/annurev.arplant.043008.092057 |
| [22] | YAMAMURO C, IHARA Y, WU X, et al. Loss of function of a rice brassinosteroid insensitive1 homolog prevents internode elongation and bending of the lamina joint[J]. Plant Cell, 2000, 12(9): 1591-1606. DOI:10.2307/3871176 |
| [23] |
DING X C. Dynamic analysis for endogenous phytohormones of bamboo shoots (Phyllostachys heterocycla var. pubescens) during different growth and differentiation stage[J].
J Bamboo Res, 1997, 16(2): 53-62. 丁兴萃. 毛竹笋体生长发育过程中内源激素的动态分析[J]. 竹子研究汇刊, 1997, 16(2): 53-62. |
| [24] |
DONG L L, ZHAO H S, LI L C, et al. Expression characteristics and promoter activity analysis of PeSCL3 gene from Phyllostachys edulis[J].
J Trop Subtrop Bot, 2016, 24(3): 252-258. 董丽莉, 赵韩生, 李利超, 等. 毛竹PeSCL3基因表达特征及其启动子活性研究[J]. 热带亚热带植物学报, 2016, 24(3): 252-258. DOI:10.11926/j.issn.1005-3395.2016.03.002 |
| [25] |
ZHANG C L. The comprehensive analysis of shoot-culm and study of auxin-related genes of Phyllostachys edulis[D]. Beijing: Chinese Academy of Forestry Sciences, 2014: 24-30.
张春玲. 毛竹笋-竹生长发育过程系统分析与生长素相关基因研究[D]. 北京: 中国林业科学研究院, 2014: 24-30. |
| [26] | JIANG K Y, ZHOU M B. Recent advances in bamboo molecular biology[J]. J Trop Subtrop Bot, 2014, 22(6): 632-642. DOI:10.11926/j.issn.1005-3395.2014.06.012 |
| [27] | PENG Z H, LU Y, LI L B, et al. The draft genome of the fast-growing non-timber forest species moso bamboo (Phyllostachys heterocycla)[J]. Nat Genet, 2013, 45(4): 456-461. DOI:10.1038/ng.2569 |
| [28] | ZHAO H S, PENG Z H, FEI B H, et al. BambooGDB:A bamboo genome database with functional annotation and an analysis platform[J]. Database, 2014, 2014: bau006 DOI:10.1093/database/bau006 |
| [29] | THOMPSON J D, HIGGINS D G, GIBSON T J. CLUSTAL W:Improving the sensitivity of progressive multiple sequence alignment through sequence weighting, position-specific gap penalties and weight matrix choice[J]. Nucl Acid Res, 1994, 22(22): 4673-4680. DOI:10.1093/nar/22.22.4673 |
| [30] | NEBERT D W, GONZALEZ F J. P450 genes:Structure, evolution, and regulation[J]. Annu Rev Biochem, 1987, 56(1): 945-993. DOI:10.1146/annurev.bi.56.070187.004501 |
| [31] | FAN C J, MA J M, GUO Q R, et al. Selection of reference genes for quantitative real-time PCR in bamboo (Phyllostachys edulis)[J]. PLoS One, 2013, 8(2): e56573 DOI:10.1371/journal.pone.0056573 |
| [32] | LIVAK K J, SCHMITTGEN T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-∆∆CT method[J]. Methods, 2001, 25(4): 402-408. DOI:10.1006/meth.2001.1262 |
| [33] | MOORE M J, QUERY C C, SHARP P A. Splicing of precursors to mRNA by the spliceosome[M]//GESTELAND R F, ATKINS J F. The RNA World. Cold Spring Harbor: Cold Spring Harbor Laboratory Press, 1993: 303-357. |
| [34] | ZHANG J, ZHANG X Y, WANG Y Q, et al. Characterization of sequence elements from Malvastrum yellow vein betasatellite regu-lating promoter activity and DNA replication[J]. Virol J, 2012, 9: 234 DOI:10.1186/1743-422X-9-234 |
| [35] | CLOUSE S D, SASSE J M. Brassinosteroids:Essential regulators of plant growth and development[J]. Annu Rev Plant Physiol Plant Mol Biol, 1998, 49: 427-451. DOI:10.1146/annurev.arplant.49.1.427 |
| [36] | TAIZ L, ZEIGER E. ZEIGER E. Plant Physiology[M]. 3rd ed. Sunderland: Sinauer Associates, 2002: 461-488. |
| [37] | FUJIOKA S, YOKOTA T. Biosynthesis and metabolism of brassino-steroids[J]. Annu Rev Plant Biol, 2003, 54: 137-164. DOI:10.1146/annurev.arplant.54.031902.134921 |
| [38] | HONG Z, UEGUCHI-TANAKA M, UMEMURA K, et al. A rice brassinosteroid-deficient mutant, ebisu dwarf (d2), is caused by a loss of function of a new member of cytochrome P450[J]. Plant Cell, 2003, 15(12): 2900-2910. DOI:10.1105/tpc.014712 |
| [39] | HATEGAN L, GODZA B, KOZMA-BOGNAR L, et al. Differential expression of the brassinosteroid receptor-encoding BRI1 gene in Arabidopsis[J]. Planta, 2014, 239(5): 989-1001. DOI:10.1007/s00425-014-2031-4 |
| [40] | ZHAO H S, DONG L L, SUN H Y, et al. Comprehensive analysis of multi-tissue transcriptome data and the genome-wide investigation of GRAS family in Phyllostachys edulis[J]. Sci Rep, 2016, 6: 27640 DOI:10.1038/srep27640 |
 2018, Vol. 26
2018, Vol. 26


