磷酸烯醇式丙酮酸羧化酶(phosphoenolpyruvate carboxylase, PEPC, EC 4.1.1.31)是一种变构调节酶, 它的主要功能是在Mg2+和HCO3-存在的情况下,催化磷酸烯醇式丙酮酸(phosphoenolpyruvate, PEP)的β-C羧基化并生成草酰乙酸(oxaloacetate, OAA)和无机磷酸(inorganic phosphate, Pi)[1-4]。除动物和真菌外,PEPC广泛存在于植物、藻类、蓝细菌、光合细菌及一些非光合细菌中[5]。目前已有多个植物PEPC得到鉴定,在拟南芥(Arabidopsis thaliana)中有Atppc1、Atppc2、Atppc3和Atppc4等4个编码基因[6],在玉米(Zea mays)[7]、甘蔗(Saccharum spp.)[8]、大豆(Glycine max)[9]、水稻(Oryza sativa)[5]等植物中也报道了多个PEPC基因。早期报道了大豆PEPC基因家族的GmPEPC1、GmPEPC4、GmPEPC7、GmPEPC16和GmPEPC17等5个成员,后续报道了10个GmPEPC家族成员,并对这些基因进行了重命名[4, 9]。在栽培花生(Arachis hypogaea)品种‘E11’中也克隆得到AhPEPC1-5等5个成员[10]。植物PEPC参与多种重要代谢途径,不仅作为光合作用的关键酶参与CO2的固定[11],还参与氨基酸代谢中碳骨架的回补[12-13]、氮元素的积累[13-14]、油脂的合成与积累[15-18]等过程,起到调控植物种子的形成与萌发、果实成熟、植物叶片气孔开闭,维持细胞酸碱平衡以及提高植物抗逆性等作用[12, 19]。
花生是重要的油料作物之一,种子含油量高达45%~56%,蛋白质含量达到24%[20-21]。四倍体的栽培品种花生基因组庞大而复杂,目前已经测序完成注释并公布的是2个二倍体花生祖先种野生蔓花生(Arachis duranensis)和A. ipaёnsis (即花生属A基因组和B基因组)的全基因组序列[21-23]。本研究通过分析蔓花生的数据库信息,找到AdPEPC家族基因,并分析其结构、序列特征及进化关系,同时通过检测其不同成员在各种组织中的表达,为进一步探讨AdPEPC基因参与花生生长过程中物质积累和代谢调控的功能机制奠定基础。
1 材料和方法 1.1 材料本研究所用二倍体祖先种蔓花生(Arachis dura-nensis)新鲜根、茎、叶、花等材料,于2016年5月上旬采自广东省农业科学院白云区钟落潭种植基地,采集后立即用液氮速冻,-80℃冰箱保存备用。AdPEPC基因序列信息从花生基因组数据库PeanutBase(http://peanutbase.org/)[23]获得。
1.2 AdPEPC基因的查询和确认以拟南芥AtPPC1 (AT1G53310)、AtPPC2 (AT2-G42600)、AtPPC3 (AT3G14940)、AtPPC4 (AT1G-68750)等4个PEPC蛋白序列[6]对花生基因组数据库PeanutBase (http://peanutbase.org/)进行搜索,在蔓花生基因组中查找同源蛋白的相应基因序列作为候选基因。将候选基因编码的蛋白序列在PFAM (http://pfam.xfam.org/)和SMART (http://smart.embl-heidelberg.de/)蛋白结构域在线分析预测网站上分析候选蛋白结构域,存在PEPcase (PF00311)结构域的确认为PEPC蛋白家族成员,将下载得到的所有核苷酸序列和氨基酸序列以FASTA格式保存。
1.3 AdPEPC基因家族的染色体定位和结构分析用Gene Structure Display Server (GSDS)在线网站(http://gsds.cbi.pku.edu.cn/)[24]进行基因外显子、内含子结构分析。使用MapInspect软件进行染色体定位分析。
1.4 AdPEPC蛋白的生物信息学分析使用DNAMAN软件进行多序列比对分析;用MEGA6.0软件对蔓花生的PEPC与不同物种的PEPC氨基酸序列进行同源比对后,使用N-J法(邻接法)构建系统进化树。使用在线工具ExPASy (http://www.expasy.ch/tools/protparam.html)中的Prot-Param (http://web.expasy.org/protparam/)进行理化性质预测。
1.5 总RNA提取与基因表达分析取蔓花生生长旺盛期的根、茎、叶、花等组织材料,采用OMEGA公司的Plant RNA Kit试剂盒提取总RNA。提取的RNA用1%的琼脂糖凝胶电泳检测其完整度,并用核酸仪测定浓度及纯度。使用TIANGEN公司的Fast Quant RT Kit (with gDNase)试剂盒进行反转录得到cDNA。荧光定量反应体系及程序参照天根公司的Fast Quant RT Kit (with gDNase)试剂盒说明书,在LightCycler480Ⅱ(Roche)仪器上进行。反应体系总体积为20 μL, 包含10 μL的PCR Mix (SYBR Green),正反向引物各0.8 μL,cDNA模板2 μL,H2O 6.4 μL。反应程序为95℃预变性3 min;然后95℃变性10 s, 60℃退火15 s, 72℃延伸20 s,共45个循环。AdPEPC基因特异引物及18S rRNA内参引物见表 1。
| 表 1 荧光定量PCR引物 Table 1 Primers for fluorescent quantitative PCR |
利用拟南芥PEPC蛋白序列搜索蔓花生基因组数据库,共获得9个AdPEPC基因序列,基因座位号分别为Aradu.TPI8X、Aradu.H5W76、Aradu. A52DW、Aradu.H2SP1、Aradu.3E0D8、Aradu.5IN8U、Aradu.2E0TL、Aradu.HLF4L和Aradu.D77KI,根据基因组数据库中的注释分别命名(表 2)。这9个AdPEPC分别分布在第3、5、7、8、9、10号染色体上(图 1: A)。基因结构分析显示,这些基因的序列长度差异较大,总长度为3 584~1 2956 bp,包含多个外显子和内含子,ORF长度为702~3 168 bp (图 1: B)。
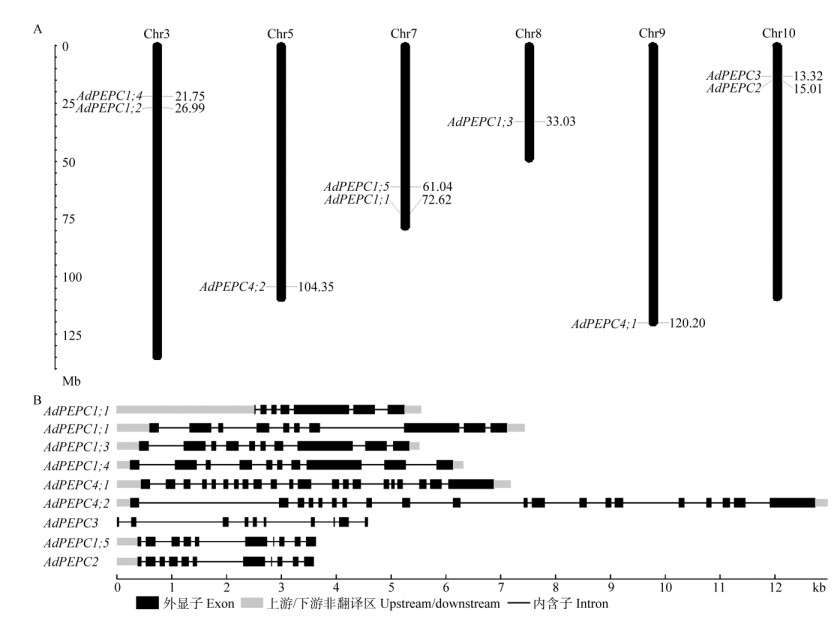
|
图 1 蔓花生AdPEPC家族基因的染色体分布(A)和基因结构(B) Fig. 1 Chromosome distribution (A) and structure (B) of AdPEPC family genes in Arachis duranensis |
| 表 2 蔓花生AdPEPC基因结构 Table 2 Structure of AdPEPC genes in Arachis duranensis |
对9个蔓花生AdPEPC基因、4个拟南芥AtPEPC基因、10个大豆GmPEPC基因和来自大肠杆菌的PEPC编码的氨基酸序列构建进化树(图 2)。结果表明,AdPEPC基因可以分为3类,第一类基因(AdPEPC1;1、AdPEPC1;2、AdPEPC1;3和AdPEPC1;4)编码的氨基酸序列同源性较高,达60.91%~91.53%, 进化关系上与拟南芥和大豆中的植物型PEPC聚为一支,属于植物型PEPC;第二类的AdPEPC4;1和AdPEPC4;2基因编码的氨基酸序列间的同源性为78.41%,进化关系上与拟南芥和大豆中的细菌型PEPC以及大肠杆菌中的PEPC聚为一支,属于细菌型PEPC;第三类基因(AdPEPC3、AdPEPC1;5和AdPEPC2)编码的氨基酸序列与上述两个分支进化距离较远,与植物型和细菌型PEPC的氨基酸序列同源性不足20%,这类基因编码的蛋白明显比前两类小很多,因此将其归为序列较短的PEPC类型。
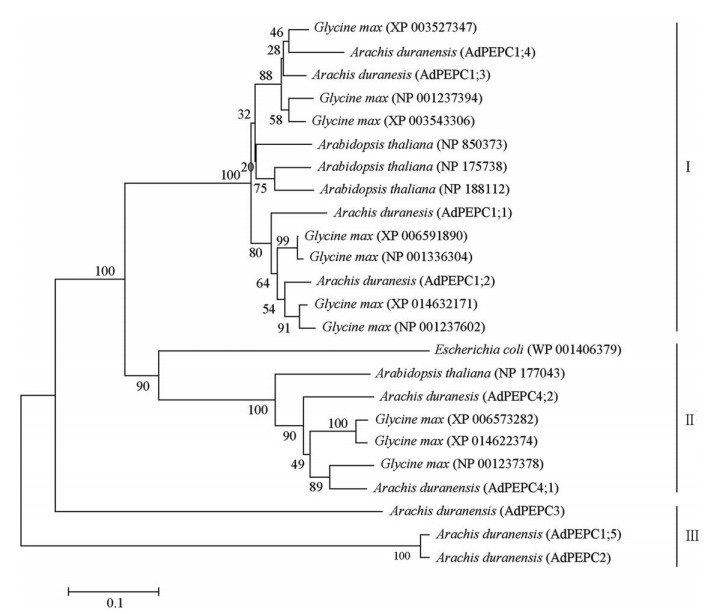
|
图 2 蔓花生和其它物种24个PEPC蛋白的进化树。分支上的数字为自展值。 Fig. 2 Phylogenetic tree of 24 PEPC proteins in Arachis duranensis and other species. Numbers on branches present bootstrap values. |
对不同物种PEPC的氨基酸序列分析表明这些蛋白有多个保守的功能结构域[4, 6, 25-26],包括N-末端磷酸化位点、PEP结合位点、葡萄糖-6-磷酸结合位点、Mg2+结合位点、HCO3-结合位点和天冬氨酸结合位点等。分析表明,蔓花生的第一类AdPEPC蛋白C-末端具有QNTG序列,其中大部分在N-端有1个反向磷酸化位点(-SIDAQL-),符合植物型PEPC的序列特征[4, 25-26];第二类AdPEPC蛋白C-末端为R/KNTG序列,属于细菌型PEPC[3, 25],这两种类型AdPEPC都含有主要的保守结构域(图 3)。而序列较短的第三类AdPEPC蛋白无反向磷酸化位点以及C-末端保守域,既不属于细菌型PEPC,也不属于植物型PEPC,只含有HCO3-结合位点和部分PEP结合位点,多个关键保守序列缺失。其中AdPEPC1;5和AdPEPC2的C-末端3个氨基酸序列中含有不带N的LG同源序列,可能属于古细菌型PEPC[26]。

|
图 3 AdPEPC蛋白家族成员的多序列比对及保守结构域分析。保守结构域:VLTAHPT (Box Ⅰ); DXRQE (Box Ⅱ); GYSDSG/AKDAG (Box Ⅲ); FHGRGGXXGRGG (Box Ⅳ); GSRPXXR (Box Ⅴ)。 Fig. 3 Multiple alignment and conserved domain analysis of AdPEPC in Arachis duranensis. Conserved domains: VLTAHPT (Box Ⅰ); DXRQE (Box Ⅱ); GYSDSG/AKDAG (Box Ⅲ); FHGRGGXXGRGG (Box Ⅳ); GSRPXXR (Box Ⅴ). |
对9个AdPEPC的理化性质进行预测分析, 结果表明(表 3),植物型AdPEPC分子量为78.4~ 112.29 kD,细菌型AdPEPC分子量为117.32~ 118.48 kD, 序列较短类型AdPEPC的分子量为25.25~54.66 kD。大部分AdPEPC蛋白的等电点为4.94~6.85,为带负电荷蛋白,只有细菌型的AdPEPC4;2等电点为8.2,带正电荷。大部分AdPEPC蛋白的不稳定系数小于40,预测为稳定蛋白,只有植物型的AdPEPC1;1和序列较短的AdPEPC3预测为不稳定蛋白。对蛋白进行亲/疏水性预测,9个AdPEPC的GEAVY值均为负值,说明这些蛋白均为亲水性蛋白。
| 表 3 蔓花生9个PEPC家族成员蛋白的理化性质 Table 3 Physical and chemical properties of 9 PEPCs in Arachis duranensis |
对蔓花生根、茎、叶、花等组织中AdPEPC基因进行实时荧光定量PCR分析,结果表明(图 4), 除AdPEPC1;2和AdPEPC4;2在茎中的表达量较高外,其他基因尤其是AdPEPC2、AdPEPC1;5和AdPEPC1;3在花中的表达量明显高于其他组织;除AdPEPC1;1、AdPEPC1;2、AdPEPC4;1和AdPEPC1;3在叶中的表达量略高于根之外,其他基因在根中的表达量高于叶片;AdPEPC1;5基因在叶中未检测到表达;AdPEPC3在根、茎、叶、花中均检测不到表达。
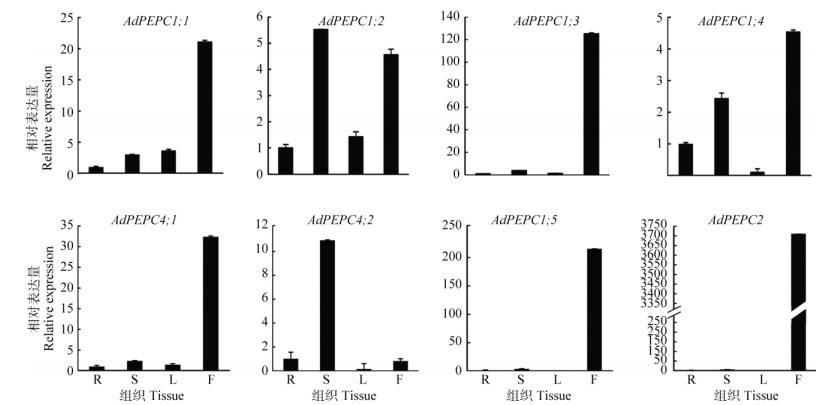
|
图 4 AdPEPC基因家族在蔓花生不同组织中的表达。18S rRNA为内参基因; R:根; S:茎; L:叶; F:花。 Fig. 4 Expression of family genes in different tissues of Arachis duranensis. 18S rRNA was used as reference gene; R: Root; S: Stem; L: Leaf; F: Flower. |
植物PEPC的保守功能结构域是影响PEPC活性的关键区域,本研究结果表明,大部分AdPEPC具有与大肠杆菌、拟南芥、水稻、甘蔗和大豆等物种PEPC相似的N-末端、C-末端和BoxⅠ~Ⅴ等典型保守序列特征[4, 6-8, 25],大部分AdPEPC含有在大豆PEPC中经实验验证的功能结合位点[4](图 3)。除植物型和细菌型AdPEPC外,在蔓花生中存在3个序列较短的AdPEPC,由于大段序列的缺失,与植物型和细菌型AdPEPC的氨基酸序列同源性很低。这些差异可预示不同的家族成员可能具有不同的功能和作用模式。
有研究比较了栽培花生品种中5个AhPEPC基因在组织中的表达情况,不同基因在不同的花生品种中表达模式存在较大差异,部分成员在根、叶或种子发育部分时期表达量较高,但未对花中的表达进行检测[10, 27]。本研究结果表明,AdPEPC家族基因大部分成员在花和茎中的表达量较高,这可能与蔓花生生长缓慢、叶片细小而蔓生茎较发达等生长特点有关,同时盛花期的花组织也处于代谢相关基因表达活跃的发育阶段。值得注意的是,AdPEPC1;5和AdPEPC2在花中的表达量远高于其他组织,这2个基因编码的氨基酸序列特征与进化上较保守的古细菌型PEPC相似[26, 28]。古细菌型PEPC通常分子量较小,具有不同于其他PEPC的变构调节特征[28], 但催化机制与其他PEPC相同[28]。有研究表明,一些细菌型PEPC基因在高等植物花粉等组织中有明显高于根、茎、叶等组织的表达模式,推测这些基因的功能对于花粉发育具有重要意义[29],蔓花生中在花组织高表达的AdPEPC也可能在花发育过程具有较专一的特殊功能。序列最短的AdPEPC3在4种组织中均检测不到表达,该基因编码的氨基酸序列中多个保守序列及末端特征序列缺失,基因组数据注释中无上游5′非翻译区(5′ UTR)起始调控序列, 推测该基因为假基因。关于这些基因的功能需要在后续的研究中进行更为深入的分析。
| [1] | O'Leary B, Rao S K, Plaxton W C. Phosphorylation of bacterial-type phosphoenolpyruvate carboxylase at Ser425 provides a further tier of enzyme control in developing castor oil seeds[J]. Biochem J, 2011, 433(1): 65-74. DOI:10.1042/BJ20101361 |
| [2] | IZUI K, MATSUMURA H, FURUMOTO T, et al. Phosphoenol pyruvate carboxylase:A new era of structural biology[J]. Annu Rev Plant Biol, 2004, 55: 69-84. DOI:10.1146/annurev.arplant.55.031903.141619 |
| [3] |
GUAN J J, CHEN J Q, WANG X F. Bioinformatic analysis on bacterial-type phosphoenolpyruate carboxylase in plants[J].
Acta Agric Zhejiang, 2011, 23(2): 203-208. 关晶晶, 陈锦清, 汪小福. 植物细菌型磷酸烯醇式丙酮酸羧化酶基因的生物信息学分析[J]. 浙江农业学报, 2011, 23(2): 203-208. DOI:10.3969/j.issn.1004-1524.2011.02.002 |
| [4] | WANG N, ZHONG X J, CONG Y H, et al. Genome-wide analysis of phosphoenolpyruvate carboxylase gene family and their response to abiotic stresses in soybean[J]. Sci Rep, 2016, 6: 38448 DOI:10.1038/srep38448 |
| [5] | MURAMATSU M, SUZUKI R, YAMAZAKI T, et al. Comparison of plant-type phosphoenolpyruvate carboxylases from rice:Identification of two plant-specific regulatory regions of the allosteric enzyme[J]. Plant Cell Physiol, 2015, 56(3): 468-480. DOI:10.1093/pcp/pcu189 |
| [6] | SÁNCHEZ R, CEJUDO F J. Identification and expression analysis of a gene encoding a bacterial-type phosphoenolpyruvate carboxylase from Arabidopsis and rice[J]. Plant Physiol, 2003, 132(2): 949-957. DOI:10.1104/pp.102.019653 |
| [7] | DONG L Y, MASUDA T, KAWAMURA T, et al. Cloning, expression, and characterization of a root-form phosphoenolpyruvate carboxylase from Zea mays:Comparison with the C4-form enzyme[J]. Plant Cell Physiol, 1998, 39(8): 865-873. DOI:10.1093/oxfordjournals.pcp.a029446 |
| [8] | BESNARD G, PIN ON G, D'HONT A, et al. Characterisation of the phosphoenolpyruvate carboxylase gene family in sugarcane (Saccharum spp.)[J]. Theor Appl Genet, 2003, 107(3): 470-478. DOI:10.1007/s00122-003-1268-2 |
| [9] | SULLIVAN S, JENKINS G I, NIMMO H G. Roots, cycles and leaves:Expression of the phosphoenolpyruvate carboxylase kinase gene family in soybean[J]. Plant Physiol, 2004, 135(4): 2078-2087. DOI:10.1104/pp.104.042762 |
| [10] | YU S L, PAN L J, YANG Q L, et al. Identification and expression analysis of the phosphoenolpyruvate carboxylase gene family in peanut (Arachis hypogaea L.)[J]. Agric Sci China, 2010, 9(4): 477-487. DOI:10.1016/S1671-2927(09)60120-6 |
| [11] | KANDOI D, MOHANTY S, GOVINDJEE, et al. Towards efficient photosynthesis:Overexpression of Zea mays phosphoenolpyruvate carboxylase in Arabidopsis thaliana[J]. Photosynth Res, 2016, 130(1/2/3): 47-72. DOI:10.1007/s11120-016-0224-3 |
| [12] |
ZHANG X J, WANG G C, HE L W, et al. Cloning and analysis of the phosphoenolpyruvate carboxylase gene of Porphyra haitanesis (Rho-dophyta)[J].
Mar Sci, 2011, 35(4): 70-76. 张晓娟, 王广策, 何林文, 等. 坛紫菜磷酸烯醇式丙酮酸羧化酶基因的克隆与序列分析[J]. 海洋科学, 2011, 35(4): 70-76. |
| [13] | MASUMOTO C, MIYAZAWA S I, OHKAWA H, et al. Phospho-enolpyruvate carboxylase intrinsically located in the chloroplast of rice plays a crucial role in ammonium assimilation[J]. Proc Natl Acad Sci USA, 2010, 107(11): 5226-5231. DOI:10.1073/pnas.0913127107 |
| [14] | SUGIHARTO B, SUGIYAMA T. Effects of nitrate and ammonium on gene expression of phosphoenolpyruvate carboxylase and nitrogen metabolism in maize leaf tissue during recovery from nitrogen stress[J]. Plant Physiol, 1992, 98(4): 1403-1408. DOI:10.1104/pp.98.4.1403 |
| [15] |
ZHANG Z Q, WANG J M, WANG X J, et al. The characteristics of PEPCase activity and accumulation of oil, protein and major protein subunits during seed development of rape (Brassica napus)[J].
Chin J Oil Crop Sci, 2009, 31(1): 14-18. 张占琴, 王金梅, 王学军, 等. 油菜籽粒发育过程中PEPCase活性与油脂、蛋白及亚基积累的特点[J]. 中国油料作物学报, 2009, 31(1): 14-18. DOI:10.3321/j.issn:1007-9084.2009.01.003 |
| [16] | SEBEI K, OUERGHI Z, KALLEL H, et al. Evolution of phosphor-enolpyruvate carboxylase activity and lipid content during seed matu-ration of two spring rapeseed cultivars (Brassica napus L.)[J]. Compts Rendus Biol, 2006, 329(9): 719-725. DOI:10.1016/j.crvi.2006.06.002 |
| [17] | GENNIDAKIS S, RAO S, GREENHAM K, et al. Bacterial-and plant-type phosphoenolpyruvate carboxylase polypeptides interact in the hetero-oligomeric Class-2 PEPC complex of developing castor oil seeds[J]. Plant J, 2007, 52(5): 839-849. DOI:10.1111/j.1365-313X.2007.03274.x |
| [18] | PAN L J, YANG Q L, CHI X Y, et al. Functional analysis of the phosphoenolpyruvate carboxylase on the lipid accumulation of peanut (Arachis hypogaea L.) seeds[J]. J Integr Agric, 2013, 12(1): 36-44. DOI:10.1016/S2095-3119(13)60203-8 |
| [19] |
ZHANG Y H, MENG Q L, JI C L, et al. Effects of aluminum stress on photosynthesis and active oxygen metabolism in C4 Ppc transgenic rice[J].
Biotechnol Bull, 2014(7): 81-85. 张云华, 孟庆玲, 计成林, 等. 铝胁迫对转C4磷酸烯醇式丙酮酸羧化酶基因水稻光合作用和活性氧代谢的影响[J]. 生物技术通报, 2014(7): 81-85. DOI:10.13560/j.cnki.biotech.bull.1985.2014.07.024 |
| [20] |
LIAO B S. Simple analysis of Chinese peanut oil industry competi-tiveness[J].
J Peanut Sci, 2003, 32(S1): 11-15. 廖伯寿. 中国花生油脂产业竞争力浅析[J]. 花生学报, 2003, 32(S1): 11-15. DOI:10.14001/j.issn.1002-4093.2003.s1.003 |
| [21] | CHEN X P, LI H J, PANDEY M K, et al. Draft genome of the peanut A-genome progenitor (Arachis duranensis) provides insights into geocarpy, oil biosynthesis, and allergens[J]. Proc Natl Acad Sci USA, 2016, 113(24): 6785-6790. DOI:10.1073/pnas.1600899113 |
| [22] |
SONG H, ZHAO S Z, HOU L, et al. Bioinformatics analysis on LOX genes related to disease resistance in whole genome of Arachis duranensis and Arachis ipaënsis[J].
Shandong Agric Sci, 2015, 47(10): 1-7. 宋辉, 赵术珍, 侯蕾, 等. 野生花生全基因组抗病相关LOX基因的生物信息学分析[J]. 山东农业科学, 2015, 47(10): 1-7. DOI:10.14083/j.issn.1001-4942.2015.10.001 |
| [23] | DASH S, CANNON E K S, KALBERER S R, et al. Peanut base and other bioinformatic resources for peanut[M]//STALKER H T, WILSON R F. Peanuts Genetics, Processing, and Utilization. Boulder, Urbana:AOCS Press, 2016:241-252. doi:10.1016/B978-1-63067-038-2.00008-3. |
| [24] |
GUO A Y, ZHU Q H, CHEN X, et al. GSDS:A gene structure display server[J].
Hereditas (Beijing), 2007, 29(8): 1023-1026. 郭安源, 朱其慧, 陈新, 等. GSDS:基因结构显示系统[J]. 遗传, 2007, 29(8): 1023-1026. DOI:10.16288/j.yczz.2007.08.004 |
| [25] |
WANG Z H, TONG C B, YUAN W Z, et al. Bacterial-type PEPC genes identification and expression in developing seeds from four oil crops[J].
Chin J Oil Crop Sci, 2013, 35(1): 8-16, 23. 王志慧, 童超波, 袁午舟, 等. 四种油料作物中的细菌型PEPC基因的鉴定及在发育种子中的表达[J]. 中国油料作物学报, 2013, 35(1): 8-16, 23. DOI:10.7505/j.issn.1007-9084.2013.01.002 |
| [26] | XU W X, AHMED S, MORIYAMA H, et al. The importance of the strictly conserved, C-terminal glycine residue in phosphoenolpyruvate carboxylase for overall catalysis:Mutagenesis and truncation of GLY-961 in the sorghum C4 leaf isoform[J]. J Biol Chem, 2006, 218(25): 17238-17245. DOI:10.1074/jbc.M602299200 |
| [27] |
PAN L J, LIU F Z, WAN Y S, et al. Expression comparison of the phosphoenolpyruvate carboxylase gene in high oil-content peanut varieties Jihua 4 and its parent[J].
J Peanut Sci, 2015, 44(4): 37-41. 潘丽娟, 刘风珍, 万勇善, 等. PEPC基因在高油品种吉花4号及其亲本中的表达比较[J]. 花生学报, 2015, 44(4): 37-41. DOI:10.14001/j.issn.1002-4093.2015.04.007 |
| [28] | MATSUMURA H, IZUI K, MIZUGUCHI K. A novel mechanism of allosteric regulation of archaeal phosphoenolpyruvate carboxylase:A combined approach to structure-based alignment and model assessment[J]. Protein Eng Des Sel, 2006, 19(9): 409-419. DOI:10.1093/protein/gzl025 |
| [29] | IGAWA T, FUJIWARA M, TANAKA I, et al. Characterization of bacterial-type phosphoenolpyruvate carboxylase expressed in male gametophyte of higher plants[J]. BMC Plant Biol, 2010, 10: 200 DOI:10.1186/1471-2229-10-200 |
 2018, Vol. 26
2018, Vol. 26



