桉树自然分布于澳大利亚及华莱士线以东的澳洲大陆附近的太平洋岛屿。桉树生长快速,抗逆性较强,用途非常广泛,是热带和亚热带地区的重要造林树种。我国引种桉树已有127年的历史,现在我国华南、西南和华东南地区进行了广泛的引种和栽培。据2016年底最新数据统计,我国桉树人工林面积已达4.50×106 hm2[1]。
桉树遗传资源丰富,1998年底确认的桉树有945个分类群[2-3]。我国早期的桉树引种具有很大的随机性,最早的种子可能来源已经发生变异的地理种群,或非正常的变异个体,在此基础上发展起来的新栽培群体,其特征与自然分布区内的正常种群很可能存在较大的不同[4]。而且,桉树同一亚属内的种间容易产生自然杂交种,Griffin等[5]列出了494个自然发生的种间杂交组合,其中141个涉及20种主要人工桉树林栽培种。桉树各属和亚属的种群分离趋异的间断分布特点,使得属内一些亲缘关系较远或没有亲缘关系的种在表型上表现出相似性, 这些都增加鉴定引种地的不同桉树种的困难。因此,建立一套桉树种间种质资源的精准鉴定体系势在必行。
植物种质资源是植物利用和改良的物质基础, 更是实施各个育种途径的原材料。20世纪80年代以来,我国引种桉树的种类迅速增加。由于桉树在外观上具有一定的相似性,不同树种常笼统地称为“桉树”;而且有关桉树分类的文献和资料比较有限,以致于经常造成鉴定错误,且错误结果广泛传播, 影响甚大[6]。因此,对我国引种的桉树进行种间种质资源的精准鉴定体系的研究迫在眉睫。
SSR (simple sequence repeat)分子标记,又称为微卫星标记(microsatellite),由于其多态性高,重复性和稳定性好等优点,已广泛应用于各物种的DNA指纹图谱绘制、亲缘关系分析、品种鉴定、遗传多样性分析等研究[7-10]。本研究基于SSR标记技术和DNA测序技术,对28种桉树115份种质材料进行PCR扩增产物直接测序,根据4个在同种不同个体DNA扩增序列完全一致的SSR位点的微卫星信息特征和两端侧翼序列的核苷酸特征对28种桉树构建特异鉴定条码,以期达到精准鉴定的目的,为桉树品种(无性系)种质鉴定体系的构建奠定基础,为桉树遗传育种的研究和生产实践提供依据。
1 材料和方法 1.1 材料供试材料为28种桉树115份种质材料(表 1), 分别取自云南省昆明市海口林场、广西壮族自治区柳州市黄冕林场、广东省湛江市南方国家级林木种苗示范基地和云南省楚雄州一平浪林场。每种桉树采集若干个体的新鲜幼嫩叶片,保存于-80℃低温冰箱。参照桉树分类系统[11-14],28种桉树均属于桉属(Eucalyptus),其中24种为双蒴盖亚属(subgenus Symphyomyrtus),4种为单蒴盖亚属(subgenus Monocalyptus)。
| 表 1 28种桉树基本信息 Table 1 Information of 28 Eucalypts species |
桉树叶片总DNA的提取采用CTAB法[15],并参照Gan等[16]和李发根[17]的方法进行适当改良。提取结果利用Throme核酸蛋白测定仪和1.5%琼脂糖凝胶电泳进行浓度和质量测定,并将提取样品稀释至约100 ng μL-1,保存于-20℃冰箱备用。
1.3 SSR-PCR扩增反应本研究从110对SSR引物中筛选出种内PCR扩增片段大小基本一致的引物20对,利用特异SSR引物进行SSR-PCR扩增,其产物直接测序,最终选出4对种内扩增序列完全保守的特异SSR引物(表 2), 对28种桉树115份种质材料进行分析。
| 表 2 SSR引物序列 Table 2 Sequences of SSR primers |
SSR-PCR扩增反应体系总体积为50 μL,其中模板DNA约100 ng,100 μmol L-1引物各5.0 μL, 2×TopTaq Master Mix (1.25 U Taq DNA聚合酶, 0.2 mmol L-1 dNTPs,1.5 mmol L-1 MgCl2) 25 μL。采用Touch-Down PCR扩增反应程序:首先94℃预变性4 min,94℃变性30 s,50℃~60℃复性30 s, 72℃延伸1 min,共35个循环;最后72℃延伸10 min。PCR扩增产物经1.5%琼脂糖凝胶电泳检测后, 送至英潍捷基(上海)贸易有限公司(广州实验室)进行序列测定。
1.4 数据统计和分析使用软件Genious 7.1对所得序列进行核对,并进行相应的正反序列拼接和手工调整,以保证序列的准确性。软件SSRHunter对4个SSR位点的所有扩增序列进行SSR位点的检索,分析桉树种间和同种桉树不同个体间的微卫星重复单元和重复次数的差异性;通过软件BioEdit 7.09对4个位点在不同种间的扩增序列进行Clustal W比对分析,统计各位点在不同种间的扩增序列中碱基变化情况,找出侧翼保守序列中的特异性碱基,即扩增序列中某处碱基在多数种间均存在变异(替换、插入或缺失)。通过综合4个SSR位点的微卫星重复次数和特异碱基组合建立28种桉树种间种质资源的鉴定条码。最后,通过软件Modeltest 2.1.5选择最佳建树模型[18]的基础上,分别利用软件PhyML-3.1和WinPaup-4.10以最大似然法(maximum likelihood, ML)与最大简约法(maximum parsimony, MP)重建系统发生树,分析桉树种间亲缘关系的远近。
2 结果和分析 2.1 SSR-PCR扩增结果根据4对SSR引物对28种桉树115份种质材料的扩增结果,桉树种内不同个体间的PCR扩增序列完全一致,表明4个SSR位点在桉树种内没有长度多态性。从图 1可见,4个SSR位点在不同桉树种的扩增片段虽然存在较小的差异性(引物eSSR-GR029扩增的白腊树桉DNA, 泳道6-1),但各桉树样品的扩增片段大小基本一致,特别是同一桉树种不同个体间的扩增片段大小几乎一致,且均为单态位点,说明4个SSR位点的多态性丰富程度较低。
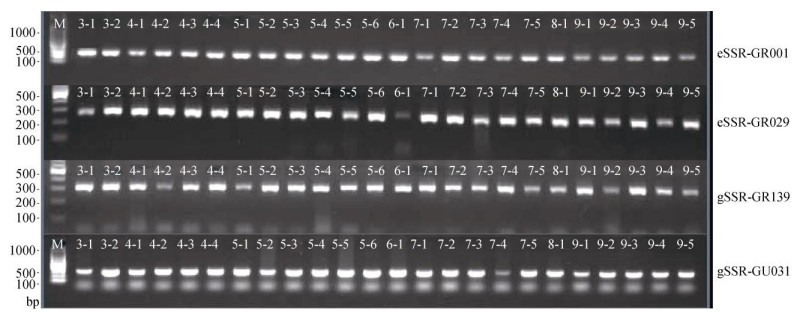
|
图 1 4对SSR引物的部分种质的扩增结果。M: Marker; 3-1, 3-2:奥米奥圆叶桉; 4-1~4-4:巴吉桉; 5-1~5-6:小叶桉; 6-1:白腊树桉; 7-1~7-5:毛皮桉; 8-1:三花桉; 9-1~9-5:山桉。 Fig. 1 Amplification of part germplasms by four pairs of SSR primer. M: Marker; 3-1, 3-2: Eucalyptus neglecta; 4-1-4-4: E. badjensis; 5-1-5-6: E. parula; 6-1: E. fraxinoides; 7-1-7-5: E. macarthurii; 8-1: E. triflora; 9-1-9-5: E. dalrympleana. |
位点eSSR-GR0001、eSSR-GR029、gSSR-GR139和gSSR-GU031的扩增序列中,对种内各种质材料的扩增序列完全一致,即4个SSR位点在28种桉树中的扩增序列完全保守(侧翼序列和微卫星序列完全保守),种内不同个体间在4个SSR位点处的扩增序列完全无差异。
对4个SSR位点的扩增序列进行SSR位点检索表明,当桉树种内个体间的序列无差异时,桉树种间的微卫星序列的差异也不明显。在位点eSSR-GR001和位点eSSR-GR029的扩增序列中(表 3), 不同桉树种的微卫星重复单元和重复次数完全一致。位点gSSR-GR139的115条扩增序列中,单蒴盖亚属的HK06、HK08、YPL26、YPL28的微卫星重复单元为CTC,重复次数7~9。双蒴盖亚属的25种桉树微卫星重复单元均为CTT,重复次数为4~10。位点gSSR-UR009的115条扩增序列中,微卫星重复单元均为GCC,而单蒴盖亚属的微卫星重复次数均为3,双蒴盖亚属的24种桉树的微卫星重复次数为5~8。因此,微卫星重复单元和重复次数的变化,与桉树种间亲缘关系的远近存在着相关性。
| 表 3 4对SSR引物对28种桉树SSR位点的扩增 Table 3 Amplification sequences of 28 Eucalyptus species by 4 SSRs |
4个位点在种间的微卫星序列变化主要是由于微卫星重复次数的变化引起。有研究表明,SSR重复次数在很大程度上与序列长度多态性(simple sequence length polymorphism, SSLP)相关[19]。简单序列长度多态性在不同动植物品系中的重复次数不同,可以用来快速检测出等位基因间的差异。
根据4个SSR位点微卫星序列的重复次数变化情况,微卫星序列的重复单元的碱基组成在28种桉树间的差异较小。若组合4个位点的重复次数进行条码编辑,不仅简单明了,而且根据数字的组合,能够区分其中部分桉树种类。从表 3可以看到,28种桉树根据4个SSR位点微卫星重复次数组成的鉴定条码有9种数字条码,其中HK04巴吉桉,HK06白蜡树桉,HK11樟脑桉,HK18头果桉,SX22细叶桉和YPL26滨河白桉的数字条码特异,即通过4个SSR位点微卫星重复次数组成的数字条码能够将此6种桉树进行区分。HK08三花桉和YPL28斜叶桉的4个SSR位点中微卫星重复单元和重复次数完全一致,均属于桉属单蒴盖亚属;HM19柳桉, SX23尾叶桉,SX25巨桉和YPL27大叶桉的4个SSR位点中微卫星重复单元和重复次数完全一致, 均属于桉属双蒴盖亚属横脉组。
因此,基于4个SSR位点微卫星序列的长度差异性,能够将巴吉桉、白蜡树桉、樟脑桉、头果桉、细叶桉和滨河白桉区分开来,但仍有22种桉树不能区分。
2.3 侧翼保守序列的特征分析SSR位点侧翼序列一般是相对保守的单拷贝序列,本研究对4个SSR位点的侧翼序列进行分析表明,位点eSSR-GR001、eSSR-GR029、gSSR-GR139和gSSR-GU031对桉树种内的扩增序列完全一致, 而不同种桉树在同一SSR位点的扩增序列存在着一定的差异。
利用DNAMAN软件对4个SSR位点扩增序列进行比对分析,桉树种间的扩增序列相似度Identity较高。4个位点扩增序列的相似度值由高到低依次为位点eSSR-GR001 (99.34%) > 位点eSSR-GR029 (99.24%) > 位点gSSR-GU031 (95.58%) > 位点gSSR-GR139 (95.05%)(图 2)。

|
图 2 28种桉树基于位点gSSR-GR139的扩增序列比对 Fig. 2 Alignment of 28 Eucalyptus species based on gSSR-GR139 loci |
在28种桉树中各位点扩增序列的比对分析表明,侧翼保守序列中存在较多的单核苷酸的变异(替换、缺失、插入),多个连续核苷酸变异的较少。4个SSR位点共有76处单核苷酸(single nucleotidetype)变异,占总变异的79.2%,二核苷酸(di-nucleotide-type)变异共6处,三核苷酸(tri-nucleotidetype)及三核苷酸以上的变异共14处。
每个SSR位点的侧翼序列中选择1~2处特异性核苷酸组合成碱基条码。通过序列比对进行特异性核苷酸的定位,4个SSR位点的96处碱基变异中找出4处特异核苷酸,分别位于位点eSSR-GR001扩增序列的92~94 bp处、eSSR-GR029扩增序列的249 bp处、gSSR-GR139扩增序列的255~256 bp处、gSSR-GU031扩增序列的183 bp处(图 2)。将4处特异核苷酸进行组合,4个SSR位点在碱基条码中的顺序依次为位点eSSR-GR001、eSSR-GR029、gSSR-GR139和gSSR-GU031,构建侧翼序列特异碱基字母条码(表 4)。
| 表 4 4个SSR位点侧翼序列特异碱基条码表 Table 4 Barcodes of special combination of bases on 4 SSRs' flanking sequences |
从表 4可见,侧翼序列中特异核苷酸组成的条码共10个,能够将HK08三花桉,HK11樟脑桉, SX20窿缘桉精准鉴定出来。HK01多枝桉、HK07毛皮桉、HK09山桉、HK14灰桉、HK15史密斯桉和HK17多利桉在4处特异碱基处的碱基完全相同,同属于桉属双蒴盖亚属蓝桉组;同样,YPL26滨河白桉和YPL28斜叶桉在4处特异碱基处的碱基完全相同,均属于桉属单蒴盖亚属。
2.4 桉树种间种质资源鉴定分析结合表 3的数字条码和表 4的碱基条码,基于4个SSR微卫星序列能够从28种桉树中精准鉴定的种有9种,共计得到16种鉴定条码,部分桉树种间共享同一种鉴定条码(表 5)。构建4个SSR位点的微卫星序列和侧翼保守序列中的特异核苷酸组合的鉴定条码,能够进行精准鉴定的种有:HK04巴吉桉、HK05小叶桉、HK06白蜡树桉、HK08三花桉、HK11樟脑桉、HK13新英格兰桉、HK18头果桉、SX20窿缘桉和SX22细叶桉。
| 表 5 基于SSR标记的28种桉树种质鉴定条码表 Table 5 Identification barcodes of 28 Eucalyptus species based on SSR marker |
共享同一鉴定条码的不同桉树种中,HK01多枝桉、HK07毛皮桉、HK09山桉、HK14灰桉、HK15史密斯桉和HK17多利桉在分类中同属于桉属双蒴盖亚属蓝桉组;HK12假美叶桉和HK16本沁桉属于桉属双蒴盖亚属蓝桉组;HM19柳桉和SX25巨桉属于桉属双蒴盖亚属横脉组;SX23尾叶桉和YPL27大叶桉属于桉属双蒴盖亚属横脉组;YPL26滨河白桉和YPL28斜叶桉属于桉属单蒴盖亚属; HK02迪恩桉、HK03奥米奥圆叶桉和SX24赤桉属于桉属双蒴盖亚属;HK10邓恩桉和SX21维塔桉属于桉属双蒴盖亚属。
2.5 桉树种间亲缘关系分析综合分析4个SSR位点在28种桉树中的扩增序列,利用最大简约法和最大似然法构建系统发育树(图 3)。根据软件DnaSP 5.10.01对4个SSR位点的综合序列进行多态性评估,核苷酸多态性Pi= 0.012,单倍型多样度Hd=0.989,变异位点比率为0.082。最大似然法构建系统发育树的最佳建树模型为TIM1+I。最大简约法分析得到一致性指数CI= 0.679,维持性指数RI=0.804。
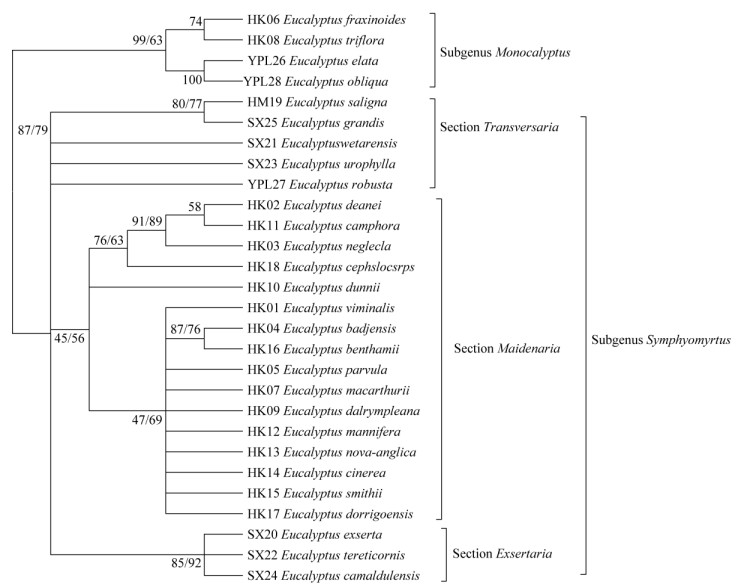
|
图 3 基于4个SSR位点的综合MP法系统发育树。枝点处数字表示ML/MP法1 000次启发式搜索值。 Fig. 3 MP phylogenetic based on four SSRs sites. The numbers on branch points represent heuristic searching of 1 000 times by ML/MP methods. |
依据前人的分类系统[11-14],28种桉树均属于桉属,包括4种单蒴盖亚属和24种双蒴盖亚属。由图 3可见,28种桉树基于4个SSR位点中扩增序列的聚类状态与传统分类学完全一致,可分为两大类群,属于单蒴盖亚属的4种桉树聚为一大类群, 属于双蒴盖亚属的24种桉树聚为另一大类群。24种双蒴盖亚属桉树的分组情况与传统分类学基本一致,可分为横脉组、蓝桉组和窿缘组3个亚群。但蓝桉组亚群中HK02迪恩桉在传统分类学中属于横脉组,与HM19和YPL27等是近缘种关系[20], 而图 3表明HK02迪恩桉与HK11樟脑桉的遗传距离更近,HK11樟脑桉属于桉属双蒴盖亚属蓝桉组。在形态特征上,横脉组的成龄叶上下两面同色,脉序或多或少横向排列;而蓝桉组的实生苗幼态叶无柄[21]。HK02迪恩桉的形态特征与横脉组和蓝桉组的均不一致:幼态叶具柄,对生,叶卵形或椭圆形, 幼茎呈红色,成熟叶片两面不同色,脉序排列没有明显的横向排列,树枝、树干及树皮均呈红色[22-23]。因此,HK02迪恩桉的分类地位还有待深入研究。
3 讨论和结论SSR标记中微卫星DNA序列的主要特征是重复单元的重复次数不同和重复程度的不完全。微卫星DNA序列在生物进化过程中存在着大量的变异,位于非编码区的突变多为“中性突变”,对生物生长和发育都不会产生太大的影响,一般不受选择的影响,大部分变异都能够得到保存。而基因组SSR的丰富度和对应的各种功能与效应都与他们的突变率相关,与表达序列标签SSR上的点突变率相比, 要高很多[24]。
本研究中4个SSR位点具有在种内个体间的扩增序列完全保守的特征。4个SSR位点在同种桉树不同个体间的SSR-PCR扩增序列完全一致,排除了因个体间SSR位点中重复单元和重复次数的变异而导致的种间差异,确保了4个SSR位点在同种桉树内微卫星序列和侧翼序列的完全保守。
分析4个SSR位点的遗传多样性表明,微卫星DNA序列中位点gSSR-GR139和gSSR-GU031的遗传多样性明显高于位点eSSR-GR001和eSSR-GR029,即来源于基因组SSR位点比EST-SSR位点具有更高的变异性,这与刘果等[25]报道桉树EST-SSR引物的多态性要低于Genomic-SSR引物的结论一致。
在位点gSSR-GR139的扩增序列中,形态分类中属于单蒴盖亚属的4种桉树的微卫星重复单元一致,属于双蒴盖亚属的24种桉树的微卫星重复单元分布一致;在位点gSSR-UR009的扩增序列中, 形态分类中属于单蒴盖亚属的4种桉树的微卫星重复次数完全相同。这与Stephanie等[19]认为不同种间(种群间)微卫星重复次数(重复单元的长度)不一致与种间亲缘关系存在一定关系的结论相一致。
本研究对4个SSR位点进行微卫星序列及两端侧翼保守序列进行分析,表明微卫星的变异主要是由于重复序列和侧翼序列的改变引起的。这与Matsuoka等[26]和Sonnante等[27]分别报道玉米(Zea mays)和朝鲜蓟菜(Cynara cardunculus var. scolymus)的SSR位点变异主要来源于侧翼序列的插入/缺失和点突变的结论相同。
对4个SSR位点扩增序列中的侧翼序列进行比对分析,桉树种间的侧翼序列中单碱基的变异比较普遍,占总变异的79.2%。这与王静毅[24]报道4个位点的侧翼序列均存在插入/缺失突变,等位基因的长度变异主要是由SSR重复次数及侧翼序列的插入/缺失和点突变造成的结论一致。
Stebbins[28]曾指出桉树能够在相对均匀的种群间产生杂种,双蒴盖亚属是桉属内最大的亚属,为方便划分为数众多的组系,亚属内组的划分在某种程度上存在主观性,某些组内的树种之间完全是可交可孕的[6]。根据Griffin[5]对蜡叶标本的检查和统计,横脉组、蓝桉组以及窿缘组的树种彼此之间产生自然杂种的频率很高。本研究中构建桉树树种精准鉴定条码出现共享同一鉴定条码的桉树组合,也证实横脉组、蓝桉组和窿缘组树种间存在较近的遗传关系,各组间部分树种的遗传关系比较亲密。同一亚属共享同一鉴定条码的桉树种间有可能进行自然或人工杂交,产生杂交种。这为桉树杂交育种工作提供了新思路和生物学依据。从表 5可知, HK02迪恩桉和HK03奥米奥圆叶桉、SX24赤桉的鉴定条码完全一致,而迪恩桉在形态分类中属于横脉组,奥米奥圆叶桉属于蓝桉组,赤桉属于窿缘组种,由此证实了蓝桉组、横脉组和窿缘组具有比较近的遗传关系,也为3个组间树种出现频繁杂交提供了证据。
利用4个SSR位点的扩增序列构建系统发育树,结果表明,28种桉树基于4个SSR位点扩增序列的聚类结果与传统分类系统[11-14]的分类地位基本一致。但属于横脉组的HK02迪恩桉明显表现出与蓝桉组的亲缘关系更近,且迪恩桉的形态特征与横脉组和蓝桉组的均不一致,由此可以推断,在形态分类中将双蒴盖亚属内的迪恩桉划分为横脉组具有一定的主观性。
本研究通过构建基于4个SSR位点的微卫星重复次数和侧翼序列特异碱基组合的鉴定条码,对28种桉树进行了种质资源的精准鉴定。15个鉴定条码,能够精确鉴定的桉树有9种(32.1%),即9种桉树有特异性的鉴定条码。共享同一鉴定条码的桉树种间存在较近的亲缘关系,这为桉树杂交育种工作提供了新的选种方法,也为全面系统地构建桉树种间种质资源精准鉴定体系得出了初步的研究成果, 为桉树品种(无性系)种质资源的精准鉴定体系的建立奠定了基础。
| [1] |
XIE Y J.
Eucalyptus[M]. Beijing: China Forestry Publishing House, 2015: 35-37.
谢耀坚. 真实的桉树[M]. 北京: 中国林业出版社, 2015: 35-37. |
| [2] | WILCOX M D. A Catalogue of the Eucalypts[M]. Auckland: Groome Poyry Ltd, 1997: 114-116. |
| [3] | WILCOX M D. Supplement to a Catalogue of the Eucalypts[M]. Auckland: Groome Poyry Ltd, 1998: 9-11. |
| [4] |
QI S X. Eucalypt introduction and development status in China[J].
Guangxi For Sci, 2006, 35(4): 250-252. 祁述雄. 中国引种桉树与发展现状[J]. 广西林业科学, 2006, 35(4): 250-252. DOI:10.3969/j.issn.1006-1126.2006.04.016 |
| [5] | GRIFFIN A R, BURGESS I P, WOLF L. Patterns of natural and manipulated hybridisation in the genus Eucalyptus L'hérit:A review[J]. Aust J Bot, 1988, 36(1): 41-66. DOI:10.1071/BT9880041 |
| [6] |
QI S X.
Eucalyptus in China[M]. Beijing: China Forestry Publishing House, 2002: 33-55.
祁述雄. 中国桉树[M]. 北京: 中国林业出版社, 2002: 33-55. |
| [7] |
HONG Y B, LIANG X Q, CHEN X P, et al. Construction of genetic linkage map in peanut (Arachis hypogaea L.) cultivars[J].
Acta Agron Sin, 2009, 35(3): 395-402. 洪彦彬, 梁炫强, 陈小平, 等. 花生栽培种SSR遗传图谱的构建[J]. 作物学报, 2009, 35(3): 395-402. DOI:10.3724/SP.J.1006.2009.00395 |
| [8] |
LIANG Y Q, ZHANG J J, LIANG J J, et al. Genetic diversity among persimmon germplasms in Henan Province[J].
Sci Silv Sin, 2015, 51(6): 71-80. 梁玉琴, 张嘉嘉, 梁晋军, 等. 河南省柿种质资源的遗传多样性[J]. 林业科学, 2015, 51(6): 71-80. DOI:10.11707/j.1001-7488.20150609 |
| [9] |
GUO Z D, XIA X Y, AN L J, et al. Genetic relationship analysis and cultivar identification of blueberry based on SSR markers[J].
J Plant Genet Resour, 2015, 16(5): 1020-1026. 郭照东, 夏秀英, 安利佳, 等. 基于SSR标记的越橘亲缘关系分析及品种鉴定[J]. 植物遗传资源学报, 2015, 16(5): 1020-1026. DOI:10.13430/j.cnki.jpgr.2015.05.013 |
| [10] |
ZHANG C L, GUO Z H, ZHANG X Q, et al. An analysis of species identification and genetic divergence of drooping wildrye (Elymus nutans) and siberian wildrye (E. sibiricus) by SSR markers[J].
J Plant Genet Resour, 2016, 17(3): 416-422. 张成林, 郭志慧, 张新全, 等. 利用SSR标记对垂穗披碱草和老芒麦进行物种鉴定和遗传变异分析[J]. 植物遗传资源学报, 2016, 17(3): 416-422. DOI:10.13430/j.cnki.jpgr.2016.03.003 |
| [11] | PRYOR L D, JOHNSON L A S. A Classification of the Eucalypts[M]. Canberra: The Australian National University, 1971: 102. |
| [12] | HILL K D, JOHNSON L A S. Systematic studies in the Eucalypts:7. A revision of the bloodwoods, genus Corymbia (Myrtaceae)[J]. Telopea, 1995, 6(2/3): 185-504. DOI:10.7751/telopea19953017 |
| [13] | BROOKER M I H. A new classification of the genus Eucalyptus L'Hér. (Myrtaceae)[J]. Aust Syst Bot, 2000, 13(1): 79-148. DOI:10.1071/SB98008 |
| [14] | NICOLLE D. Classification of the Eucalypts (Angophora, Corymbia and Eucalyptus), Version 2[EB/OL]. (2015-04). http://www.dn.com.au/Classification-Of-The-Eucalypts.pdf. |
| [15] | DOYLE J J, DOYLE J L. Isolation of plant DNA from fresh tissue[J]. Focus, 1990, 12(1): 13-15. |
| [16] | GAN S M, SHI J S, LI M, et al. Moderate-density molecular maps of Eucalyptus urophylla S. T. Blake and E. tereticornis Smith genomes based on RAPD markers[J]. Genetica, 2003, 118(1): 59-67. DOI:10.1023/A:1022966018079 |
| [17] |
LI F G. Linkage map construction and growth QTL detection in Eucalyptus urophylla and E. tereticornis based on STS markers[D]. Beijing:Chinese Academy of Forestry, 2010:50-62.
李发根. 尾叶桉和细叶桉STS标记连锁图谱构建及生长性状QTL定位研究[D]. 北京: 中国林业科学研究院, 2010: 50-62. |
| [18] | POSADA D, CRANDALL K A. MODELTEST:Testing the model of DNA substitution[J]. Bioinformatics, 1998, 14(9): 817-818. DOI:10.1093/bioinformatics/14.9.817 |
| [19] | BARTHE S, GUGERLI F, BARKLEY N A, et al. Always look on both sides:Phylogenetic information conveyed by simple sequence repeat allele sequences[J]. PLoS One, 2012, 7(7): e40699 DOI:10.1371/journal.pone.0040699 |
| [20] |
HU T Y, LI X Q. Research on introduction of Encalyptus grandis and its normal region[J].
J Sichuan For Sci Technol, 1999, 20(4): 8-13. 胡天宇, 李晓清. 巨桉引种栽培及适生区域的研究[J]. 四川林业科技, 1999, 20(4): 8-13. DOI:10.16779/j.cnki.1003-5508.1999.04.002 |
| [21] |
WANG H R.
A Chinese Appreciation of Eucalypts[M]. Beijing: Science Press, 2010: 1-16, 93.
王豁然. 桉树生物学概论[M]. 北京: 科学出版社, 2010: 1-16, 93. |
| [22] |
DONG J X, WU C W. Towards horticultural and ornamental value of 27 introduced Eucalyptus species[J].
Yunnan For Sci Technol, 2003(4): 39-41. 董静曦, 吴赤卫. 27种引种桉树的园艺观赏价值[J]. 云南林业科技, 2003(4): 39-41. DOI:10.16473/j.cnki.xblykx1972.2003.04.007 |
| [23] |
HAN Z, LIN J Y, HUANG Y F. The introduced resource and application on gardening virescence of Eucalypt in China[J].
Guang-dong For Sci Technol, 2007, 23(1): 109-111. 韩宙, 林家怡, 黄永芳. 我国桉树引种资源及其在园林绿化上的应用[J]. 广东林业科技, 2007, 23(1): 109-111. |
| [24] |
WANG J Y. Development and characterization of microsatellite markers for phylogenetic analysis of Musa[D]. Haikou:Hainan University, 2010:43-71.
王静毅. SSR分子标记开发和在香蕉种质资源遗传分析中的应用[D]. 海口: 海南大学, 2010: 43-71. |
| [25] |
LIU G, ZHANG D Q, XIE Y J, et al. Rapid screening and trans-ferability analysis of genomic-SSR and EST-SSR primers in Eucalypt[J].
Sci Silv Sin, 2013, 49(2): 127-133. 刘果, 张党权, 谢耀坚, 等. 桉树genomic-SSR和EST-SSR引物的快速筛选与通用性研究[J]. 林业科学, 2013, 49(2): 127-133. DOI:10.11707/j.1001-7488.20130219 |
| [26] | MATSUOKA Y, MITCHELL S E, KRESOVICH S, et al. Micro-satellites in Zea:Variability, patterns of mutations, and use for evolu-tionary studies[J]. Theor Appl Genet, 2002, 104(2/3): 436-450. DOI:10.1007/s001220100694 |
| [27] | SONNANTE G, CARLUCCIO A V, de PAOLIS A, et al. Identification of artichoke SSR markers:Molecular variation and patterns of diversity in genetically cohesive taxa and wild allies[J]. Genet Resour Crop Evol, 2008, 55(7): 1029-1046. DOI:10.1007/s10722-008-9310-5 |
| [28] | STEBBINS Jr G L. Variation and Evolution in Plants[M]. New York: Columbia University Press, 1950: 174-184. |
 2018, Vol. 26
2018, Vol. 26

