茶树(Camellia sinensis)遗传多样性研究已从早期的形态学标记进入分子标记时代。随着生物技术的发展,分子标记技术经历了RFLP、RAPD、AFLP、ISSR、SSR和SNP等发展阶段,其中SSR分子标记技术,因其具有重复性好、多态性高、呈共显性、易于检测和鉴别能力高等优点,而被广泛应用。分子标记技术在茶树种质资源研究中的应用快速发展[1-8], 利用SSR分子标记技术对茶树种质资源遗传多样性的研究报道较多[9-15],促进了茶叶生产选种和茶树遗传改良工作。SSR分子标记已成为指纹图谱构建及亲子鉴定的重要手段。利用SSR分子标记构建茶树指纹图谱的报道也不断增多[16-21]。同时SSR标记是茶树遗传连锁图构建及QTL定位的重要工具[1-2, 22]。
福建是我国的产茶大省,产制乌龙茶、红茶、白茶、绿茶四大茶类和再加工类的茉莉花茶,除绿茶外,均由福建首创。福建省茶园面积居全国第五位,但毛茶产量、产值均位居全国第一。福建省茶产业发达的物质基础是拥有丰富的茶树种质资源, 茶树品种位居全国前列,但当前对福建省茶树品种的遗传多样性研究报道极少[5, 18, 23]。至2016年底, 福建省选育的国家级、省级茶树品种共45个(绿茶品种16个,乌龙茶品种29个),其中国家审(认、鉴)定品种26个;福建省审(认)定品种19个。本研究通过SSR分子标记技术,构建43个(缺2个品种:大叶乌龙、春闺)福建省选育的国家级与省级审(认、鉴)定品种的指纹图谱,以及分析其遗传多样性和亲缘关系,较系统地了解福建省茶树品种的遗传背景,从而为茶树品种遗传改良与亲本选配及栽培选种提供参考依据。
1 材料和方法 1.1 材料材料为43个福建省选育的国家级、省级品种(表 1),所有参试品种于2014年11月采自福建省农业科学院茶叶研究所2号山种质资源圃,每个品种采摘1片叶子提取DNA。
| 表 1 参试品种 Table 1 Cultivars tested |
本试验所用38对SSR引物均来自Ma等和Tan等[1-2]。茶树基因组DNA提取使用天根生化科技(北京)有限公司的植物基因组DNA提取试剂盒(TIANGEN,DP305)。PCR反应总体积为20 μL,其成分为:模板DNA 1 μL,10×PCR buffer 2 μL,2 mmol L-1 dNTPs 1.5 μL,25 mmol L-1 MgCl2 1.2 μL, 10 μmol L-1SSR正反向引物各0.3 μL, 1U Taq DNA聚合酶,用ddH2O补齐20 μL。PCR扩增程序为:94℃预变性5 min,94℃变性45 s,57℃退火45 s,72℃延伸50 s,共35个循环,最后72℃延伸7 min。PCR产物用10%聚丙烯酰胺凝胶(PAGE)电泳检测[1-3]。用于构建指纹图谱的引物序列见表 2。
| 表 2 构建指纹图谱所用SSR引物序列 Table 2 Primer sequences of SSR used to construct fingerprints |
根据PAGE胶电泳图统计条带,对于同一引物的扩增产物,迁移位置相同的条带记为1个位点,即扩增阳性(有较为清晰条带出现)赋值为“1”,扩增阴性(无条带或者弱带)赋值为“0”,所得数据输入Excel建立原始数据矩阵。其中任一位点若有1个或1个以上个体不同于其他个体即为一个多态位点。以上所得数据按SHAN邻接法对供试种质资源进行UPGMA遗传相似性聚类, 并绘制树状聚类图。数据统计分析在NTSYS软件中进行。
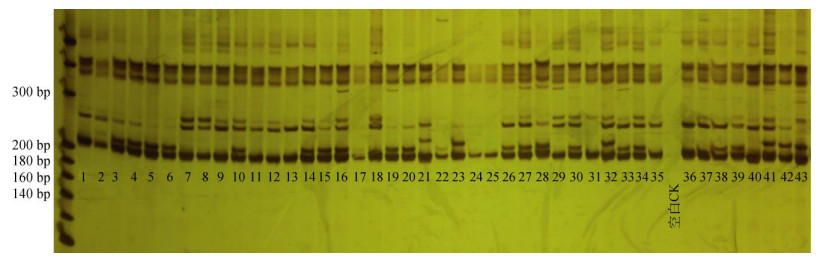
2 结果和分析 2.1 SSR指纹图谱构建本试验共采用38对SSR引物对43个福建省选育茶树品种进行PCR扩增,剔除扩增效果差的引物后,选取23对扩增条带清晰,多态性较好的引物进行数据统计和分析。图 1为SSR引物A134对43个参试品种PCR扩增电泳图。为了准确而高效地区分43个福建省选育的茶树品种,最终选取扩增条带少、多态性较高的7对SSR引物,用于构建43个福建省选育茶树品种指纹图谱(表 3)。
|
图 1 SSR引物A134对43个茶树品种的PCR扩增 Fig. 1 PCR amplification of 43 cultivars by A134 primer |
| 表 3 43个福建省选育茶树品种的SSR指纹图谱 Table 3 SSR fingerprints of 43 tea cultivars in Fujian Province |
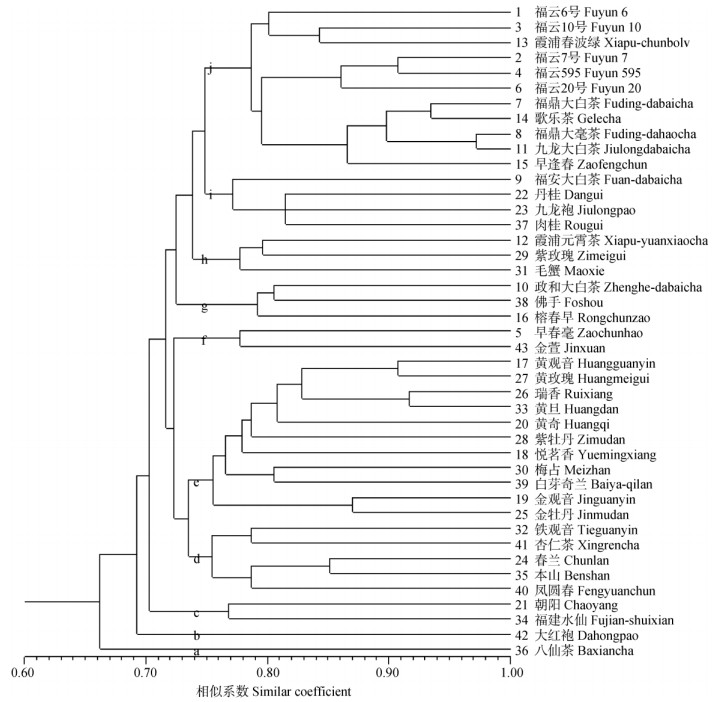
基于福建省43个茶树品种的SSR标记的PCR扩增结果进行遗传聚类分析(图 2)。结果表明,在相似系数为0.75处,43个品种可以划分为10个类群(a~j),其中a和b类群都只有1个品种,分别为‘八仙茶’和‘大红袍’,遗传距离最远的是‘八仙茶’,其次是‘大红袍’。c类群含有2个乌龙茶品种,为‘福建水仙’和‘朝阳’。其余39个茶树品种分别聚为d、e、f、g、h、i、j共7个类群。从图 2中可见16个绿茶品种有11个聚在j类群,其中‘福鼎大毫茶’与‘九龙大白茶’是遗传距离最近的两个品种。27个乌龙茶品种中有16个聚在d、e两个类群,其中d类群5个品种,e类群11个品种。而介于e类群(乌龙茶)与j类群(绿茶)之间的4个小类群(f、g、h、i类群共有12个品种)既有绿茶品种也有乌龙茶品种。总体而言,SSR标记能够把绿茶品种和乌龙茶品种进行模糊区分。
|
图 2 43个福建省选育茶树品种的SSR分子标记遗传聚类图 Fig. 2 Genetic clustering diagram of 43 tea cultivars in Fujian Province based on SSR molecular markers |
从图 2可以直观地看出43个品种的遗传多样性,进一步分析表明,品种间的遗传距离及遗传多样性与品种的发源地、品种的亲本来源、品种形态特征等有很强的相关性。
遗传距离最远的4个品种为‘八仙茶’、‘大红袍’、‘福建水仙’及‘朝阳’,这与他们的发源地及特殊的生物学特征有关,‘八仙茶’是从福建省诏安县秀篆镇寨坪村采用单株育种法选育而成,诏安与广东潮汕地区毗邻,由于‘八仙茶’形态特征和茶叶内质都与广东凤凰种群相似,推测可能是凤凰单枞的后代。而‘朝阳’是从四川的‘崇庆枇杷茶’有性后代中采用单株育种法育成。‘八仙茶’和‘朝阳’这两个品种与其他品种的地理距离都较远。‘大红袍’和‘福建水仙’分别发源于闽北茶区的武夷山市与建阳市,有独特的形态和种性特征,‘福建水仙’株型高大,分枝稀少、叶片宽大肥厚、结实率极低;‘大红袍’为武夷四大名枞之首,制茶品质独特。
乌龙茶品种比绿茶品种的遗传多样性丰富。16个绿茶品种有11个聚于j类群,主要为福云系列品种(如‘福云6号’、‘福云10号’、‘福云7号’、‘福云595’、‘福云20号’等)以及来源于福鼎市的品种(如‘福鼎大白茶’、‘歌乐茶’、‘福鼎大毫茶’、‘早逢春’等),遗传背景较为接近,相似系数较高。而27个乌龙茶品种的聚类结果则表现得更为多样,距离最远的4个品种全部为乌龙茶品种,而且乌龙茶品种的遗传距离整体比绿茶品种远。乌龙茶品种遗传多样性比绿茶品种高的原因主要有两个,首先乌龙茶品种中来自地方品种的比例高,这些地方品种发源地各不相同,遗传背景差异较大;第二,在人工选育品种中,绿茶品种的杂交亲本基本都是‘福鼎大白茶’和‘云南大叶种’,而乌龙茶品种的杂交亲本相对要更加丰富,有‘铁观音’、‘黄旦’、‘大红袍’、‘肉桂’、‘白芽奇兰’等。
相同亲本来源的品种表现出更高的相似系数, 从而聚于一簇。如绿茶品种中的福云系列,‘福云6号’、‘福云10号’、‘福云7号’、‘福云595’、‘福云20号’等,都聚于j类群,其亲本均为‘福鼎大白茶’和‘云南大叶种’。乌龙茶品种中亲本为‘黄旦’的品种有‘黄观音’、‘黄玫瑰’、‘瑞香’、‘黄奇’等,这5个品种不仅形态特征有相似之处,而且呈现出极高的遗传相似系数,从而聚在一起(e类群),聚类结果与亲缘关系和形态特征相互应证。
发源地相同的品种,也表现出较高的相似系数而聚于一簇。如来源于福鼎市的品种‘福鼎大白茶’、‘歌乐茶’、‘福鼎大毫茶’、‘早逢春’等聚为一类(j类群)。而同样来源于安溪的品种如‘铁观音’、‘杏仁茶’、‘本山’、‘凤圆春’等也聚为一类(d类群)。
3 讨论SSR分子标记技术在茶树种质资源遗传多样性和指纹图谱构建中已被广泛应用。如应用SSR标记对云南[12, 15]、广西[7]、浙江[10]和江北[11]茶区茶树资源的遗传多样性和遗传结构进行了研究。在指纹图谱构建方面,构建了云南[17]、黔南[20]、广西[21]等地的茶树分子指纹图谱。目前利用分子标记研究福建茶树种质资源的报道极少,陈常颂等用RAPD分子标记对福建省选育的茶树品种进行遗传多样性分析[23],由于RAPD技术存在重复性较差、条带多、无法构建指纹图谱等缺点,现已很少使用。但对比其实验结果发现,大部分结论仍与本文是一致的, 如来源地相同的品种亲缘关系较近,有共同亲本的品种遗传相似性较高等。这可能是实验中采用较多的RAPD引物,获得了数量较多的条带,弥补了RAPD技术不稳定的缺陷,从而降低了实验误差。王让剑等利用SSR标记对福建省的15个茶树品种进行遗传差异分析,并构建了15个品种的SSR指纹图谱[18]。由于文章中参试品种少,而且使用的引物数量也少(6对),获得的可用条带太少(26个位点), 所以在比较品种间的遗传多样性时误差较大。但是其中某些结论与本文是一致的,如黄观音、黄旦、黄玫瑰等3个品种遗传相似性高,离金观音最近的品种是金牡丹,以及地理距离远的品种遗传距离也远。但以上两篇文章没有对福建省选育的品种进行广泛的指纹图谱构建和充分的遗传多样性分析。
本研究共用7对SSR引物构建了43个福建省选育的茶树品种指纹图谱,能够清楚地区分不同茶树品种。但由于实验中采用PAGE胶电泳,其分辨能力有限,造成等位基因丰富的引物在统计条带时难以区分,加之参试品种较多更是增加了统计的难度,所以为了减少试验误差,试验选择等位基因少、条带清晰、间隔明显的引物,而把条带多、密集、多态性丰富的引物淘汰。最后用了相对多的引物(7对)构建指纹图谱,这还与参试品种中部分品种的遗传相似性太高有关,需要用更多的引物才能区分开。当然如果采用分辨率和灵敏度更高的毛细管电泳是解决以上问题的最好办法,由于毛细管电泳可以明确的知道每条带的碱基数,从而可以使用等位基因丰富的引物,然后用更少的SSR标记构建指纹图谱。所以后续开展指纹图谱构建的相关工作,将会使用毛细管电泳方法,建立更精准的指纹图谱。茶树SSR分子标记指纹图谱的构建对品种鉴别、真假品种鉴定、苗木纯度检测、品种权保护等都有重要的意义。
遗传相似系数和聚类分析表明,福建省选育的茶树品种中,乌龙茶品种比绿茶品种的遗传多样性丰富;共同亲本的茶树品种表现出较高的相似系数而聚为一簇;发源地相同的品种,也表现出较高的相似系数而聚为一簇,可见地理距离与遗传距离呈现出很高的正相关性。因此也对福建省选育的茶树品种的遗传多样性、亲缘关系、遗传背景有了较系统的认识。
当前福建省人工选育茶树品种遇到遗传背景狭窄的问题,如早期的‘福鼎大白茶’与‘云南大叶种’杂交选育的福云系列品种,还有‘铁观音’与‘黄旦’杂交选育的一系列茶树新品种,都由于亲本来源狭窄造成品种间的遗传距离太近。通过对福建省茶树品种的遗传多样性分析,为茶树品种的遗传改良提供一定的参考依据,特别是在育种过程中选择遗传距离更远的亲本,不仅能够提高品种的遗传多样性,也有利于杂种优势的利用,对改变当前遗传背景狭窄状况有重要的作用。同时对茶树生产过程中的选种和引种都有指导意义,通过选择亲缘关系远的品种,不仅有利于提高当地茶园的遗传多样性, 还对提高茶产品种类以及病虫害抗性都有重要的作用。
| [1] | Ma J Q, Yao M Z, Ma C L, et al. Construction of a SSR-based genetic map and identification of QTLs for catechins content in tea plant (Camellia sinensis)[J]. PLoS One, 2014, 9(3): e93131 DOI:10.1371/journal.pone.0093131 |
| [2] | TAN L Q, WANG L Y, WEI K, et al. Floral transcriptome sequencing for SSR marker development and linkage map construction in the tea plant (Camellia sinensis)[J]. PLoS One, 2013, 8(11): e81611 DOI:10.1371/journal.pone.0081611 |
| [3] |
ZHANG C C, LIU Y, JIANG Y H, et al. Application of SSR markers in cultivar identification of clonal tea plant in Zhejiang Province, China[J].
J Plant Genet Resour, 2014, 15(5): 926-931. 张成才, 刘园, 姜燕华, 等. SSR标记鉴定浙江省主要无性系茶树品种的研究[J]. 植物遗传资源学报, 2014, 15(5): 926-931. DOI:10.13430/j.cnki.jpgr.2014.05.002 |
| [4] |
ZHANG J R, WEI C L. Establishment a SSR-PCR system of tea plant[Camellia sinensis (L.) O. Kuntze] based on the 4300 DNA analysis system[J].
J Tea Sci, 2014, 34(5): 481-488. 张洁茹, 韦朝领. 基于4300 DNA分析系统的茶树SSR发掘方法优化与建立[J]. 茶叶科学, 2014, 34(5): 481-488. DOI:10.3969/j.issn.1000-369X.2014.05.009 |
| [5] |
CHEN Z H, SHAN R Y, LIN Z H, et al. Genetic diversity of Fuyun half-sib teas from Fujian determined by using SSR markers[J].
Acta Tea Sin, 2015, 56(4): 238-243. 陈志辉, 单睿阳, 林郑和, 等. 利用SSR标记分析福建省选育福云半同胞系茶树品种遗传多样性[J]. 茶叶学报, 2015, 56(4): 238-243. DOI:10.3969/j.issn.1007-4872.2015.04.007 |
| [6] |
JIN J Q, CUI H R, GONG X C, et al. Studies on tea plants (Camellia sinensis) germplasms using EST-SSR marker[J].
Hereditas, 2007, 29(1): 103-108. 金基强, 崔海瑞, 龚晓春, 等. 用EST-SSR标记对茶树种质资源的研究[J]. 遗传, 2007, 29(1): 103-108. DOI:10.3321/j.issn:0253-9772.2007.01.019 |
| [7] |
ZHOU Y H, QIAO X Y, MA C L, et al. Genetic diversity and structure of tea landraces from Guangxi based on EST-SSR analysis[J].
Sci Silv Sin, 2011, 47(3): 59-67. 周炎花, 乔小燕, 马春雷, 等. 广西茶树地方品种遗传多样性和遗传结构的EST-SSR分析[J]. 林业科学, 2011, 47(3): 59-67. DOI:10.11707/j.1001-7488.20110310 |
| [8] |
ZHOU M, LI Y Y, SUN X M, et al. Genetic diversity analysis of Xiangzhuqing tea king and other tea trees based on EST-SSR markers[J].
SW China J Agric Sci, 2016, 29(2): 231-239. 周萌, 李友勇, 孙雪梅, 等. 基于EST-SSR分子标记对香竹箐茶树王的遗传多样性分析[J]. 西南农业学报, 2016, 29(2): 231-239. DOI:10.16213/j.cnki.scjas.2016.02.005 |
| [9] |
WANG L Y, JIANG Y H, DUAN Y S, et al. Genetic diversity of tea landraces using SSR markers[J].
Acta Agron Sin, 2010, 36(12): 2191-2195. 王丽鸳, 姜燕华, 段云裳, 等. 利用SSR分子标记分析茶树地方品种的遗传多样性[J]. 作物学报, 2010, 36(12): 2191-2195. DOI:10.3724/SP.J.1006.2010.02191 |
| [10] |
QIAO T T, MA C L, ZHOU Y H, et al. EST-SSR genetic diversity and population structure of tea landraces and developed cultivars (lines) in Zhejiang Province, China[J].
Acta Agron Sin, 2010, 36(5): 744-753. 乔婷婷, 马春雷, 周炎花, 等. 浙江省茶树地方品种与选育品种遗传多样性和群体结构的EST-SSR分析[J]. 作物学报, 2010, 36(5): 744-753. DOI:10.3724/SP.J.1006.2010.00744 |
| [11] |
YAO M Z, LIU Z, CHEN L, et al. Genetic diversity and structure of tea germplasm originated from region of north Yangtze River based on EST-SSR markers[J].
J Tea Sci, 2009, 29(3): 243-250. 姚明哲, 刘振, 陈亮, 等. 利用EST-SSR分析江北茶区茶树资源的遗传多样性和遗传结构[J]. 茶叶科学, 2009, 29(3): 243-250. DOI:10.13305/j.cnki.jts.2009.03.008 |
| [12] |
JIANG H B, SONG W X, YI B, et al. Genetic diversity of tea germplasm resources in Yunnan province based on phenotypic charac-teristics[J].
Acta Agron Sin, 2013, 39(11): 2000-2008. 蒋会兵, 宋维希, 矣兵, 等. 云南茶树种质资源的表型遗传多样性[J]. 作物学报, 2013, 39(11): 2000-2008. DOI:10.3724/SP.J.1006.2013.02000 |
| [13] |
WU Q H, ZHUANG D H, ZHU H, et al. Genetic diversity of Fenghuang-Dancong tea plant germplasms by ISSR markers[J].
Chin J Trop Crops, 2015, 36(3): 499-503. 吴清韩, 庄东红, 朱慧, 等. 凤凰单丛茶树资源遗传多样性的ISSR分析[J]. 热带作物学报, 2015, 36(3): 499-503. DOI:10.3969/j.issn.1000-2561.2015.03.009 |
| [14] |
HUANG X X, TANG T, JIANG Y L, et al. Genetic diversity of wild tea plant in different altitude in Qianjiazhai[J].
J Tea Sci, 2015, 35(4): 347-353. 黄晓霞, 唐探, 姜永雷, 等. 千家寨不同海拔野生茶树的EST-SSR遗传多样性研究[J]. 茶叶科学, 2015, 35(4): 347-353. DOI:10.3969/j.issn.1000-369X.2015.04.009 |
| [15] |
ZHOU M, LI Y Y, SUN X M, et al. Genetic diversity assessment of ancient tea plants in Yunnan Province of China revealed by EST-SSR markers[J].
Acta Agric Boreali-Sin, 2013, 28(S1): 91-96. 周萌, 李友勇, 孙雪梅, 等. 基于EST-SSR标记的云南大茶树遗传多样性分析[J]. 华北农学报, 2013, 28(S1): 91-96. DOI:10.7668/hbnxb.2013.S1.018 |
| [16] |
ZHANG Z F, MA J Q. Analysis of genetic diversity and construction of molecular fingerprinting for new tea tree varieties based on SSR markers[J].
Hunan Agric Sci, 2012(19): 1-4. 章志芳, 马建强. 基于SSR标记的茶树新品种遗传多样性分析及指纹图谱构建[J]. 湖南农业科学, 2012(19): 1-4. DOI:10.3969/j.issn.1006-060X.2012.19.001 |
| [17] |
LIU B Y, SUN X M, LI Y Y, et al. Analysis of genetic diversity and construction of DNA fingerprinting with EST-SSR markers for improved clonal tea cultivars in Yunnan Province[J].
J Tea Sci, 2012, 32(3): 261-268. 刘本英, 孙雪梅, 李友勇, 等. 基于EST-SSR标记的云南无性系茶树良种遗传多样性分析及指纹图谱构建[J]. 茶叶科学, 2012, 32(3): 261-268. DOI:10.13305/j.cnki.jts.2012.03.011 |
| [18] |
WANG R J, YANG J, KONG X R, et al. Genetic variances and DNA fingerprints of 15 teas in Fujian[J].
Fujian J Agric Sci, 2014, 29(10): 970-975. 王让剑, 杨军, 孔祥瑞, 等. 福建15个茶树品种SSR遗传差异分析与指纹图谱建立[J]. 福建农业学报, 2014, 29(10): 970-975. DOI:10.3969/j.issn.1008-0384.2014.10.008 |
| [19] |
HUANG D J, MA J Q, CHEN L. Research progress on DNA molecular fingerprinting of tea plant (Camellia sinensis)[J].
J Tea Sci, 2015, 35(6): 513-519. 黄丹娟, 马建强, 陈亮. 茶树DNA分子指纹图谱研究进展[J]. 茶叶科学, 2015, 35(6): 513-519. DOI:10.3969/j.issn.1000-369X.2015.06.001 |
| [20] |
CHEN S J, ZHANG M Z, YAO Y X, et al. Establishment of DNA fingerprinting for tea germplasm from Qiannan prefecture by SSR markers[J].
J Plant Genet Resour, 2017, 18(1): 106-111. 陈世军, 张明泽, 姚玉仙, 等. 基于SSR标记的黔南茶树种质资源DNA指纹图谱构建[J]. 植物遗传资源学报, 2017, 18(1): 106-111. DOI:10.13430/j.cnki.jpgr.2017.01.013 |
| [21] |
CHEN Y Y, YUAN S S, WU C L, et al. Molecular fingerprint and diversity analysis of wild tea trees in Jinxiu County, Guangxi Province[J].
Guangdong Agric Sci, 2016, 43(3): 60-65. 陈莹玉, 袁思思, 吴春兰, 等. 广西金秀野生茶遗传多样性及分子指纹图谱研究[J]. 广东农业科学, 2016, 43(3): 60-65. DOI:10.3969/j.issn.1004-874X.2016.03.013 |
| [22] | Ma J Q, Huang L, Ma C L, et al. Large-scale SNP discovery and genotyping for constructing a high-density genetic map of tea plant using specific-locus amplified fragment sequencing (SLAF-seq)[J]. PLoS One, 2015, 10(6): e0128798 DOI:10.1371/journal.pone.0128798 |
| [23] |
CHEN C S, CHEN Z H, ZHONG Q S, et al. Comparison of growth period and biochemical composition on Fujian tea cultivars and genetic diversity analysis[J].
J Tea Sci, 2014, 34(2): 172-179. 陈常颂, 陈志辉, 钟秋生, 等. 闽育茶树品种生育期与生化成分比较及遗传多样性分析[J]. 茶叶科学, 2014, 34(2): 172-179. DOI:10.3969/j.issn.1000-369X.2014.02.012 |
 2017, Vol. 25
2017, Vol. 25


