2. 河北农业大学园艺学院, 河北 保定 071001
2. College of Horticulture, Agricultural University of Hebei, Baoding 071001, Hebei, China
MicroRNA (miRNA)是由茎环结构的前体RNA经过Dicer-like核酸内切酶加工后形成的非编码小分子RNA,长度一般为21~30 nt[1]。miRNA最早发现于线虫[2],随后在拟南芥(Arabidopsis thaliana)、水稻(Oryza sativa)、小麦(Triticum aestivum)和玉米(Zea mays)等植物中大量发现[3-6]。研究表明,植物miRNA通过切割靶基因的方式导致靶基因沉默来参与几乎所有的生物代谢途径,对植物的生长发育以及响应非生物胁迫过程具有重要的调控作用。水稻miR397和miR156参与愈伤组织从未分化到分化的转化过程,对分生组织的维持和胚胎的发生具有重要调节作用[7]。miR408是营养生长的强大调节剂,其组成型表达能够增强拟南芥幼苗和成年植株的生长[8]。拟南芥中miR397、miR398、miR408和miR857可以通过下调编码含铜蛋白(质体蓝素和铜/锌超氧化物歧化酶)的基因表达,来应对环境铜的变化,以维持植物体内铜的平衡[9]。拟南芥中miR398还能通过切割两个Cu/Zn超氧化物歧化酶基因(CSD1和CSD2)的mRNA来实现对其调控,而表达抗miR398切割的CSD2能够比过量表达CSD2的植株更具有抗强光、重金属以及氧化胁迫的能力[10]。过量表达miR408能够提高转基因植株的耐盐、抗寒和抗氧化胁迫能力,但其对干旱和渗透胁迫的敏感性也增加[11]。人类生存环境的变化, 迫使人们不断寻求抗逆、高产、优质的植物资源, 挖掘其中具有重要育种价值的基因资源用于现代植物分子聚合育种。
竹类植物资源被认为是一种可持续利用的再生资源,是热带亚热带地区森林资源的重要组成部分,具有重要的生态、经济价值。我国竹类植物资源丰富,种类繁多,但其特殊的生殖特性严重限制了其常规育种的发展。随着现代生物技术的发展, 分子育种将为竹子的育种带来新的契机,挖掘具有潜在育种价值的基因资源是开展分子育种的基础, 毛竹基因组草图[12]和竹子基因组数据库的建立为开展竹子基因资源的挖掘奠定了基础。除了参与竹子生长发育和逆境适应相关的结构基因、调节基因,关于竹子miRNA的研究也日趋受到重视。如通过小RNA测序在毛竹(Phyllostachys edulis)叶片中发现了92个保守miRNA和95个新的miRNA, 在麻竹(Dendrocalamus latiflorus)叶片中发现了已知的成熟miRNA 69个和新miRNA 62个[13]。通过对PheDof1、PheMADS14和6个miRNA (miR159a.1、miR160a、miR168-3p、miR390a、miR393和miR5139) 的表达分析,揭示出miRNA在毛竹花发育和成花转变中发挥着重要作用[14];对phe-miR397和phe-miR1432在光照、干旱、NaCl和温度胁迫以及ABA和GA3处理后的表达研究表明它们可能参与调控毛竹抵御非生物胁迫,且与内源激素的调节相关联[15]。麻竹miR172a通过调控其靶基因DlAP2来实现对麻竹花芽发育的调控[16]。然而,对竹类植物中miRNA的研究刚刚开始,尚有更多的miRNA需要深入研究。本文以毛竹为材料,从中分离了miR398和miR408的前体序列,并对其上游启动子序列进行了分析,对多种非生物胁迫下毛竹中miR398和miR408表达的变化进行了研究,以期为进一步揭示miR398和miR408在竹子抗逆中的作用提供参考,并为未来培育竹子抗逆新品种提供理论基础。
1 材料和方法 1.1 材料和处理毛竹(Phyllostachys edulis)种子播种萌发后,盆栽于营养钵内,栽培基质为腐殖质土。培养条件为: 25℃,光照强度150~200 μmol m-2s-1,光周期光/暗=16 h/8 h,至长出5个叶片(约0.5年)用于试验。分别取盆栽毛竹实生苗的根、茎、叶片和叶鞘液氮速冻后存-80℃备用。
选取长势一致的毛竹盆栽苗,一部分分别在黑暗、强光(1500 μmol m-2s-1)和低温(4℃)下处理3 h; 另一部分分别用1%蔗糖、CuSO4(50 µmol L-1)[17]、NaCl (300 mmol L-1)[18]、GA3 (100 µmol L-1)和ABA (100 µmol L-1)[19]处理3 h,处理后取叶片样品。
1.2 总RNA提取与cDNA合成采用Trizol法[20]分别提取毛竹根、茎、叶片、叶鞘以及胁迫处理后叶片的总RNA,于37℃用RNase-free DNase Ⅰ (Promega, USA)处理30 min,以去除基因组DNA污染。分别采用1%琼脂糖凝胶电泳和超微量分光光度计(Nanodrop 2000, Thermo美国)来检测RNA的质量和浓度。利用反转录试剂盒(Promega, USA)提供的通用引物,将毛竹叶片RNA合成cDNA。
根据BambooGDB数据库中毛竹miR398与miR408成熟序列及前体序列(PH01001517/PH01003-062), 按照茎环法[21]分别设计反转录引物、正向引物和反向通用引物(Universal reversal primer, URP), 正向引物3′端序列与成熟miRNA一致,反转录引物与成熟miRNA 3′端序列的碱基完全互补(表 1)。引物由上海生物工程技术服务有限公司合成。分别利用反转录引物将符合质量要求的上述RNA合成cDNA, 每个miRNA对应的样品进行一次反转录,重复3次。
| 表 1 PCR所用引物 Table 1 List of PCR primer sequences |
采用改良CTAB法[22]提取毛竹叶片基因组DNA, 并作为模板,分别采用引物对398-F/398-R和408-F/ 408-R进行PCR,扩增毛竹miR398与miR408的前体序列。PCR反应体系(20 μL):10×Pyrobest PCR Buffer 2 μL,dNTP mixed (2.5 mmol L-1) 1.6 μL,398-F或408-F (10 μmol L-1) 1 μL,10 μmol L-1的398-R或408-R 1 μL,DMSO 1.1 μL,cDNA 1.0 μL,超纯水12.2 μL,Pyrobest酶0.2 μL。反应程序:95℃预变性5 min;95℃变性15 s,60℃退火150 s,72℃延伸20 s,反应36个循环;72℃延伸5 min,10℃保温。电泳检测后进行回收、加A、连接到pGEMT-easy载体,并转化大肠杆菌涂板后挑选阳性克隆,送检测序。
1.4 生物信息学分析利用在线软件RNAfold WebSever (http://rna.tbi.univie.ac.at/cgi-bin/RNAfold.cgi)进行miRNA前体二级结构的预测,利用WebLOGO (http://weblogo. berkeley.edu/logo.cgi)对miRNA成熟序列的碱基保守性进行分析。利用公共数据库miRBase21 (http://www.mirbase.org/)和毛竹数据库(http://www.bamboogdb.org/)下载玉米、水稻、高粱(Sorghum bicolor)、二穗短柄草(Brachypodium distachyon)、大豆(Glycine max)、烟草(Nicotiana tabacum)、毛竹等植物的miR398和miR408成熟序列及前体序列, 用MEGA 6.0软件构建基于miRNA前体的系统进化树。下载miR398和miR408前体上游1500 bp的启动子序列,利用PlantCARE (http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)对其启动子中的作用元件进行分析。
1.5 表达模式分析以颈环反转录引物合成的miRNA特异性cDNA为模板,分别用398-F1和408-F1与通用引物(URP)配对使用,对毛竹根、茎、叶片、叶鞘以及不同胁迫处理后毛竹叶片样品中miR398和miR408的表达量进行实时定量PCR (qRT-PCR)分析,同时以U6为内参基因[23]。qRT-PCR选用Roche公司LightCycle 480 SYBR Green Ⅰ Master试剂盒, 在qTOWER2.2 PCR仪(Jena, Germany)上进行,反应体系(10 μL):Mix 5 μL,cDNA 0.4 μL,398-F1或408-F1 0.2 μL,URP 0.2 μL,超纯水4.2 μL。反应程序:95℃预变性5 min;95℃变性10 s,60℃退火10 s,45个循环。每个样品每次反应重复3次,以3次生物学重复的试验结果用2-ΔΔCT法进行分析[24]。
2 结果和分析 2.1 miRNA前体序列分析及结构预测以毛竹基因组DNA为模板,采用引物对398-F/ 398-R和408-F/408-R进行PCR扩增,产物经电泳检测,结果表明二者在450 bp左右均有一特异条带(图略),测序表明序列长度分别为438 bp和447 bp,与预测的包含毛竹前体ped-MIR398和ped-MIR408的序列大小一致,ped-MIR398与ped-MIR408分别为83 bp和92 bp。
以毛竹cDNA为模板,分别用特异引物398-F1/ UPR和408-F1/UPR对成熟序列进行扩增,扩增产物经电泳检测,在70 bp左右有一特异条带(图略),测序表明二者长度均为69 bp,去除通用引物序列后,分别包含各自成熟miRNA ped-miR398与ped-miR408的对应序列5′-GGGGCGAATTGAGAACA-CATGGTG-3′和5′-GGCACAGCACTGTAGACCC-GGCCC-3′,均为24 bp。
下载miRbase (http://mirbase.org/index.shtml)中现有的miRNA前体序列,用MEGA6.0构建基于miR398与miR408前体序列的进化树。结果表明, 来自双子叶植物和单子叶植物的miR408家族的成员分别聚成两大分支,其中ped-MIR408虽然与其他单子叶成员聚类到一起,但并没有与其它成员聚类到相同的分枝,这意味着ped-MIR408的进化可能更为原始(图 1: A)。miR398家族的成员虽然也聚类成两个大的分支,但并非是单子叶植物和双子叶植物分枝,其中来自双子叶植物烟草、大豆和可可树(Theobroma cacao)的miR398前体却与其它单子叶植物的成员聚类到一起(图 1: B),这表明依据植物miRNA前体序列构建的进化树并非与现有的形态学分类完全一致。

|
图 1 基于前体序列构建的系统进化树(A: MIR408; B: MIR398)。每个分子上的数字表示1000次重复搜索的靴带值。 Fig. 1 Phylogenetic tree based on sequences of precursors (A: MIR408; B: MIR398). Numbers on major branches indicate bootstrap estimates for 1000 replicate analysis. |
利用RNAfold WebSever预测前体序列的二级结构,结果表明ped-MIR398与ped-MIR408均能形成稳定的茎环结构,且在茎环结构5′端的臂上分别为其成熟序列ped-miR398 (5′-GGGGCGAAUUG-AGAACACAUGGUG-3′)(图 2: A)和ped-miR408 (5′-GGCACAGCACUGUAGACCCGGCCC-3′)(图 2: B), 它们与来自其它单子叶植物的miR398与miR408前体序列有着相似的结构,表明ped-MIR398与ped-MIR408在毛竹中是真实存在的。

|
图 2 ped-MIR398(A)和ped-MIR408(B)前体的二级结构 Fig. 2 Secondary structure of ped-MIR398 (A) and ped-MIR408 (B) |
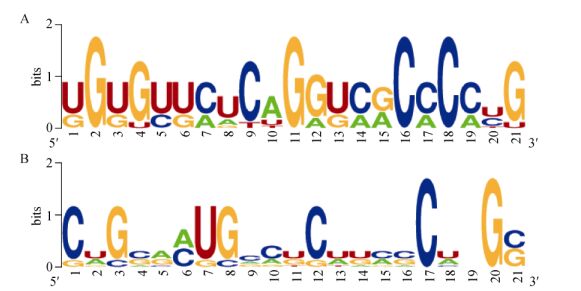
对已知miR398与miR408成熟序列的碱基保守性进行分析,结果表明,miR398家族成员的成熟序列的碱基保守性整体都较高,尤其是第2、4、9、11、16、18和21位的碱基保守性均较强(图 3: A),ped-miR398成熟序列的碱基保守性与其它成员一致。而miR408家族成熟序列的碱基保守性差异较大,仅在第1、3、7、8、12、17和20位上的碱基保守性较强(图 3: B),表明miR408在进化过程中比较活跃,在功能上ped-miR408可能与其它家族成员存在一定的差异,有待于进一步试验证实。
|
图 3 miR398(A)和miR408(B)成熟序列碱基保守性分析 Fig. 3 Conservative analysis of nucleotides in miR398 (A) and miR408 (B) |
虽然miRNA具有众多的调控功能,但其表达也受到启动子的调控。利用PlantCARE对毛竹ped-MIR398与ped-MIR408前体上游启动子序列进行预测,表明其中除了包含TATA-box和CAAT-box等启动子基本元件外,还含有多种顺式作用元件和应答元件(表 2),响应的环境因子涉及光、温度和激素等,由此推测ped-MIR398和ped-MIR408的表达可能会受到这些环境因子胁迫的诱导或抑制。
| 表 2 ped-MIR398和ped-MIR398上游启动子序列中顺势作用元件 Table 2 Cis-elements in the promoter sequences upstream ped-MIR398 and ped-MIR398 |
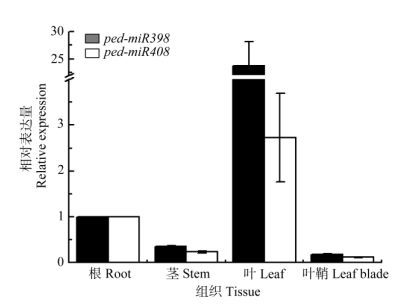
为揭示ped-miR398与ped-miR408的组织表达模式,采用实时定量PCR的方法对其在毛竹的根、茎、叶片和叶鞘中的表达进行了分析。结果表明, ped-miR398与ped-miR408在各组织中均有表达, 为组成型表达模式,且均在叶中表达丰度最高,根中次之,茎和叶鞘中表达量较少(图 4)。
|
图 4 ped-miR398与ped-miR408在不同组织中的表达分析 Fig. 4 Expression analysis of ped-miR398 and ped-miR408 in different tissues |
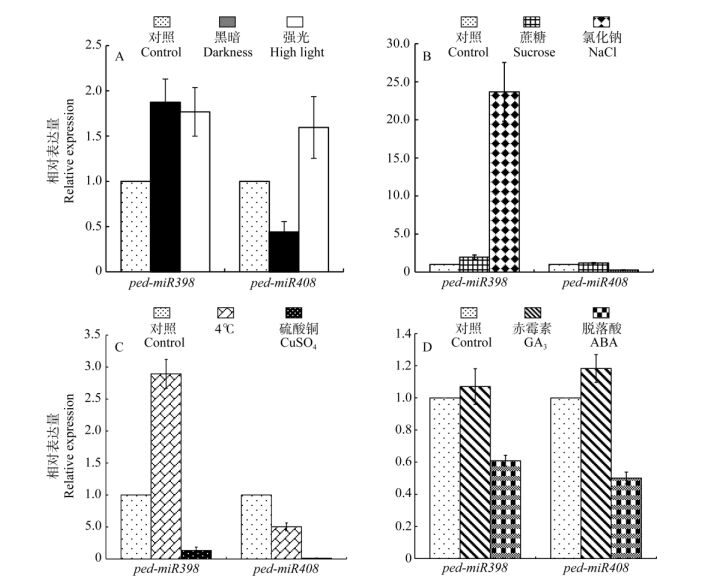
为探讨外界因子对ped-miR398与ped-miR408表达的影响,对不同处理的毛竹叶片中ped-miR398与ped-miR408的表达进行了定量分析。结果表明, ped-miR398与ped-miR408受强光和黑暗处理的反应并不一致,其中ped-miR398的相对表达量均上调,且黑暗处理对其影响更大,处理3 h后其表达量约为对照的1.9倍;而ped-miR408经黑暗处理后表达受到抑制,处理3 h后的表达量仅为对照的44%,强光诱导3 h后的表达量约为对照的1.6倍(图 5: A)。蔗糖处理后,ped-miR398与ped-miR408的表达均呈现不同程度的上调,分别约为对照的1.9倍和1.2倍; NaCl处理后,ped-miR398与ped-miR408的表达呈现相反的趋势,ped-miR398的表达显著上调,处理3 h后约为对照的23.7倍,ped-miR408的表达下调,处理3 h后仅为对照的30% (图 5: B)。低温(4℃)处理后,ped-miR398与ped-miR408的表达也呈现相反的趋势,处理3 h后ped-miR398的表达上调,为对照的2.9倍,而ped-miR408的表达则下调,为对照的一半(图 5: C)。CuSO4处理后,受高浓度的Cu2+胁迫ped-miR398与ped-miR408的表达均受到抑制,处理3 h后分别为对照的13%和1% (图 5: C)。GA3和ABA处理后,ped-miR398与ped-miR408的表达趋势相似,GA3处理3 h后ped-miR398与ped-miR408的表达上调,分别为对照的1.07倍和1.18倍,ABA处理3 h后分别下调为对照的60%和50% (图 5: D)。
|
图 5 不同处理条件下ped-miR398与ped-miR408的表达分析 Fig. 5 Expression of ped-miR398 and ped-miR408 under different treatments |
非编码RNA在基因表达调控中的作用越来越受到人们的关注[25-26],其中miRNA是近年来研究的热点之一。miRNAs作为一类新的具有调控基因表达能力的非编码RNA,在调控植物的生长发育、适应各种逆境胁迫过程中均发挥着重要作用。
miR398是首个被发现与受逆境胁迫调控相关的miRNA,在受到干旱、高盐和ABA等胁迫时能做出应答[27]。miR408首次发现于拟南芥中,虽对miR408的研究较少,但也有研究表明其在抗盐、抗冻等非生物胁迫方面也发挥着重要作用[28]。miRNA在不同植物物种间存在保守性[29],这也意味着不同物种间的miRNA可能发挥着相似的作用。对本研究克隆的毛竹miRNA分析表明,其前体序列ped-MIR408比较保守,而ped-MIR398则在进化上更具多样性,但其各自的成熟miRNA则都比较保守, 且miR398家族成员的碱基保守性整体高于miR408家族,表明同一家族miRNA功能的相似性,但具体ped-miR398和ped-miR408的调控功能需要进一步研究。
miRNAs虽然具有调控功能,但其自身的表达同样受到外界因子的影响。通过对毛竹ped-MIR398和ped-MIR408上游启动子区域的分析表明,有多种与逆境、激素相关的顺式作用元件,这意味着它们可能参与逆境胁迫以及激素的应答调控。ped-miR398和ped-miR408在强光、黑暗、蔗糖、NaCl、低温(4℃)、CuSO4、GA3和ABA等处理下的表达变化进一步证实了这一点。然而,不同胁迫、不同激素对miRNA表达的影响存在着一定的差异。如黑暗和强光均诱导ped-miR398的表达,而ped-miR408的表达则受到黑暗的抑制、强光的诱导; ped-miR398和ped-miR408在蔗糖胁迫下表达量均上调,在铜胁迫下则均下调;低温胁迫下ped-miR398表达量上调而ped-miR408表达量下调;GA3诱导ped-miR398和ped-miR408的表达,而ABA则抑制其表达。由此表明,miRNAs参与逆境胁迫和激素应答的机制可能十分复杂[18]。
miRNA的调控功能是通过调控基因的表达来实现的,包括转录调控、mRNA降解和翻译抑制等方式,其中miRNA与其靶位点mRNA互补结合, 引起靶mRNA的降解是植物中比较常见的方式,如毛竹ped-miR164b通过降解其靶基因PeSNAC1来参与器官边缘的发育和抗逆调控[30]。另外,miRNA的表达与功能受到转录因子、单核苷酸多态性、RNA编辑、甲基化以及生物钟等多种植物自身因素的调节[31]。因此,miRNAs的表达与调控是一个复杂的调控网络,1个miRNA既可以调控单个基因的表达,也可以调控多个基因的表达,某个基因的表达可以受到多个miRNA组合的精细调控。然而, 对竹子miRNA的研究刚刚起步,我们推测有多家族的miRNA参与了竹子生长发育调控[32],尚有众多miRNA的功能需要深入挖掘,且一些新的miRNA需要去发现。本研究对毛竹ped-miR398和ped-miR408的表达分析,为进一步开展二者及其对应靶基因之间的调控关系研究提供了初步参考,为揭示其在竹子生长发育过程中的调控机制奠定了基础。
| [1] | de Lima J C, Loss-Morais G, Margis R. MicroRNAs play critical roles during plant development and in response to abiotic stresses[J]. Genet Mol Biol, 2012, 35(4): 1069-1077. DOI:10.1590/S1415-47572012000600023 |
| [2] | Lee R C, Feinbaum R L, Ambros V. The C. elegans hetero-chronic gene lin-4 encodes small RNAs with antisense complemen-tarity to lin-14[J]. Cell, 1993, 75(5): 843-854. DOI:10.1016/0092-8674(93)90529-Y |
| [3] | Sunkar R, Girke T, Jain P K, et al. Cloning and characterization of microRNAs from rice[J]. Plant Cell, 2005, 17(5): 1397-1411. DOI:10.1105/tpc.105.031682 |
| [4] | Sunkar R, Zhu J K. Novel and stress-regulated microRNAs and other small RNAs from Arabidopsis[J]. Plant Cell, 2004, 16(8): 2001-2019. DOI:10.1105/tpc.104.022830 |
| [5] | Yao Y Y, Guo G G, Ni Z F, et al. Cloning and characterization of microRNAs from wheat (Triticum aestivum L.)[J]. Genome Biol, 2007, 8(6): R96 DOI:10.1186/gb-2007-8-6-r96 |
| [6] | Mica E, Gianfranceschi L, Pè M E. Characterization of five microRNA families in maize[J]. J Exp Bot, 2006, 57(11): 2601-2612. DOI:10.1093/jxb/erl013 |
| [7] | Luo Y C, Zhou H, Li Y, et al. Rice embryogenic calli express a unique set of microRNAs, suggesting regulatory roles of microRNAs in plant post-embryogenic development[J]. FEBS Lett, 2006, 580(21): 5111-5116. DOI:10.1016/j.febslet.2006.08.046 |
| [8] | Zhang H Y, Li L. SQUAMOSA promoter binding protein-like7 regulated microRNA408 is required for vegetative development in Arabidopsis[J]. Plant J, 2013, 74(1): 98-109. DOI:10.1111/tpj.12107 |
| [9] | ABDEL-GHANY S E, PILON M. MicroRNA-mediated systemic down-regulation of copper protein expression in response to low copper availability in Arabidopsis[J]. J Biol Chem, 2008, 283(23): 15932-15945. DOI:10.1074/jbc.M801406200 |
| [10] | Sunkar R, Kapoor A, Zhu J K. Posttranscriptional induction of two Cu/Zn superoxide dismutase genes in Arabidopsis is mediated by downregulation of miR398 and important for oxidative stress tolerance[J]. Plant Cell, 2006, 18(8): 2051-2065. DOI:10.1105/tpc.106.041673 |
| [11] | Ma C, Burd S, Lers A. miR408 is involved in abiotic stress responses in Arabidopsis[J]. Plant J, 2015, 84(1): 169-187. DOI:10.1111/tpj.12999 |
| [12] | Peng Z H, Lu Y, Li L B, et al. The draft genome of the fast-growing non-timber forest species moso bamboo (Phyllostachys heterocycla)[J]. Nat Genet, 2013, 45(4): 456-461. DOI:10.1038/ng.2569 |
| [13] | Zhao H S, Wang L L, Dong L L, et al. Discovery and comparative profiling of microRNAs in representative monopodial bamboo (Phyllostachys edulis) and sympodial bamboo (Dendrocalamus latiflorus)[J]. PLoS One, 2014, 9(7): e102375 DOI:10.1371/journal.pone.0102375 |
| [14] | Ge W, Zhang Y, Cheng Z C, et al. Main regulatory pathways, key genes and microRNAs involved in flower formation and development of moso bamboo (Phyllostachys edulis) [J/OL]. Plant Biotechn J, [2016-7-16]. doi: 10.1111/pbi.12593. |
| [15] |
Wang L L, Zhao H S, Sun H Y, et al. Cloning and expression analysis of miR397 and miR1432 in Phyllostachys edulis under stresses[J].
Sci Silv Sin, 2015, 51(6): 63-70. 王丽丽, 赵韩生, 孙化雨, 等. 毛竹miR397和miR1432的克隆及其逆境胁迫响应表达分析[J]. 林业科学, 2015, 51(6): 63-70. DOI:10.11707/j.1001-7488.20150608 |
| [16] |
Gao Z M, Lou Y F, Wang L L, et al. Cloning and expression analysis of miR172a targeted gene DlAP2 in Dendrocalamus latiflorus[J].
J Trop Subtrop Bot, 2015, 23(3): 245-251. 高志民, 娄永峰, 王丽丽, 等. 麻竹miR172a靶基因DlAP2的克隆及其表达[J]. 热带亚热带植物学报, 2015, 23(3): 245-251. DOI:10.11926/j.issn.1005-3395.2015.03.003 |
| [17] | Dugas D V, Bartel B. Sucrose induction of Arabidopsis miR398 represses two Cu/Zn superoxide dismutases[J]. Plant Mol Biol, 2008, 67(4): 403-417. DOI:10.1007/s11103-008-9329-1 |
| [18] | Jia X Y, Wang W X, Ren L G, et al. Differential and dynamic regulation of miR398 in response to ABA and salt stress in Populus tremula and Arabidopsis thaliana[J]. Plant Mol Biol, 2009, 71(1/2): 51-59. DOI:10.1007/s11103-009-9508-8 |
| [19] | Kayal E W, Navarro M, Marque G, et al. Expression profile of CBF-like transcriptional factor genes from Eucalyptus in response to cold[J]. J Exp Bot, 2006, 57(10): 2455-2469. DOI:10.1093/jxb/erl019 |
| [20] | Gao Z M, Li X P, Li L B, et al. An effective method for total RNA isolation from bamboo[J]. Chin For Sci Technol, 2006, 5(3): 52-54. |
| [21] | Chen C F, Ridzon D A, Broomer A J, et al. Real-time quantification of microRNAs by stem-loop RT-PCR[J]. Nucl Acids Res, 2005, 33(20): e179 DOI:10.1093/nar/gni178 |
| [22] |
Gao Z M, Fan S H, Gao J, et al. Extract genomic DNA from Phyllostachys edulis by CTAB-based method[J].
For Res, 2006, 19(6): 725-728. 高志民, 范少辉, 高健, 等. 基于CTAB法提取毛竹基因组DNA的探讨[J]. 林业科学研究, 2006, 19(6): 725-728. DOI:10.3321/j.issn:1001-1498.2006.06.009 |
| [23] | Ding Y F, Chen Z, Zhu C. Microarray-based analysis of cadmium-responsive microRNAs in rice (Oryza sativa)[J]. J Exp Bot, 2011, 62(10): 3563-3573. DOI:10.1093/jxb/err046 |
| [24] | Livak K J, Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT method[J]. Methods, 2001, 25(4): 402-408. DOI:10.1006/meth.2001.1262 |
| [25] | Carrington J C, Ambros V. Role of microRNAs in plant and animal development[J]. Science, 2003, 301(5631): 336-338. DOI:10.1126/science.1085242 |
| [26] | Hake S. MicroRNAs: A role in plant development[J]. Curr Biol, 2003, 13(21): R851-R852. DOI:10.1016/j.cub.2003.10.021 |
| [27] | Zhu C, Ding Y F, Liu H L. MiR398 and plant stress responses[J]. Physiol Plant, 2011, 143(1): 1-9. DOI:10.1111/j.1399-3054.2011.01477.x |
| [28] | Zhang H Y, He H, Wang X C, et al. Genome-wide mapping of the HY5-mediated genenetworks in Arabidopsis that involve both trans-criptional and post-transcriptional regulation[J]. Plant J, 2011, 65(3): 346-358. DOI:10.1111/j.1365-313X.2010.04426.x |
| [29] | Floyd S K, Bowman J L. Gene regulation: Ancient microRNA target sequences in plants[J]. Nature, 2004, 428(6982): 485-486. DOI:10.1038/428485a |
| [30] |
Wang L L, Zhao H S, Sun H Y, et al. Expression analysis of miR164b and its target gene PeNAC1 in Phyllostachys edulis under stress[J].
For Res, 2015, 28(5): 605-611. 王丽丽, 赵韩生, 孙化雨, 等. 胁迫条件下毛竹miR164b及其靶基因PeNAC1表达研究[J]. 林业科学研究, 2015, 28(5): 605-611. DOI:10.3969/j.issn.1001-1498.2015.05.001 |
| [31] | Cai Y M, Yu X M, Hu S N, et al. A brief review on the mechanisms of miRNA regulation[J]. Genom Proteom Bioinform, 2009, 7(4): 147-154. DOI:10.1016/S1672-0229(08)60044-3 |
| [32] | Zhao H S, Chen D L, Peng Z H, et al. Identification and charac-terization of microRNAs in the leaf of ma bamboo (Dendrocalamus latiflorus) by deep sequencing[J]. PLoS One, 2013, 8(10): 0078755 DOI:10.1371/journal.pone.0078755 |
 2017, Vol. 25
2017, Vol. 25


