在PCR反应体系中,加入荧光染料或者荧光探针,利用荧光信号的积累来检测整个反应的进程, 这种技术被称为实时荧光定量PCR[1],通过标准曲线对未知模板进行定量分析,其操作简易、快速、高效率,且具有高通量,高灵敏度的特性,在分子诊断、分子生物学研究、植物检疫和食品安全检测中有着广泛的应用[2]。然而,RNA的浓度和质量, 反转录的扩增效率,常使得在Real-time PCR中基因表达定量分析的精确度受到影响。为了减少由核糖核酸(RNA)质量和反转录效率引起的误差,以及消除RNA在不同样本之间的差异,通常通过内参基因对数据进行校正和标准化[3]。可靠的内参基因是在不同细胞类型、不同测试条件下都能稳定表达的管家基因,常用的内参基因有GAPDH (3-磷酸甘油醛脱氢酶基因)、TUB (β微管蛋白基因)、efl-5A (转录延伸因子基因)、TUA (α微管蛋白基因)、18S RNA (18S核糖体RNA)和actin (肌动蛋白基因)等[4]。虽然在大部分情况下内参基因的表达是稳定的,但最近的研究表明,这些稳定的内参基因在某些情况下表达会发生变化,所以,在选择内参基因的时候需要分析各种因素,以选择最适宜的内参基因或者内参基因的组合,来提高Real-time qPCR的准确性[1]。
红苞凤梨(Ananas comosus var. bracteatus)又名艳凤梨、斑叶凤梨,为凤梨科(Bromeliaceae)凤梨属多年生常绿草本。株形美丽独特,冠芽和叶片均红、白、绿相间,作插花时可保存嵌合性状1个月以上,成为国际上一种重要独特的鲜切花。但叶片嵌合性状在组织培养快繁过程中很不稳定,使得大量再生植株失去了彩叶特性而被淘汰,加大了彩叶植物培育的生产成本。为了提高红苞凤梨嵌合性状的稳定性,我们需要阐明红苞凤梨叶嵌合性状形成的分子机理[5],因此筛选稳定的内参基因在基因表达分析中起着关键的作用。本研究利用Real-time qPCR方法评价EF1、UBQ、ACT、GADPH、Histone、TUA、TUB、18S、elf-5A和α-tubulin等10个候选内参基因在红苞凤梨全绿苗、全白苗和不同发育过程中的表达水平,用geNorm、NormFinder分析其表达稳定性,以筛选出最适内参基因。为了进一步验证所筛选内参基因的可靠性,利用筛选到的内参基因分析与光合作用相关基因PetF在红苞凤梨不同叶色和不同发育阶段的表达水平,为红苞凤梨叶嵌合性状的研究奠定基础。
1 材料和方法 1.1 材料供试材料为生长良好的红苞凤梨(A. comosus var. bracteatus)组织培养全白苗和全绿苗,分别选用未展叶芽、6~8叶芽和10~12叶芽3个发育阶段(图 1)。每样本设3个生物学重复,每个生物学重复来自不同植株生长状态一致的3个叶片。

|
图 1 不同发育阶段的红苞凤梨全绿苗和全白苗。A; 愈伤组织, 示未展叶的全白芽;B:愈伤组织,示未展叶的全绿芽;C:具6~8片叶的全白苗;D:具6~8片叶的全绿苗;E:具10~12片叶的全白苗;F:具10~12片叶的全绿苗。标尺=1 cm Fig. 1 Green and albino shoots of Ananas comosus var. bracteatus at different development stages. A: Callus, showing complete white bud with rolled leaves; B: Callus, showing green bud with rolled leaves; C: Albino shoot with six to eight leaves; D: Green shoot with six to eight leaves; E: Albino shoot with ten to twelve leaves; F: Green shoot with ten to twelve leaves. Bar=1 cm |
选择EF1、UBQ、ACT、GAPDH、Histone、elF-5A、α-tubulin、TUA、TUB、18S等10个看家基因作为候选内参基因,以本课题组红苞凤梨转录组测序[7]得到的基因序列为模板,利用Primer Premier 5.0软件[6]设计引物(表 1),由上海生工公司合成。引物进行PCR扩增后,对扩增产物进行T-克隆和测序验证(上海生工)。将cDNA模板进行5个10×梯度稀释,对每个引物进行扩增效率检测,合格的引物用于荧光定量分析。
| 表 1 实时荧光定量PCR的引物序列 Table 1 Primer sequences for real-time qPCR |
以3个发育阶段的全白苗和全绿苗叶片为材料, 采用Trizol法[7]提取总RNA。利用nanodrop2000 (Thermo)超微量分光光度计进行RNA的浓度与纯度检测。质量合格的RNA按照RevertAid First Strand cDNA Synthesis Kit试剂盒(TaKaRa)说明书操作合成cDNA第一条链。
1.4 实时荧光定量PCR (Real-time qPCR)在analytikjena-qTOWER2.2荧光定量PCR仪上对10个候选内参基因按照SYBR Premix Ex TaqTM (TaKaRa)试剂盒说明书进行Real-time qPCR。反应体系:5 μL SYBR mix,上下游引物各0.5 μL,1μL cDNA模板,并加3 μL ddH2O,每个模板重复3次。扩增反应程序为95℃预变性30 s,然后95℃变性10 s,58℃退火30 s,共40个循环。扩增完成后从65℃到95℃进行熔解曲线分析,验证扩增的特异性。
1.5 数据分析geNorm软件分析 采用2-ΔΔCT计算内参基因的相对表达量,采用geNorm软件计算内参基因的平均表达稳定值(M)并进行排序(M值越小,表达就越稳定),同时计算内参基因的标准化因子配对差异度(Vn/n+1)以判定内参基因最适数目[8]。
NormFinder软件分析 NormFinder程序运行原理与geNorm程序类似,通过计算基因表达稳定值(S),然后根据稳定值排序,以表达稳定值最小的基因作为最稳的基因[9]。
2 结果和分析 2.1 荧光定量PCR引物的特异性及扩增效率试验所用引物PCR扩增产物经电泳检测均只有单一条带,且测序验证为目标基因片段,说明所用引物都能特异性地扩增各内参基因,也不存在引物二聚体,荧光定量PCR熔解曲线都只有明显的单一峰,进一步验证了扩增产物的特异性。经标准曲线分析,所有引物扩增效率均在99%~110%,回归系数为0.992~0.999 (表 2),均达到荧光定量PCR的模板和引物要求,保证了分析结果的正确性[4]。
| 表 2 Real-time RT-PCR检测定量引物的扩增效率 Table 2 Amplification efficiency of qRT-PCR primers used in real-time RT-PCR analysis |
采用geNorm软件计算内参基因的标准化因子配对差异度(Vn/n+1)以判定内参基因最适数目。由表 3、图 2可见,在红苞凤梨生长的第二、三阶段和绿、白苗中,有部分样品的Vn/n+1大于0.15, 其原因可能是样本本身跨度大,全白苗与全绿苗长势差异大所致,因此本试验取Vn/n+1最小值来确定最佳内参数[10]。
| 表 3 红苞凤梨3个发育阶段稳定性排名前五的基因 Table 3 Top five reference genes with stable expression in Ananas comosus var. bracteatus at three development stages |
在红苞凤梨的3个发育阶段中,10个候选内参基因的相对表达量差异极显著(P < 0.01)。geNorm软件计算不同内参基因的表达稳定值M均小于0.5, 第一发育阶段各内参基因的表达稳定值M的排序为TUB=α-tubulin< elF-5A < ACT < EF1 < Histone < TUA< GADPH < 18S < UBQ,标准因子配对变异度为V3/4=0.031;在第二发育阶段各内参基因的表达稳定值M的排序为Histone=α-tubulin < elF-5A < TUB < GADPH < TUA < EF1 < UBQ < 18S < ACT, 标准因子配对变异度为V2/3=0.04;在发育第三阶段各内参基因的表达稳定值M的排序为Histone=GADPH < α-tubulin < TUA < UBQ < elF-5A < ACT < EF1 < 18S < TUB, 标准因子配对变异度为V2/3=0.039。
NormFinder软件计算红苞凤梨第一发育阶段各内参基因的表达稳定值S排序为UBQ < 18S < α-tubulin < TUA=GADPH < TUB < Histone < elF-5A < ACT < EF1;在第二发育阶段中各内参基因的表达稳定值S排序为TUA < Histone < α-tubulin < EF1 < elF-5A < UBQ < 18S < TUB < GADPH < ACT;在第三发育阶段中各内参基因的表达稳定值S排序为TUA= UBQ < elF-5A < Histone < GADPH < α-tubulin < ACT < EF1 < 18S < TUB。
由图 2可知红苞凤梨在3个不同发育阶段中, V2/3处的值最小,均小于0.15,即表达最稳定。所以在筛选红苞凤梨不同发育阶段的内参基因时,选择最稳定的两个内参组合最为合适。再根据表 2, α-tubulin和Histone在3个发育阶段中的排名都较靠前,所以在红苞凤梨不同发育阶段中,以α-tubulin和Histone为最佳内参基因组合,在进行Real-time qPCR时的准确性最高。
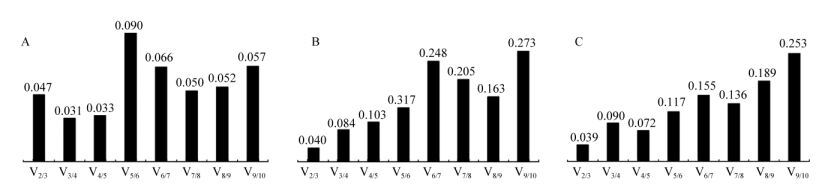
|
图 2 genorm软件分析红苞凤梨3个发育阶段的Vn/n+1。A:第一阶段; B:第二阶段; C:第三阶段。 Fig. 2 Vn/n+1 value of three development stages of Ananas comosus var. bracteatus by geNorm. A: Development stageⅠ; B: Development stage Ⅱ; C: Development stage Ⅲ. |
10个内参基因在红苞凤梨全白苗与全绿苗中的表达存在差异,从差异绿苗、白苗之间分析内参基因的稳定性。根据geNorm软件分析,在红苞凤梨全白苗与全绿苗中10个内参基因的平均表达稳定值M排序为EF1 < 18S=elF-5A < α-tubulin < TUA < TUB < ACT < UBQ < Histone < GAPDH,而根据NormFinder软件分析的表达稳定值S排序为18S < EF1=α-tubulin < TUA < elF-5A < TUB < ACT < UBQ < Histone < GAPDH,V3/4=0.07,因此选择3个内参基因组合最适宜(图 3)。根据表 4红苞凤梨全绿、全白苗对比10个内参基因的排名,选择排名前三的3个基因,分别是18S、EF1和α-tubulin (表 4)。
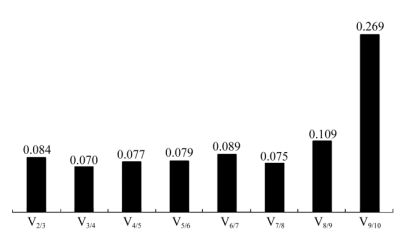
|
图 3 红苞凤梨全绿、全白苗的Vn/n+1值 Fig. 3 Vn/n+1 value of albino and green shoots of Ananas comosus var. bracteatus |
| 表 4 红苞凤梨全绿、全白苗10个内参基因稳定性排序 Table 4 Stability rank of 10 reference genes in albino and green shoots of Ananas comosus var. bracteatus |
以筛选出的Histone和α-tubulin为内参基因, 采用相同的定量PCR反应体系,分析PetF基因在红苞凤梨3个发育阶段中的表达模式。从图 4可见, 以2种内参基因分别进行PetF基因定量分析,结果表明PetF基因在3个发育阶段的变化趋势一致。因此,在红苞凤梨发育过程的PetF基因定量分析中可采用Histone和α-tubulin校正基因表达量。
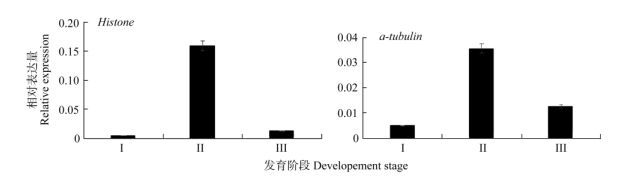
|
图 4 PetF基因在红苞凤梨叶片发育过程中的表达模式 Fig. 4 Expression of PetF in leaves of Ananas comosus var. bracteatus at different developmental stages |
取全绿和全白的红苞凤梨叶片,以筛选出的18S、EF1和α-tubulin为内参基因,验证PetF在不同叶色红苞凤梨中的表达水平。从图 5可见,采用不同的内参基因进行校正,PetF在红苞凤梨不同颜色叶片中的表达水平变化趋势一致,说明所选内参基因合适。
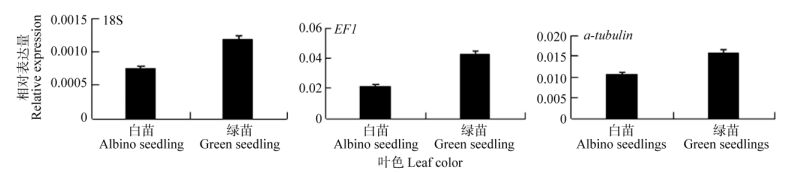
|
图 5 PetF基因在红苞凤梨不同颜色叶片中的表达 Fig. 5 Expression of PetF in albino and green leaves of Ananas comosus var. bracteatus |
Real-time qPCR技术现已成为分子生物学研究的重要工具,要想获取确实可信的基因表达结果, 除了需要有合理可行的实验设计、严格精准的实验操作、较高的RNA质量和逆转录效率、引物设计及正确的数据分析之外,稳定的内参基因也是必不可少的[2]。因此,选择合适的稳定表达的内参基因是准确进行基因定量表达的先决条件。
对于不同物种、不同发育时期、不同胁迫条件, 以及同一物种不同品种或不同组织,所选用的相对稳定表达的内参基因均有不同。汪永保等[11]对菠萝蜜(Artocarpus heterophyllus)内参基因稳定性的评价结果表明,对果实不同发育阶段宜用UBQ、GADPH和18S;对叶片不同发育阶段宜用UBQ、GADPH和β-tubulin;对花序不同发育阶段宜用UBQ、GADPH和α-tubulin。蒋婷婷等[12]认为在研究石蒜属换锦花(Lycoris sprengeri)不同组织基因表达时可选用β-tubulin、Actin和GADPH作为内参基因,不同花期的基因表达时宜用5S、EF-1α、Actin和β-tubulin作为内参基因;对中国石蒜(L. chinensis)不同组织基因表达研究最合适的内参基因是5S、UBQ、EF-1α和β-tubulin,不同花期最合适的内参基因是UBQ和β-tubulin。刘莉铭等[13]认为α-tubulin是在茎点枯病菌诱导下芝麻(Sesamum indicum)中最稳定的内参基因。樊连梅等[14]报道18S是苹果(Malus pumila)着色期基因表达的理想内参基因。张岗等[15]的研究表明18S是铁皮石斛(Dendrobium officinale)实时荧光定量PCR的最佳内参基因。苏晓娟等[16]报道18S可作为基因表达研究的内参基因。本文通过geNorm和NormFinder软件分析10个候选内参基因EF1、UBQ、ACT、GADPH、Histone、TUA、TUB、18S、elf-5A、α-tubulin在红苞凤梨全绿、全白苗和3个发育时期的表达稳定性,结果表明Histone和α-tubulin适合作为不同发育阶段基因表达研究的内参基因; 而18S、EF1和α-tubulin适合作为不同叶色基因表达研究的内参基因,这与以上研究结果较为一致。
PetF是与光合电子传递链有关的基因。本试验利用已筛选出的Histone和α-tubulin作为红苞凤梨不同发育阶段基因表达的内参基因,18S、EF1和α-tubulin作为不同叶色基因表达的内参基因,分别分析PetF在不同发育阶段和不同叶色中的表达水平。结果表明,采用这些内参基因,PetF基因的表达水平变化趋势一致,说明所选内参基因合适。
| [1] |
CHEN X, QI F K, KANG L G, et al. Advance and application of real-time fluorescent quantitative PCR[J].
J NE Agri Univ, 2010, 41(8): 148-155. 陈旭, 齐凤坤, 康立功, 等. 实时荧光定量PCR技术研究进展及其应用[J]. 东北农业大学学报, 2010, 41(8): 148-155. DOI:10.3969/j.issn.1005-9369.2010.08.029 |
| [2] | PFAFFL M W. A new mathematical model for relative quantification in real-time RT-PCR[J]. Nucl Acids Res, 2001, 29(9): e45 DOI:10.1093/nar/29.9.e45 |
| [3] |
ZHANG Y J, ZHU Z F, LU R, et al. Selection of control genes in transcription analysis of gene expression[J].
Prog Biochem Biophys, 2007, 34(5): 546-550. 张艳君, 朱志峰, 陆融, 等. 基因表达转录分析中内参基因的选择[J]. 生物化学与生物物理进展, 2007, 34(5): 546-550. DOI:10.3321/j.issn:1000-3282.2007.05.015 |
| [4] | RADONIĆ A, THULKE S, MACKAY I M, et al. Guideline to reference gene selection for quantitative real-time PCR[J]. Biochem Biophys Res Commun, 2004, 313(4): 856-862. DOI:10.1016/j.bbrc.2003.11.177 |
| [5] |
ZHONG X L. Chlorophyll synthesis rate-limiting gene mining of the albino cells of Ananas bracteatus [D]. Chengdu: Sichuan Agricultural University, 2015: 1-3Z.
钟小兰. 红苞凤梨金边嵌合体白化细胞叶绿素合成限速基因挖掘[D]. 成都: 四川农业大学, 2015: 1-3. |
| [6] |
YOU C, ZHAO D Q, LIANG C B, et al. Review of the PCR primer design method[J].
Mod Agri Sci Technol, 2011(17): 48-51. 尤超, 赵大球, 梁乘榜, 等. PCR引物设计方法综述[J]. 现代农业科技, 2011(17): 48-51. DOI:10.3969/j.issn.1007-5739.2011.17.022 |
| [7] | MA J, KANAKAL S, HE Y H, et al. Transcriptome sequence analysis of an ornamental plant, Ananas comosus var. bracteatus, revealed the potential unigenes involved in terpenoid and phenylpropanoid biosyn-thesis[J]. PLoS One, 2015, 10(3): e011915 DOI:10.1371/journal.pone.0119153 |
| [8] |
HU J C. How to screen reference genes for detecting gene expression by geNorm Software[J].
Chin J Lab Med, 2008, 31(8): 918 胡金川. 如何利用geNorm软件筛选基因表达测定的内参基因[J]. 中华检验医学杂志, 2008, 31(8): 918 DOI:10.3321/j.issn:1009-9158.2008.08.033 |
| [9] |
YANG L, YANG G H, LI C H, et al. Screening of reference genes in Anthurium andraeanum spathes for qRT-PCR analysis[J].
J Trop Subtrop Bot, 2015, 23(1): 51-58. 杨澜, 杨光穗, 李崇晖, 等. 红掌佛焰苞基因qRT-PCR分析中内参基因的筛选[J]. 热带亚热带植物学报, 2015, 23(1): 51-58. DOI:10.11926/j.issn.1005-3395.2015.01.008 |
| [10] |
TANG Y K, JIA Y Y. Method of processing real time PCR data[J].
Biotechnology, 2008, 18(3): 89-91. 唐永凯, 贾永义. 荧光定量PCR数据处理方法的探讨[J]. 生物技术, 2008, 18(3): 89-91. |
| [11] |
WANG Y B, YU Q, LI Y Z, et al. Screening of reference genes for real-time fluorescence quantitative PCR in jackfruit[J].
Chin J Trop Crops, 2014, 35(7): 1374-1381. 汪永保, 余庆, 李映志, 等. 菠萝蜜实时荧光定量PCR分析中内参基因的筛选[J]. 热带作物学报, 2014, 35(7): 1374-1381. DOI:10.3969/j.issn.1000-2561.2014.07.021 |
| [12] |
JIANG T T, GAO Y H, TONG Z K. Selection of reference genes for quantitative real-time PCR in Lycoris[J].
Acta Hort Sin, 2015, 42(6): 1129-1138. 蒋婷婷, 高燕会, 童再康. 石蒜属植物实时荧光定量PCR内参基因的选择[J]. 园艺学报, 2015, 42(6): 1129-1138. DOI:10.16420/j.issn.0513-353x.2014-0999 |
| [13] |
LIU L M, LIU H Y, TIAN B M. Selection of reference genes from sesame infected by Macrophomina phaseo-lina[J].
Acta Agron Sin, 2012, 38(3): 471-478. 刘莉铭, 刘红彦, 田保明. 茎点枯病菌诱导下芝麻内参基因的筛选[J]. 作物学报, 2012, 38(3): 471-478. DOI:10.3724/SP.J.1006.2012.00471 |
| [14] |
FAN L M, WANG C, LIU G S, et al. Screening and validation of reference genes for real-time fluorescence quantitative PCR during coloring period in apple (Malus domestica)[J].
Plant Physiol J, 2014, 50(12): 1903-1911. 樊连梅, 王超, 刘更森, 等. 苹果着色期实时定量PCR内参基因的筛选和验证[J]. 植物生理学报, 2014, 50(12): 1903-1911. DOI:10.13592/j.cnki.ppj.2014.0375 |
| [15] |
ZHANG G, ZHAO M M, ZHANG D W, et al. Reference gene selection for real-time quantitative PCR analysis of Dendrobium officinale[J].
Chin Pharm J, 2013, 48(19): 1664-1668. 张岗, 赵明明, 张大为, 等. 铁皮石斛实时定量PCR内参基因的筛选[J]. 中国药学杂志, 2013, 48(19): 1664-1668. DOI:10.11669/cpj.2013.19.015 |
| [16] |
SU X J, FAN B G, YUAN L C, et al. Selection and validation of reference genes for quantitative RT-PCR analysis of gene expression in Populus trichocarpa[J].
Chin Bull Bot, 2013, 48(5): 507-518. 苏晓娟, 樊保国, 袁丽钗, 等. 实时荧光定量PCR分析中毛果杨内参基因的筛选和验证[J]. 植物学报, 2013, 48(5): 507-518. DOI:10.3724/SP.J.1259.2013.00507 |
 2017, Vol. 25
2017, Vol. 25


